+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5pa4 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | humanized rat COMT in complex with 6-(4-fluorophenyl)-8-hydroxy-3H-quinazolin-4-one | ||||||
 要素 要素 | Catechol O-methyltransferase カテコール-O-メチルトランスフェラーゼ カテコール-O-メチルトランスフェラーゼ | ||||||
 キーワード キーワード |  TRANSFERASE (転移酵素) / TRANSFERASE (転移酵素) /  METHYLTRANSFERASE (メチルトランスフェラーゼ) / NEUROTRANSMITTER DEGRADATION METHYLTRANSFERASE (メチルトランスフェラーゼ) / NEUROTRANSMITTER DEGRADATION | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Enzymatic degradation of dopamine by COMT / Enzymatic degradation of Dopamine by monoamine oxidase / positive regulation of homocysteine metabolic process /  メチル化 / norepinephrine secretion / response to dopamine / メチル化 / norepinephrine secretion / response to dopamine /  咀嚼 / catecholamine catabolic process / catechol-containing compound metabolic process / S-adenosylhomocysteine metabolic process ...Enzymatic degradation of dopamine by COMT / Enzymatic degradation of Dopamine by monoamine oxidase / positive regulation of homocysteine metabolic process / 咀嚼 / catecholamine catabolic process / catechol-containing compound metabolic process / S-adenosylhomocysteine metabolic process ...Enzymatic degradation of dopamine by COMT / Enzymatic degradation of Dopamine by monoamine oxidase / positive regulation of homocysteine metabolic process /  メチル化 / norepinephrine secretion / response to dopamine / メチル化 / norepinephrine secretion / response to dopamine /  咀嚼 / catecholamine catabolic process / catechol-containing compound metabolic process / S-adenosylhomocysteine metabolic process / response to salt / 咀嚼 / catecholamine catabolic process / catechol-containing compound metabolic process / S-adenosylhomocysteine metabolic process / response to salt /  catechol O-methyltransferase activity / renal sodium excretion / : / : / renin secretion into blood stream / catechol O-methyltransferase activity / renal sodium excretion / : / : / renin secretion into blood stream /  発生生物学 / 発生生物学 /  カテコール-O-メチルトランスフェラーゼ / カテコール-O-メチルトランスフェラーゼ /  renal filtration / renal albumin absorption / dopamine secretion / negative regulation of dopamine metabolic process / S-adenosylmethionine metabolic process / renal filtration / renal albumin absorption / dopamine secretion / negative regulation of dopamine metabolic process / S-adenosylmethionine metabolic process /  馴化 / artery development / catecholamine metabolic process / 馴化 / artery development / catecholamine metabolic process /  short-term memory / cellular response to phosphate starvation / cerebellar cortex morphogenesis / dopamine catabolic process / norepinephrine metabolic process / glomerulus development / fear response / multicellular organismal reproductive process / short-term memory / cellular response to phosphate starvation / cerebellar cortex morphogenesis / dopamine catabolic process / norepinephrine metabolic process / glomerulus development / fear response / multicellular organismal reproductive process /  synaptic transmission, dopaminergic / cholesterol efflux / response to angiotensin / cellular response to cocaine / estrogen metabolic process / exploration behavior / response to food / response to pain / response to temperature stimulus / dopamine metabolic process / response to corticosterone / prostaglandin metabolic process / glycogen metabolic process / synaptic transmission, dopaminergic / cholesterol efflux / response to angiotensin / cellular response to cocaine / estrogen metabolic process / exploration behavior / response to food / response to pain / response to temperature stimulus / dopamine metabolic process / response to corticosterone / prostaglandin metabolic process / glycogen metabolic process /  startle response / detection of temperature stimulus involved in sensory perception of pain / response to inorganic substance / behavioral fear response / multicellular organismal response to stress / response to amphetamine / startle response / detection of temperature stimulus involved in sensory perception of pain / response to inorganic substance / behavioral fear response / multicellular organismal response to stress / response to amphetamine /  学習 / 学習 /  kidney development / response to organic substance / response to cytokine / female pregnancy / negative regulation of smooth muscle cell proliferation / multicellular organism growth / kidney development / response to organic substance / response to cytokine / female pregnancy / negative regulation of smooth muscle cell proliferation / multicellular organism growth /  visual learning / response to organic cyclic compound / visual learning / response to organic cyclic compound /  記憶 / response to toxic substance / 記憶 / response to toxic substance /  認識 / 認識 /  血圧 / response to wounding / response to estrogen / 血圧 / response to wounding / response to estrogen /  cell body / cell body /  遺伝子発現 / 遺伝子発現 /  メチル化 / メチル化 /  postsynaptic membrane / postsynapse / response to oxidative stress / vesicle / response to lipopolysaccharide / postsynaptic membrane / postsynapse / response to oxidative stress / vesicle / response to lipopolysaccharide /  樹状突起スパイン / response to hypoxia / learning or memory / response to xenobiotic stimulus / 樹状突起スパイン / response to hypoxia / learning or memory / response to xenobiotic stimulus /  神経繊維 / 神経繊維 /  樹状突起 / glutamatergic synapse / magnesium ion binding / 樹状突起 / glutamatergic synapse / magnesium ion binding /  生体膜 / 生体膜 /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||
| 生物種 |   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.99 Å 分子置換 / 解像度: 1.99 Å | ||||||
 データ登録者 データ登録者 | Ehler, A. / Lerner, C. / Rudolph, M.G. | ||||||
 引用 引用 |  ジャーナル: To be published ジャーナル: To be publishedタイトル: Crystal Structure of a COMT complex 著者: Lerner, C. / Jakob-Roetne, R. / Groebke-Zbinden, K. / Buettelmann, B. / Rudolph, M.G. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5pa4.cif.gz 5pa4.cif.gz | 61.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5pa4.ent.gz pdb5pa4.ent.gz | 42.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5pa4.json.gz 5pa4.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/pa/5pa4 https://data.pdbj.org/pub/pdb/validation_reports/pa/5pa4 ftp://data.pdbj.org/pub/pdb/validation_reports/pa/5pa4 ftp://data.pdbj.org/pub/pdb/validation_reports/pa/5pa4 | HTTPS FTP |
|---|
-グループ登録
| ID | G_1002014 (21エントリ) |
|---|---|
| タイトル | Crystal Structure of COMT complex |
| タイプ | undefined |
| 解説 | Crystal Structure of COMT complex |
-関連構造データ
| 類似構造データ |
|---|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 |  カテコール-O-メチルトランスフェラーゼ / Humanized rat COMT カテコール-O-メチルトランスフェラーゼ / Humanized rat COMT分子量: 24694.332 Da / 分子数: 1 / 断片: SOLUBLE FORM, RESIDUES 44-264 / 変異: M134I, Y138C / 由来タイプ: 組換発現 / 由来: (組換発現)   Rattus norvegicus (ドブネズミ) / 遺伝子: Comt / 発現宿主: Rattus norvegicus (ドブネズミ) / 遺伝子: Comt / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3)参照: UniProt: P22734,  カテコール-O-メチルトランスフェラーゼ カテコール-O-メチルトランスフェラーゼ |
|---|
-非ポリマー , 5種, 101分子 







| #2: 化合物 | ChemComp-MG / |
|---|---|
| #3: 化合物 | ChemComp-SAH /  S-アデノシル-L-ホモシステイン S-アデノシル-L-ホモシステイン |
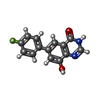
| #4: 化合物 | ChemComp-7JD / |
| #5: 化合物 | ChemComp-SO4 /  硫酸塩 硫酸塩 |
| #6: 水 | ChemComp-HOH /  水 水 |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.22 Å3/Da / 溶媒含有率: 44.59 % |
|---|---|
結晶化 | 温度: 295 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 9 / 詳細: AMMONIUM SULPHATE, CHES, PH 9 |
-データ収集
| 回折 | 平均測定温度: 100 K | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SLS SLS  / ビームライン: X10SA / 波長: 1 Å / ビームライン: X10SA / 波長: 1 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: PSI PILATUS 6M / 検出器: PIXEL / 日付: 2010年10月4日 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長 : 1 Å / 相対比: 1 : 1 Å / 相対比: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 1.99→45.12 Å / Num. obs: 15538 / % possible obs: 99 % / Observed criterion σ(I): -3 / 冗長度: 6.39 % / Biso Wilson estimate: 30.202 Å2 / CC1/2: 0.991 / Rmerge(I) obs: 0.221 / Rrim(I) all: 0.241 / Χ2: 0.946 / Net I/σ(I): 6.89 / Num. measured all: 99295 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1 / Rejects: 0
|
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: inhouse model 解像度: 1.99→45.12 Å / Cor.coef. Fo:Fc: 0.936 / Cor.coef. Fo:Fc free: 0.903 / SU B: 6.274 / SU ML: 0.165 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.234 / ESU R Free: 0.203 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD 詳細: HYDROGENS HAVE BEEN USED IF PRESENT IN THE INPUT U VALUES : REFINED INDIVIDUALLY
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: BABINET MODEL WITH MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 77.07 Å2 / Biso mean: 26.132 Å2 / Biso min: 16.45 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 1.99→45.12 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.99→2.042 Å / Total num. of bins used: 20
|
 ムービー
ムービー コントローラー
コントローラー

































 PDBj
PDBj



