登録情報 データベース : PDB / ID : 5a3wタイトル Crystal structure of human PLU-1 (JARID1B) in complex with Pyridine-2, 6-dicarboxylic Acid (PDCA) LYSINE-SPECIFIC DEMETHYLASE 5B キーワード / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 HOMO SAPIENS (ヒト)手法 / / / 解像度 : 2 Å データ登録者 Srikannathasan, V. / Johansson, C. / Gileadi, C. / Kopec, J. / Strain-Damerell, C. / Kupinska, K. / Burgess-Brown, N.A. / von Delft, F. / Arrowsmith, C.H. / Bountra, C. ...Srikannathasan, V. / Johansson, C. / Gileadi, C. / Kopec, J. / Strain-Damerell, C. / Kupinska, K. / Burgess-Brown, N.A. / von Delft, F. / Arrowsmith, C.H. / Bountra, C. / Edwards, A.M. / Oppermann, U. ジャーナル : Nat.Chem.Biol. / 年 : 2016タイトル : Structural Analysis of Human Kdm5B Guides Histone Demethylase Inhibitor Development.著者: Johansson, C. / Velupillai, S. / Tumber, A. / Szykowska, A. / Hookway, E.S. / Nowak, R.P. / Strain-Damerell, C. / Gileadi, C. / Philpott, M. / Burgess-Brown, N. / Wu, N. / Kopec, J. / Nuzzi, ... 著者 : Johansson, C. / Velupillai, S. / Tumber, A. / Szykowska, A. / Hookway, E.S. / Nowak, R.P. / Strain-Damerell, C. / Gileadi, C. / Philpott, M. / Burgess-Brown, N. / Wu, N. / Kopec, J. / Nuzzi, A. / Steuber, H. / Egner, U. / Badock, V. / Munro, S. / Lathangue, N.B. / Westaway, S. / Brown, J. / Athanasou, N. / Prinjha, R. / Brennan, P.E. / Oppermann, U. 履歴 登録 2015年6月3日 登録サイト / 処理サイト 改定 1.0 2015年6月17日 Provider / タイプ 改定 1.1 2016年5月25日 Group 改定 1.2 2016年6月1日 Group 改定 1.3 2016年6月29日 Group 改定 1.4 2018年1月24日 Group / カテゴリ / Item
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード OXIDOREDUCTASE (酸化還元酵素) / LYSINE-SPECIFIC DEMETHYLASE 5B
OXIDOREDUCTASE (酸化還元酵素) / LYSINE-SPECIFIC DEMETHYLASE 5B 機能・相同性情報
機能・相同性情報 histone binding /
histone binding /  nucleic acid binding /
nucleic acid binding /  クロマチンリモデリング / negative regulation of DNA-templated transcription /
クロマチンリモデリング / negative regulation of DNA-templated transcription /  クロマチン / positive regulation of gene expression / regulation of DNA-templated transcription /
クロマチン / positive regulation of gene expression / regulation of DNA-templated transcription /  DNA binding / zinc ion binding /
DNA binding / zinc ion binding /  核質 /
核質 /  細胞核 /
細胞核 /  細胞質基質
細胞質基質
 HOMO SAPIENS (ヒト)
HOMO SAPIENS (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2 Å
分子置換 / 解像度: 2 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Nat.Chem.Biol. / 年: 2016
ジャーナル: Nat.Chem.Biol. / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5a3w.cif.gz
5a3w.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5a3w.ent.gz
pdb5a3w.ent.gz PDB形式
PDB形式 5a3w.json.gz
5a3w.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/a3/5a3w
https://data.pdbj.org/pub/pdb/validation_reports/a3/5a3w ftp://data.pdbj.org/pub/pdb/validation_reports/a3/5a3w
ftp://data.pdbj.org/pub/pdb/validation_reports/a3/5a3w リンク
リンク 集合体
集合体
 要素
要素
 HOMO SAPIENS (ヒト) / プラスミド: PFB-LIC-BSE / 細胞株 (発現宿主): SF9
HOMO SAPIENS (ヒト) / プラスミド: PFB-LIC-BSE / 細胞株 (発現宿主): SF9
 SPODOPTERA FRUGIPERDA (ツマジロクサヨトウ)
SPODOPTERA FRUGIPERDA (ツマジロクサヨトウ) 酸化還元酵素; 電子対供与作用を持つ; 分子酸素を取り込むないしは分子酸素を還元する; 2-オキソグルタル酸類を片方の電子供与体とする; 酸素分をそれぞれの電子供与体に取り込む
酸化還元酵素; 電子対供与作用を持つ; 分子酸素を取り込むないしは分子酸素を還元する; 2-オキソグルタル酸類を片方の電子供与体とする; 酸素分をそれぞれの電子供与体に取り込む

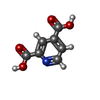












 Lutidinic acid
Lutidinic acid リン酸塩
リン酸塩 HEPES
HEPES エチレングリコール
エチレングリコール 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I03 / 波長: 0.97625
/ ビームライン: I03 / 波長: 0.97625  : 0.97625 Å / 相対比: 1
: 0.97625 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー



































 PDBj
PDBj








