| 登録情報 | データベース: PDB / ID: 4iw3
|
|---|
| タイトル | Crystal structure of a Pseudomonas putida prolyl-4-hydroxylase (P4H) in complex with elongation factor Tu (EF-Tu) |
|---|
 要素 要素 | - Elongation factor Tu-A
- Putative uncharacterized protein
|
|---|
 キーワード キーワード | METAL BINDING PROTEIN/TRANSLATION / 2-oxoglutarate oxygenase / oxygen sensing / protein synthesis regulation / double-stranded beta helix / jellyroll fold / prolyl-4-hydroxylase /  translation (翻訳 (生物学)) / METAL BINDING PROTEIN-TRANSLATION complex translation (翻訳 (生物学)) / METAL BINDING PROTEIN-TRANSLATION complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
 L-ascorbic acid binding / L-ascorbic acid binding /  guanosine tetraphosphate binding / guanosine tetraphosphate binding /  dioxygenase activity / oxidoreductase activity, acting on paired donors, with incorporation or reduction of molecular oxygen / dioxygenase activity / oxidoreductase activity, acting on paired donors, with incorporation or reduction of molecular oxygen /  translation elongation factor activity / iron ion binding / translation elongation factor activity / iron ion binding /  GTPase activity / GTP binding / GTPase activity / GTP binding /  細胞質類似検索 - 分子機能 細胞質類似検索 - 分子機能Prolyl 4-hydroxylase alpha subunit, Fe(2+) 2OG dioxygenase domain / 2OG-Fe(II) oxygenase superfamily /  Prolyl 4-hydroxylase, alpha subunit / Prolyl 4-hydroxylase alpha subunit homologues. / q2cbj1_9rhob like domain / Translation elongation factor EFTu/EF1A, bacterial/organelle / Prolyl 4-hydroxylase, alpha subunit / Prolyl 4-hydroxylase alpha subunit homologues. / q2cbj1_9rhob like domain / Translation elongation factor EFTu/EF1A, bacterial/organelle /  Elongation factor Tu, domain 2 / Elongation factor Tu (EF-Tu), GTP-binding domain / Translation elongation factor EFTu/EF1A, C-terminal / Elongation factor Tu C-terminal domain ...Prolyl 4-hydroxylase alpha subunit, Fe(2+) 2OG dioxygenase domain / 2OG-Fe(II) oxygenase superfamily / Elongation factor Tu, domain 2 / Elongation factor Tu (EF-Tu), GTP-binding domain / Translation elongation factor EFTu/EF1A, C-terminal / Elongation factor Tu C-terminal domain ...Prolyl 4-hydroxylase alpha subunit, Fe(2+) 2OG dioxygenase domain / 2OG-Fe(II) oxygenase superfamily /  Prolyl 4-hydroxylase, alpha subunit / Prolyl 4-hydroxylase alpha subunit homologues. / q2cbj1_9rhob like domain / Translation elongation factor EFTu/EF1A, bacterial/organelle / Prolyl 4-hydroxylase, alpha subunit / Prolyl 4-hydroxylase alpha subunit homologues. / q2cbj1_9rhob like domain / Translation elongation factor EFTu/EF1A, bacterial/organelle /  Elongation factor Tu, domain 2 / Elongation factor Tu (EF-Tu), GTP-binding domain / Translation elongation factor EFTu/EF1A, C-terminal / Elongation factor Tu C-terminal domain / Oxoglutarate/iron-dependent dioxygenase / Fe(2+) 2-oxoglutarate dioxygenase domain profile. / Translation elongation factor EF1A/initiation factor IF2gamma, C-terminal / Translation factors / Tr-type G domain, conserved site / Translational (tr)-type guanine nucleotide-binding (G) domain signature. / Translation elongation factor EFTu-like, domain 2 / Elongation factor Tu domain 2 / Translational (tr)-type GTP-binding domain / Elongation factor Tu GTP binding domain / Translational (tr)-type guanine nucleotide-binding (G) domain profile. / Elongation Factor Tu (Ef-tu); domain 3 / Small GTP-binding protein domain / Elongation factor Tu, domain 2 / Elongation factor Tu (EF-Tu), GTP-binding domain / Translation elongation factor EFTu/EF1A, C-terminal / Elongation factor Tu C-terminal domain / Oxoglutarate/iron-dependent dioxygenase / Fe(2+) 2-oxoglutarate dioxygenase domain profile. / Translation elongation factor EF1A/initiation factor IF2gamma, C-terminal / Translation factors / Tr-type G domain, conserved site / Translational (tr)-type guanine nucleotide-binding (G) domain signature. / Translation elongation factor EFTu-like, domain 2 / Elongation factor Tu domain 2 / Translational (tr)-type GTP-binding domain / Elongation factor Tu GTP binding domain / Translational (tr)-type guanine nucleotide-binding (G) domain profile. / Elongation Factor Tu (Ef-tu); domain 3 / Small GTP-binding protein domain /  Translation protein, beta-barrel domain superfamily / P-loop containing nucleotide triphosphate hydrolases / Jelly Rolls / Translation protein, beta-barrel domain superfamily / P-loop containing nucleotide triphosphate hydrolases / Jelly Rolls /  Βバレル / Βバレル /  サンドイッチ / P-loop containing nucleoside triphosphate hydrolase / サンドイッチ / P-loop containing nucleoside triphosphate hydrolase /  ロスマンフォールド / 3-Layer(aba) Sandwich / Mainly Beta / Alpha Beta類似検索 - ドメイン・相同性 ロスマンフォールド / 3-Layer(aba) Sandwich / Mainly Beta / Alpha Beta類似検索 - ドメイン・相同性 GUANOSINE-5'-DIPHOSPHATE / : /  N-OXALYLGLYCINE / Fe2OG dioxygenase domain-containing protein / Elongation factor Tu-A類似検索 - 構成要素 N-OXALYLGLYCINE / Fe2OG dioxygenase domain-containing protein / Elongation factor Tu-A類似検索 - 構成要素 |
|---|
| 生物種 |   Pseudomonas putida (シュードモナス・プチダ) Pseudomonas putida (シュードモナス・プチダ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.697 Å 分子置換 / 解像度: 2.697 Å |
|---|
 データ登録者 データ登録者 | Scotti, J.S. / McDonough, M.A. / Schofield, C.J. |
|---|
 引用 引用 |  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2014 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2014
タイトル: Human oxygen sensing may have origins in prokaryotic elongation factor Tu prolyl-hydroxylation
著者: Scotti, J.S. / Leung, I.K.H. / Ge, W. / Bentley, M.A. / Paps, J. / Kramer, H.B. / Lee, J. / Aik, W. / Choi, H. / Paulsen, S.M. / Bowman, L.A.H. / Loik, N.D. / Horita, S. / Ho, C.H. / Kershaw, ...著者: Scotti, J.S. / Leung, I.K.H. / Ge, W. / Bentley, M.A. / Paps, J. / Kramer, H.B. / Lee, J. / Aik, W. / Choi, H. / Paulsen, S.M. / Bowman, L.A.H. / Loik, N.D. / Horita, S. / Ho, C.H. / Kershaw, N.J. / Tang, C.M. / Claridge, T.D.W. / Preston, G.M. / McDonough, M.A. / Schofield, C.J. |
|---|
| 履歴 | | 登録 | 2013年1月23日 | 登録サイト: RCSB / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2014年1月29日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2014年3月5日 | Group: Other |
|---|
| 改定 1.2 | 2014年9月17日 | Group: Database references |
|---|
| 改定 1.3 | 2014年10月1日 | Group: Database references |
|---|
| 改定 1.4 | 2019年11月20日 | Group: Data collection / Database references / カテゴリ: reflns / struct_ref_seq_dif
Item: _reflns.pdbx_Rmerge_I_obs / _struct_ref_seq_dif.details |
|---|
| 改定 1.5 | 2023年11月8日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_struct_conn_angle / struct_conn / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_asym_id / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr2_auth_asym_id / _pdbx_struct_conn_angle.ptnr2_auth_comp_id / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr2_label_atom_id / _pdbx_struct_conn_angle.ptnr2_label_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード translation (翻訳 (生物学)) / METAL BINDING PROTEIN-TRANSLATION complex
translation (翻訳 (生物学)) / METAL BINDING PROTEIN-TRANSLATION complex 機能・相同性情報
機能・相同性情報 L-ascorbic acid binding /
L-ascorbic acid binding /  guanosine tetraphosphate binding /
guanosine tetraphosphate binding /  dioxygenase activity / oxidoreductase activity, acting on paired donors, with incorporation or reduction of molecular oxygen /
dioxygenase activity / oxidoreductase activity, acting on paired donors, with incorporation or reduction of molecular oxygen /  translation elongation factor activity / iron ion binding /
translation elongation factor activity / iron ion binding /  GTPase activity / GTP binding /
GTPase activity / GTP binding /  細胞質
細胞質
 Pseudomonas putida (シュードモナス・プチダ)
Pseudomonas putida (シュードモナス・プチダ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.697 Å
分子置換 / 解像度: 2.697 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2014
ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2014 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4iw3.cif.gz
4iw3.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4iw3.ent.gz
pdb4iw3.ent.gz PDB形式
PDB形式 4iw3.json.gz
4iw3.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/iw/4iw3
https://data.pdbj.org/pub/pdb/validation_reports/iw/4iw3 ftp://data.pdbj.org/pub/pdb/validation_reports/iw/4iw3
ftp://data.pdbj.org/pub/pdb/validation_reports/iw/4iw3 リンク
リンク 集合体
集合体


 要素
要素
 Pseudomonas putida (シュードモナス・プチダ)
Pseudomonas putida (シュードモナス・プチダ)
 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: Q88CM1
Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: Q88CM1
 Pseudomonas putida (シュードモナス・プチダ)
Pseudomonas putida (シュードモナス・プチダ)
 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: Q88QP8
Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: Q88QP8
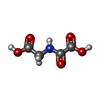







 N-Oxalylglycine
N-Oxalylglycine グアノシン二リン酸
グアノシン二リン酸 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I04 / 波長: 0.9795 Å
/ ビームライン: I04 / 波長: 0.9795 Å : 0.9795 Å / 相対比: 1
: 0.9795 Å / 相対比: 1 
 分子置換
分子置換 解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj








