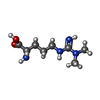
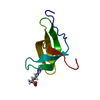
登録情報 データベース : PDB / ID : 4a4hタイトル Solution structure of SPF30 Tudor domain in complex with asymmetrically dimethylated arginine SURVIVAL OF MOTOR NEURON-RELATED-SPLICING FACTOR 30 キーワード 機能・相同性 生物種 HOMO SAPIENS (ヒト)手法 / データ登録者 Tripsianes, K. / Madl, T. / Machyna, M. / Fessas, D. / Englbrecht, C. / Fischer, U. / Neugebauer, K.M. / Sattler, M. ジャーナル : Nat.Struct.Mol.Biol. / 年 : 2011タイトル : Structural Basis for Dimethyl-Arginine Recognition by the Tudor Domains of Human Smn and Spf30 Proteins著者 : Tripsianes, K. / Madl, T. / Machyna, M. / Fessas, D. / Englbrecht, C. / Fischer, U. / Neugebauer, K.M. / Sattler, M. 履歴 登録 2011年10月12日 登録サイト / 処理サイト 改定 1.0 2011年11月30日 Provider / タイプ 改定 1.1 2011年12月14日 Group 改定 1.2 2011年12月28日 Group 改定 1.3 2013年11月6日 Group / Derived calculations改定 2.0 2023年11月15日 Group Atomic model / Data collection ... Atomic model / Data collection / Database references / Derived calculations / Other カテゴリ atom_site / chem_comp_atom ... atom_site / chem_comp_atom / chem_comp_bond / database_2 / pdbx_database_status / pdbx_nmr_spectrometer / struct_site Item _atom_site.Cartn_x / _atom_site.Cartn_y ... _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.auth_atom_id / _atom_site.label_atom_id / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_cs / _pdbx_database_status.status_code_mr / _pdbx_nmr_spectrometer.model / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード RNA BINDING PROTEIN (RNA結合タンパク質)
RNA BINDING PROTEIN (RNA結合タンパク質) 機能・相同性情報
機能・相同性情報 RNA splicing, via transesterification reactions /
RNA splicing, via transesterification reactions /  カハール体 / mRNA Splicing - Major Pathway /
カハール体 / mRNA Splicing - Major Pathway /  spliceosomal complex /
spliceosomal complex /  転写後修飾 / nuclear speck / apoptotic process /
転写後修飾 / nuclear speck / apoptotic process /  RNA binding /
RNA binding /  核質 /
核質 /  細胞核 /
細胞核 /  細胞質
細胞質
 HOMO SAPIENS (ヒト)
HOMO SAPIENS (ヒト) 溶液NMR /
溶液NMR /  simulated annealing
simulated annealing  データ登録者
データ登録者 引用
引用 ジャーナル: Nat.Struct.Mol.Biol. / 年: 2011
ジャーナル: Nat.Struct.Mol.Biol. / 年: 2011 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4a4h.cif.gz
4a4h.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4a4h.ent.gz
pdb4a4h.ent.gz PDB形式
PDB形式 4a4h.json.gz
4a4h.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/a4/4a4h
https://data.pdbj.org/pub/pdb/validation_reports/a4/4a4h ftp://data.pdbj.org/pub/pdb/validation_reports/a4/4a4h
ftp://data.pdbj.org/pub/pdb/validation_reports/a4/4a4h リンク
リンク 集合体
集合体
 要素
要素
 HOMO SAPIENS (ヒト) / 発現宿主:
HOMO SAPIENS (ヒト) / 発現宿主: 
 ESCHERICHIA COLI (大腸菌) / 参照: UniProt: O75940
ESCHERICHIA COLI (大腸菌) / 参照: UniProt: O75940 溶液NMR
溶液NMR 試料調製
試料調製 : AVANCE / 磁場強度: 750 MHz
: AVANCE / 磁場強度: 750 MHz 解析
解析 simulated annealing / ソフトェア番号: 1
simulated annealing / ソフトェア番号: 1  ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj