登録情報 データベース : PDB / ID : 3syuタイトル Re-refined coordinates for pdb entry 1det - ribonuclease T1 carboxymethylated at GLU 58 in complex with 2'GMP Guanyl-specific ribonuclease T1 キーワード / 機能・相同性 生物種 Aspergillus oryzae (米麹菌)手法 / / 解像度 : 1.95 Å データ登録者 Smart, O.S. / Womack, T.O. / Bricogne, G. ジャーナル : Biochemistry / 年 : 1996タイトル : Crystal structure of ribonuclease T1 carboxymethylated at Glu58 in complex with 2'-GMP.著者 : Ishikawa, K. / Suzuki, E. / Tanokura, M. / Takahashi, K. 履歴 登録 2011年7月18日 登録サイト / 処理サイト 改定 1.0 2012年3月28日 Provider / タイプ 改定 1.1 2012年5月2日 Group 改定 1.2 2023年9月13日 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Refinement description カテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_struct_conn_angle / struct_conn / struct_site Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id 改定 2.0 2023年11月15日 Group / Data collectionカテゴリ atom_site / atom_site_anisotrop ... atom_site / atom_site_anisotrop / chem_comp_atom / chem_comp_bond Item _atom_site.auth_atom_id / _atom_site.label_atom_id ... _atom_site.auth_atom_id / _atom_site.label_atom_id / _atom_site_anisotrop.pdbx_auth_atom_id / _atom_site_anisotrop.pdbx_label_atom_id / _chem_comp_atom.atom_id / _chem_comp_bond.atom_id_2
すべて表示 表示を減らす Remark 0 THIS ENTRY 3SYU REFLECTS AN ALTERNATIVE MODELING OF X-RAY DATA IN 1DET. ORIGINAL DATA DETERMINED BY ... THIS ENTRY 3SYU REFLECTS AN ALTERNATIVE MODELING OF X-RAY DATA IN 1DET. ORIGINAL DATA DETERMINED BY AUTHOR: K.ISHIKAWA,E.SUZUKI,M.TANOKURA,K.TAKAHASHI
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード HYDROLASE (加水分解酵素) /
HYDROLASE (加水分解酵素) /  ENDORIBONUCLEASE
ENDORIBONUCLEASE 機能・相同性情報
機能・相同性情報 ribonuclease T1 activity /
ribonuclease T1 activity /  ribonuclease T1 / cell septum /
ribonuclease T1 / cell septum /  endonuclease activity /
endonuclease activity /  lyase activity /
lyase activity /  RNA binding
RNA binding
 Aspergillus oryzae (米麹菌)
Aspergillus oryzae (米麹菌) X線回折 / RE-REFINEMENT / 解像度: 1.95 Å
X線回折 / RE-REFINEMENT / 解像度: 1.95 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Biochemistry / 年: 1996
ジャーナル: Biochemistry / 年: 1996 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3syu.cif.gz
3syu.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3syu.ent.gz
pdb3syu.ent.gz PDB形式
PDB形式 3syu.json.gz
3syu.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/sy/3syu
https://data.pdbj.org/pub/pdb/validation_reports/sy/3syu ftp://data.pdbj.org/pub/pdb/validation_reports/sy/3syu
ftp://data.pdbj.org/pub/pdb/validation_reports/sy/3syu リンク
リンク 集合体
集合体
 要素
要素
 Aspergillus oryzae (米麹菌) / 株: ATCC 42149 / RIB 40 / 参照: UniProt: P00651, EC: 3.1.27.3
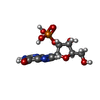
Aspergillus oryzae (米麹菌) / 株: ATCC 42149 / RIB 40 / 参照: UniProt: P00651, EC: 3.1.27.3 グアニル酸
グアニル酸 水
水 X線回折
X線回折 試料調製
試料調製 解析
解析 : RE-REFINEMENT
: RE-REFINEMENT ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj