+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2olc | ||||||
|---|---|---|---|---|---|---|---|
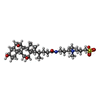
| タイトル | Crystal structure of 5-methylthioribose kinase in complex with ADP-2Ho | ||||||
 要素 要素 | Methylthioribose kinase | ||||||
 キーワード キーワード |  TRANSFERASE (転移酵素) / kinase ADP-2Ho complex TRANSFERASE (転移酵素) / kinase ADP-2Ho complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 S-methyl-5-thioribose kinase / S-methyl-5-thioribose kinase /  S-methyl-5-thioribose kinase activity / L-methionine salvage from S-adenosylmethionine / L-methionine salvage from methylthioadenosine / S-methyl-5-thioribose kinase activity / L-methionine salvage from S-adenosylmethionine / L-methionine salvage from methylthioadenosine /  リン酸化 / リン酸化 /  ATP binding ATP binding類似検索 - 分子機能 | ||||||
| 生物種 |   Bacillus subtilis (枯草菌) Bacillus subtilis (枯草菌) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単波長異常分散 / 解像度: 2 Å 単波長異常分散 / 解像度: 2 Å | ||||||
 データ登録者 データ登録者 | Ku, S.Y. / Smith, G.D. / Howell, P.L. | ||||||
 引用 引用 |  ジャーナル: Acta Crystallogr.,Sect.D / 年: 2007 ジャーナル: Acta Crystallogr.,Sect.D / 年: 2007タイトル: ADP-2Ho as a phasing tool for nucleotide-containing proteins. 著者: Ku, S.Y. / Smith, G.D. / Howell, P.L. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2olc.cif.gz 2olc.cif.gz | 171.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2olc.ent.gz pdb2olc.ent.gz | 132.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2olc.json.gz 2olc.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ol/2olc https://data.pdbj.org/pub/pdb/validation_reports/ol/2olc ftp://data.pdbj.org/pub/pdb/validation_reports/ol/2olc ftp://data.pdbj.org/pub/pdb/validation_reports/ol/2olc | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||||||||||||||||||||
| 2 | 
| ||||||||||||||||||||||||||||||
| 単位格子 |
| ||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: Component-ID: 1 / Ens-ID: 1 / End auth comp-ID: LYS / End label comp-ID: LYS / Refine code: 3
| ||||||||||||||||||||||||||||||
| 詳細 | The biological unit is a dimer in the assymetric unit |
- 要素
要素
| #1: タンパク質 | 分子量: 45139.973 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Bacillus subtilis (枯草菌) / 遺伝子: mtnK / プラスミド: pTRCHis2 / 生物種 (発現宿主): Escherichia coli / 発現宿主: Bacillus subtilis (枯草菌) / 遺伝子: mtnK / プラスミド: pTRCHis2 / 生物種 (発現宿主): Escherichia coli / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: O31663, Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: O31663,  S-methyl-5-thioribose kinase S-methyl-5-thioribose kinase#2: 化合物 | ChemComp-HO / #3: 化合物 | ChemComp-CPS / |  CHAPS detergent CHAPS detergent#4: 化合物 |  アデノシン二リン酸 アデノシン二リン酸#5: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.57 Å3/Da / 溶媒含有率: 52.14 % |
|---|---|
結晶化 | 温度: 298 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7.5 詳細: 22%(w/v) PEG2000MME, 0.1M TrisHCl pH 7.5 and 0.3M sodium acetate, vapor diffusion, hanging drop, temperature 298K |
-データ収集
| 回折 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | 冗長度: 26.94 % / Av σ(I) over netI: 17.1 / 数: 233838 / Rmerge(I) obs: 0.101 / Χ2: 6.55 / D res high: 2.52 Å / D res low: 50 Å / Num. obs: 32188 / % possible obs: 99.3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Diffraction reflection shell | ID: 1
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 2→71.73 Å / Num. obs: 63253 / % possible obs: 98.8 % / Observed criterion σ(I): 5 / 冗長度: 26.94 % / Rmerge(I) obs: 0.093 / Χ2: 0.98 / Net I/σ(I): 21.9 / Scaling rejects: 12878 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1,2
|
-位相決定
位相決定 | 手法:  単波長異常分散 単波長異常分散 | |||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Phasing dm | FOM : 0.77 / FOM acentric: 0.77 / FOM centric: 0.78 / 反射: 32134 / Reflection acentric: 28119 / Reflection centric: 4015 | |||||||||||||||||||||||||||||||||||||||||||||||||
| Phasing dm shell |
|
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  単波長異常分散 / 解像度: 2→107.83 Å / Cor.coef. Fo:Fc: 0.941 / Cor.coef. Fo:Fc free: 0.907 / SU B: 9.094 / SU ML: 0.138 / TLS residual ADP flag: LIKELY RESIDUAL / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.189 / ESU R Free: 0.167 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS 単波長異常分散 / 解像度: 2→107.83 Å / Cor.coef. Fo:Fc: 0.941 / Cor.coef. Fo:Fc free: 0.907 / SU B: 9.094 / SU ML: 0.138 / TLS residual ADP flag: LIKELY RESIDUAL / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.189 / ESU R Free: 0.167 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 27.565 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2→107.83 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | Dom-ID: 1 / Auth asym-ID: A / Ens-ID: 1 / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2→2.052 Å / Total num. of bins used: 20
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ | Refine-ID: X-RAY DIFFRACTION / Selection: ALL
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj