+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2jph | ||||||
|---|---|---|---|---|---|---|---|
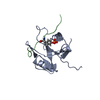
| タイトル | NMR solution structure of the Rho GTPase binding domain of human plexin-b1 | ||||||
 要素 要素 | Plexin-B1 | ||||||
 キーワード キーワード |  SIGNALING PROTEIN / SIGNALING PROTEIN /  PROTEIN BINDING (タンパク質) / PROTEIN BINDING (タンパク質) /  protein (タンパク質) / ubiquitin fold protein (タンパク質) / ubiquitin fold | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報semaphorin receptor binding / semaphorin-plexin signaling pathway involved in axon guidance / negative regulation of osteoblast proliferation /  inhibitory synapse assembly / semaphorin receptor complex / semaphorin receptor activity / negative regulation of cell adhesion / RHOD GTPase cycle / Sema4D induced cell migration and growth-cone collapse / GTPase activating protein binding ...semaphorin receptor binding / semaphorin-plexin signaling pathway involved in axon guidance / negative regulation of osteoblast proliferation / inhibitory synapse assembly / semaphorin receptor complex / semaphorin receptor activity / negative regulation of cell adhesion / RHOD GTPase cycle / Sema4D induced cell migration and growth-cone collapse / GTPase activating protein binding ...semaphorin receptor binding / semaphorin-plexin signaling pathway involved in axon guidance / negative regulation of osteoblast proliferation /  inhibitory synapse assembly / semaphorin receptor complex / semaphorin receptor activity / negative regulation of cell adhesion / RHOD GTPase cycle / Sema4D induced cell migration and growth-cone collapse / GTPase activating protein binding / Sema4D mediated inhibition of cell attachment and migration / ossification involved in bone maturation / regulation of cytoskeleton organization / positive regulation of Rho protein signal transduction / positive regulation of axonogenesis / semaphorin-plexin signaling pathway / inhibitory synapse assembly / semaphorin receptor complex / semaphorin receptor activity / negative regulation of cell adhesion / RHOD GTPase cycle / Sema4D induced cell migration and growth-cone collapse / GTPase activating protein binding / Sema4D mediated inhibition of cell attachment and migration / ossification involved in bone maturation / regulation of cytoskeleton organization / positive regulation of Rho protein signal transduction / positive regulation of axonogenesis / semaphorin-plexin signaling pathway /  regulation of cell migration / regulation of cell migration /  GTPase activator activity / neuron projection morphogenesis / positive regulation of GTPase activity / G alpha (12/13) signalling events / GTPase activator activity / neuron projection morphogenesis / positive regulation of GTPase activity / G alpha (12/13) signalling events /  遊走 / transmembrane signaling receptor activity / regulation of cell shape / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / intracellular signal transduction / extracellular region / 遊走 / transmembrane signaling receptor activity / regulation of cell shape / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / intracellular signal transduction / extracellular region /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  溶液NMR / torsion angle dynamics, 溶液NMR / torsion angle dynamics,  simulated annealing simulated annealing | ||||||
 データ登録者 データ登録者 | Tong, Y. / Buck, M. | ||||||
 引用 引用 |  ジャーナル: Structure / 年: 2008 ジャーナル: Structure / 年: 2008タイトル: Insights into Oncogenic Mutations of Plexin-B1 Based on the Solution Structure of the Rho GTPase Binding Domain 著者: Tong, Y. / Hota, P.K. / Hamaneh, M.B. / Buck, M. #1: ジャーナル: Structure / 年: 2005 タイトル: When monomers are preferred: a strategy for the identification and disruption of weakly oligomerized proteins. 著者: Tong, Y. / Hughes, D. / Placanica, L. / Buck, M. #2: ジャーナル: J.Biomol.NMR / 年: 2005 タイトル: 1H, 15N and 13C Resonance assignments and secondary structure determination reveal that the minimal Rac1 GTPase binding domain of plexin-B1 has a ubiquitin fold. 著者: Tong, Y. / Buck, M. #3: ジャーナル: Science / 年: 2004 タイトル: The Semaphorin 4D receptor Plexin-B1 is a GTPase activating protein for R-Ras. 著者: Oinuma, I. / Ishikawa, Y. / Katoh, H. / Negishi, M. #4: ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2000 タイトル: The semaphorin receptor plexin-B1 specifically interacts with active Rac in a ligand-dependent manner. 著者: Vikis, H.G. / Li, W. / He, Z. / Guan, K.L. #5: ジャーナル: CURR.BIOL. / 年: 2001 タイトル: Plexin-B semaphorin receptors interact directly with active Rac and regulate the actin cytoskeleton by activating Rho. 著者: Driessens, M.H. / Nobes, C.D. / Self, A. / Jordens, I. / Goodman, C.S. / Hall, A. #6:  ジャーナル: J.Biol.Chem. / 年: 2007 ジャーナル: J.Biol.Chem. / 年: 2007タイトル: Binding of Rac1, Rnd1, and RhoD to a novel Rho GTPase interaction motif destabilizes dimerization of the plexin-B1 effector domain 著者: Tong, Y. / Chugha, P. / Hota, P.K. / Alviani, R.S. / Li, M. / Tempel, W. / Shen, L. / Park, H.W. / Buck, M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2jph.cif.gz 2jph.cif.gz | 743.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2jph.ent.gz pdb2jph.ent.gz | 624.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2jph.json.gz 2jph.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/jp/2jph https://data.pdbj.org/pub/pdb/validation_reports/jp/2jph ftp://data.pdbj.org/pub/pdb/validation_reports/jp/2jph ftp://data.pdbj.org/pub/pdb/validation_reports/jp/2jph | HTTPS FTP |
|---|
-関連構造データ
| 類似構造データ | |
|---|---|
| その他のデータベース |
|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 13590.607 Da / 分子数: 1 / 断片: Sequence database residues 1743-1862 / 変異: W1830F / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: PLXNB1, KIAA0407, SEP / 生物種 (発現宿主): Escherichia coli / 発現宿主: Homo sapiens (ヒト) / 遺伝子: PLXNB1, KIAA0407, SEP / 生物種 (発現宿主): Escherichia coli / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21 (DE3) / 参照: UniProt: O43157 Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21 (DE3) / 参照: UniProt: O43157 |
|---|
-実験情報
-実験
| 実験 | 手法:  溶液NMR 溶液NMR | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||
| 試料状態 | イオン強度: 0.1 / pH: 6.8 / 圧: ambient / 温度: 298 K |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: torsion angle dynamics,  simulated annealing / ソフトェア番号: 1 simulated annealing / ソフトェア番号: 1 詳細: Following Nederveen AJ et al., Proteins 2005, 59: 662-672 but in presence of a 10angstrom explicit water shell, final minimization in presence of 4.5 Angstrom explicit water shell | ||||||||||||||||||||||||||||||||
| 代表構造 | 選択基準: fewest violations | ||||||||||||||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the least restraint violations 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj




