登録情報 データベース : PDB / ID : 2cf8タイトル Complex of recombinant human thrombin with an inhibitor HIRUDIN IIIA THROMBIN HEAVY CHAIN THROMBIN LIGHT CHAIN キーワード / / / / / / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 HOMO SAPIENS (ヒト)HIRUDO MEDICINALIS (医用ビル)手法 / / / 解像度 : 1.3 Å データ登録者 Schweizer, E. / Hoffmann-Roeder, A. / Olsen, J.A. / Obst-Sander, U. / Wagner, B. / Kansy, M. / Banner, D.W. / Diederich, F. ジャーナル : Org.Biomol.Chem. / 年 : 2006タイトル : Multipolar Interactions in the D Pocket of Thrombin: Large Differences between Tricyclic Imide and Lactam Inhibitors.著者 : Schweizer, E. / Hoffmann-Roeder, A. / Olsen, J.A. / Seiler, P. / Obst-Sander, U. / Wagner, B. / Kansy, M. / Banner, D.W. / Diederich, F. 履歴 登録 2006年2月17日 登録サイト / 処理サイト 改定 1.0 2006年6月14日 Provider / タイプ 改定 1.1 2011年7月13日 Group Advisory / Atomic model ... Advisory / Atomic model / Database references / Derived calculations / Non-polymer description / Structure summary / Version format compliance 改定 1.2 2012年12月12日 Group 改定 1.3 2013年8月7日 Group / Other改定 1.4 2013年10月23日 Group 改定 1.5 2017年3月29日 Group 改定 1.6 2023年12月13日 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Other / Refinement description カテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_database_status / pdbx_initial_refinement_model / pdbx_struct_conn_angle / struct_conn / struct_conn_type / struct_site Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_sf / _pdbx_struct_conn_angle.ptnr1_PDB_ins_code / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr1_symmetry / _pdbx_struct_conn_angle.ptnr2_auth_comp_id / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr2_label_atom_id / _pdbx_struct_conn_angle.ptnr2_label_comp_id / _pdbx_struct_conn_angle.ptnr3_PDB_ins_code / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.ptnr3_symmetry / _pdbx_struct_conn_angle.value / _struct_conn.conn_type_id / _struct_conn.id / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.pdbx_ptnr1_PDB_ins_code / _struct_conn.pdbx_ptnr2_PDB_ins_code / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr1_symmetry / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_conn.ptnr2_symmetry / _struct_conn_type.id / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id
すべて表示 表示を減らす Remark 700 SHEET DETERMINATION METHOD: DSSP THE SHEETS PRESENTED AS "HB" IN EACH CHAIN ON SHEET RECORDS BELOW ... SHEET DETERMINATION METHOD: DSSP THE SHEETS PRESENTED AS "HB" IN EACH CHAIN ON SHEET RECORDS BELOW IS ACTUALLY A 6-STRANDED BARREL THIS IS REPRESENTED BY A 7-STRANDED SHEET IN WHICH THE FIRST AND LAST STRANDS ARE IDENTICAL.
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード ACUTE PHASE (急性期タンパク質) /
ACUTE PHASE (急性期タンパク質) /  BLOOD COAGULATION (凝固・線溶系) / CALCIUM-BINDING /
BLOOD COAGULATION (凝固・線溶系) / CALCIUM-BINDING /  GLYCOPROTEIN (糖タンパク質) / HYDOLASE /
GLYCOPROTEIN (糖タンパク質) / HYDOLASE /  SERINE PROTEASE (セリンプロテアーゼ) /
SERINE PROTEASE (セリンプロテアーゼ) /  SERINE PROTEASE INHIBITOR COMPLEX (セルピン) / COMPLEX HYDROLASE-INHIBITOR / HYDROLASE-HYDROLASE INHIBITOR COMPLEX
SERINE PROTEASE INHIBITOR COMPLEX (セルピン) / COMPLEX HYDROLASE-INHIBITOR / HYDROLASE-HYDROLASE INHIBITOR COMPLEX 機能・相同性情報
機能・相同性情報 トロンビン / neutrophil-mediated killing of gram-negative bacterium /
トロンビン / neutrophil-mediated killing of gram-negative bacterium /  regulation of blood coagulation / ligand-gated ion channel signaling pathway / Defective F8 cleavage by thrombin ...positive regulation of lipid kinase activity / positive regulation of phospholipase C-activating G protein-coupled receptor signaling pathway / cytolysis by host of symbiont cells / thrombospondin receptor activity / Defective factor XII causes hereditary angioedema /
regulation of blood coagulation / ligand-gated ion channel signaling pathway / Defective F8 cleavage by thrombin ...positive regulation of lipid kinase activity / positive regulation of phospholipase C-activating G protein-coupled receptor signaling pathway / cytolysis by host of symbiont cells / thrombospondin receptor activity / Defective factor XII causes hereditary angioedema /  トロンビン / neutrophil-mediated killing of gram-negative bacterium /
トロンビン / neutrophil-mediated killing of gram-negative bacterium /  regulation of blood coagulation / ligand-gated ion channel signaling pathway / Defective F8 cleavage by thrombin / Platelet Aggregation (Plug Formation) / negative regulation of platelet activation / negative regulation of astrocyte differentiation / positive regulation of collagen biosynthetic process / negative regulation of cytokine production involved in inflammatory response / positive regulation of blood coagulation / negative regulation of fibrinolysis / Gamma-carboxylation of protein precursors / Transport of gamma-carboxylated protein precursors from the endoplasmic reticulum to the Golgi apparatus / Common Pathway of Fibrin Clot Formation / Removal of aminoterminal propeptides from gamma-carboxylated proteins /
regulation of blood coagulation / ligand-gated ion channel signaling pathway / Defective F8 cleavage by thrombin / Platelet Aggregation (Plug Formation) / negative regulation of platelet activation / negative regulation of astrocyte differentiation / positive regulation of collagen biosynthetic process / negative regulation of cytokine production involved in inflammatory response / positive regulation of blood coagulation / negative regulation of fibrinolysis / Gamma-carboxylation of protein precursors / Transport of gamma-carboxylated protein precursors from the endoplasmic reticulum to the Golgi apparatus / Common Pathway of Fibrin Clot Formation / Removal of aminoterminal propeptides from gamma-carboxylated proteins /  fibrinolysis / regulation of cytosolic calcium ion concentration / Intrinsic Pathway of Fibrin Clot Formation / Peptide ligand-binding receptors / positive regulation of release of sequestered calcium ion into cytosol /
fibrinolysis / regulation of cytosolic calcium ion concentration / Intrinsic Pathway of Fibrin Clot Formation / Peptide ligand-binding receptors / positive regulation of release of sequestered calcium ion into cytosol /  Regulation of Complement cascade / acute-phase response / Cell surface interactions at the vascular wall /
Regulation of Complement cascade / acute-phase response / Cell surface interactions at the vascular wall /  lipopolysaccharide binding / negative regulation of proteolysis / positive regulation of receptor signaling pathway via JAK-STAT /
lipopolysaccharide binding / negative regulation of proteolysis / positive regulation of receptor signaling pathway via JAK-STAT /  growth factor activity / serine-type endopeptidase inhibitor activity / positive regulation of insulin secretion /
growth factor activity / serine-type endopeptidase inhibitor activity / positive regulation of insulin secretion /  凝固・線溶系 / response to wounding / Golgi lumen / positive regulation of protein localization to nucleus / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) / positive regulation of reactive oxygen species metabolic process /
凝固・線溶系 / response to wounding / Golgi lumen / positive regulation of protein localization to nucleus / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) / positive regulation of reactive oxygen species metabolic process /  凝固・線溶系 / antimicrobial humoral immune response mediated by antimicrobial peptide / Thrombin signalling through proteinase activated receptors (PARs) /
凝固・線溶系 / antimicrobial humoral immune response mediated by antimicrobial peptide / Thrombin signalling through proteinase activated receptors (PARs) /  heparin binding / regulation of cell shape / positive regulation of cell growth / G alpha (q) signalling events / collagen-containing extracellular matrix / blood microparticle / cell surface receptor signaling pathway / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / positive regulation of protein phosphorylation / G protein-coupled receptor signaling pathway /
heparin binding / regulation of cell shape / positive regulation of cell growth / G alpha (q) signalling events / collagen-containing extracellular matrix / blood microparticle / cell surface receptor signaling pathway / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / positive regulation of protein phosphorylation / G protein-coupled receptor signaling pathway /  小胞体 /
小胞体 /  signaling receptor binding / serine-type endopeptidase activity /
signaling receptor binding / serine-type endopeptidase activity /  calcium ion binding / positive regulation of cell population proliferation /
calcium ion binding / positive regulation of cell population proliferation /  タンパク質分解 /
タンパク質分解 /  extracellular space / extracellular exosome / extracellular region /
extracellular space / extracellular exosome / extracellular region /  細胞膜
細胞膜
 HOMO SAPIENS (ヒト)
HOMO SAPIENS (ヒト)
 HIRUDO MEDICINALIS (医用ビル)
HIRUDO MEDICINALIS (医用ビル) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  フーリエ合成 / 解像度: 1.3 Å
フーリエ合成 / 解像度: 1.3 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Org.Biomol.Chem. / 年: 2006
ジャーナル: Org.Biomol.Chem. / 年: 2006 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2cf8.cif.gz
2cf8.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2cf8.ent.gz
pdb2cf8.ent.gz PDB形式
PDB形式 2cf8.json.gz
2cf8.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/cf/2cf8
https://data.pdbj.org/pub/pdb/validation_reports/cf/2cf8 ftp://data.pdbj.org/pub/pdb/validation_reports/cf/2cf8
ftp://data.pdbj.org/pub/pdb/validation_reports/cf/2cf8 リンク
リンク 集合体
集合体
 要素
要素 トロンビン
トロンビン
 HOMO SAPIENS (ヒト) / 細胞株 (発現宿主): OVARY
HOMO SAPIENS (ヒト) / 細胞株 (発現宿主): OVARY
 CRICETULUS GRISEUS (モンゴルキヌゲネズミ)
CRICETULUS GRISEUS (モンゴルキヌゲネズミ) トロンビン
トロンビン トロンビン
トロンビン
 HOMO SAPIENS (ヒト) / 細胞株 (発現宿主): OVARY
HOMO SAPIENS (ヒト) / 細胞株 (発現宿主): OVARY
 CRICETULUS GRISEUS (モンゴルキヌゲネズミ)
CRICETULUS GRISEUS (モンゴルキヌゲネズミ) トロンビン
トロンビン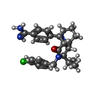






 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  SLS
SLS  / ビームライン: X10SA / 波長: 0.97001
/ ビームライン: X10SA / 波長: 0.97001  : 0.97001 Å / 相対比: 1
: 0.97001 Å / 相対比: 1  解析
解析 :
:  フーリエ合成
フーリエ合成 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj