登録情報 データベース : PDB / ID : 1rg6タイトル Solution structure of the C-terminal domain of p63 second splice variant p63 キーワード / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / データ登録者 Cadot, B. / Candi, E. / Cicero, D.O. / Desideri, A. / Mele, S. / Melino, G. / Paci, M. ジャーナル : To be Published タイトル : Solution structure of the C-terminal domain of p63著者 : Cadot, B. / Candi, E. / Cicero, D.O. / Desideri, A. / Mele, S. / Melino, G. / Paci, M. 履歴 登録 2003年11月11日 登録サイト / 処理サイト 改定 1.0 2004年11月23日 Provider / タイプ 改定 1.1 2008年4月29日 Group 改定 1.2 2011年7月13日 Group 改定 1.3 2022年3月2日 Group / Database references / Derived calculationsカテゴリ database_2 / pdbx_nmr_software ... database_2 / pdbx_nmr_software / pdbx_nmr_spectrometer / pdbx_struct_assembly / pdbx_struct_oper_list / struct_ref_seq_dif Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name / _pdbx_nmr_spectrometer.model / _struct_ref_seq_dif.details
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード GENE REGULATION (遺伝子発現の調節) / P73 SAM-LIKE DOMAIN
GENE REGULATION (遺伝子発現の調節) / P73 SAM-LIKE DOMAIN 機能・相同性情報
機能・相同性情報 WW domain binding / cranial skeletal system development /
WW domain binding / cranial skeletal system development /  sympathetic nervous system development / post-anal tail morphogenesis / embryonic forelimb morphogenesis / embryonic hindlimb morphogenesis / TP53 Regulates Transcription of Death Receptors and Ligands / Activation of PUMA and translocation to mitochondria / Regulation of TP53 Activity through Association with Co-factors / hair follicle morphogenesis / positive regulation of Notch signaling pathway / regulation of epidermal cell division / positive regulation of stem cell proliferation / TP53 regulates transcription of several additional cell death genes whose specific roles in p53-dependent apoptosis remain uncertain / TP53 Regulates Transcription of Caspase Activators and Caspases / epithelial cell development / odontogenesis of dentin-containing tooth / negative regulation of cellular senescence / TP53 Regulates Transcription of Genes Involved in Cytochrome C Release / keratinocyte proliferation / intrinsic apoptotic signaling pathway in response to DNA damage by p53 class mediator / positive regulation of osteoblast differentiation / MDM2/MDM4 family protein binding / establishment of skin barrier /
sympathetic nervous system development / post-anal tail morphogenesis / embryonic forelimb morphogenesis / embryonic hindlimb morphogenesis / TP53 Regulates Transcription of Death Receptors and Ligands / Activation of PUMA and translocation to mitochondria / Regulation of TP53 Activity through Association with Co-factors / hair follicle morphogenesis / positive regulation of Notch signaling pathway / regulation of epidermal cell division / positive regulation of stem cell proliferation / TP53 regulates transcription of several additional cell death genes whose specific roles in p53-dependent apoptosis remain uncertain / TP53 Regulates Transcription of Caspase Activators and Caspases / epithelial cell development / odontogenesis of dentin-containing tooth / negative regulation of cellular senescence / TP53 Regulates Transcription of Genes Involved in Cytochrome C Release / keratinocyte proliferation / intrinsic apoptotic signaling pathway in response to DNA damage by p53 class mediator / positive regulation of osteoblast differentiation / MDM2/MDM4 family protein binding / establishment of skin barrier /  Pyroptosis / positive regulation of apoptotic signaling pathway /
Pyroptosis / positive regulation of apoptotic signaling pathway /  Notchシグナリング / keratinocyte differentiation /
Notchシグナリング / keratinocyte differentiation /  skeletal system development / stem cell proliferation / determination of adult lifespan / TP53 Regulates Metabolic Genes / protein tetramerization / RNA polymerase II transcription regulatory region sequence-specific DNA binding /
skeletal system development / stem cell proliferation / determination of adult lifespan / TP53 Regulates Metabolic Genes / protein tetramerization / RNA polymerase II transcription regulatory region sequence-specific DNA binding /  細胞老化 /
細胞老化 /  p53 binding /
p53 binding /  精子形成 / DNA-binding transcription activator activity, RNA polymerase II-specific / neuron apoptotic process / transcription by RNA polymerase II / damaged DNA binding / DNA-binding transcription factor activity, RNA polymerase II-specific /
精子形成 / DNA-binding transcription activator activity, RNA polymerase II-specific / neuron apoptotic process / transcription by RNA polymerase II / damaged DNA binding / DNA-binding transcription factor activity, RNA polymerase II-specific /  クロマチンリモデリング / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity / negative regulation of DNA-templated transcription /
クロマチンリモデリング / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity / negative regulation of DNA-templated transcription /  樹状突起 / apoptotic process / DNA damage response /
樹状突起 / apoptotic process / DNA damage response /  chromatin binding /
chromatin binding /  クロマチン / regulation of transcription by RNA polymerase II / positive regulation of DNA-templated transcription / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II /
クロマチン / regulation of transcription by RNA polymerase II / positive regulation of DNA-templated transcription / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II /  DNA binding /
DNA binding /  核質 / identical protein binding /
核質 / identical protein binding /  metal ion binding /
metal ion binding /  細胞核 /
細胞核 /  細胞質
細胞質
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 溶液NMR /
溶液NMR /  simulated annealing
simulated annealing  データ登録者
データ登録者 引用
引用 ジャーナル: To be Published
ジャーナル: To be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1rg6.cif.gz
1rg6.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1rg6.ent.gz
pdb1rg6.ent.gz PDB形式
PDB形式 1rg6.json.gz
1rg6.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/rg/1rg6
https://data.pdbj.org/pub/pdb/validation_reports/rg/1rg6 ftp://data.pdbj.org/pub/pdb/validation_reports/rg/1rg6
ftp://data.pdbj.org/pub/pdb/validation_reports/rg/1rg6 リンク
リンク 集合体
集合体
 要素
要素
 Homo sapiens (ヒト) / 遺伝子: p63 / プラスミド: pGEXsx2 / 生物種 (発現宿主): Escherichia coli / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: p63 / プラスミド: pGEXsx2 / 生物種 (発現宿主): Escherichia coli / 発現宿主: 
 Escherichia coli BL21 (大腸菌) / 株 (発現宿主): BL21 / 参照: UniProt: Q9H3D4
Escherichia coli BL21 (大腸菌) / 株 (発現宿主): BL21 / 参照: UniProt: Q9H3D4 溶液NMR
溶液NMR 試料調製
試料調製 解析
解析 simulated annealing / ソフトェア番号: 1
simulated annealing / ソフトェア番号: 1  ムービー
ムービー コントローラー
コントローラー



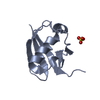



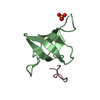
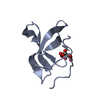


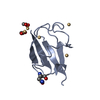

 PDBj
PDBj








