+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1myh | ||||||
|---|---|---|---|---|---|---|---|
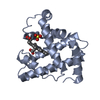
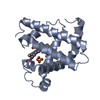
| タイトル | HIGH RESOLUTION X-RAY STRUCTURES OF PIG METMYOGLOBIN AND TWO CD3 MUTANTS MB(LYS45-> ARG) AND MB(LYS45-> SER) | ||||||
 要素 要素 | MYOGLOBIN ミオグロビン ミオグロビン | ||||||
 キーワード キーワード |  OXYGEN STORAGE OXYGEN STORAGE | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Intracellular oxygen transport /  nitrite reductase activity / nitrite reductase activity /  酸化還元酵素; 他の含窒素化合物が電子供与する / 酸化還元酵素; 他の含窒素化合物が電子供与する /  血液 / 血液 /  筋形質 / 筋形質 /  酸化還元酵素; 過酸化物を電子受容体にする; ペルオキシダーゼ / removal of superoxide radicals / 酸化還元酵素; 過酸化物を電子受容体にする; ペルオキシダーゼ / removal of superoxide radicals /  peroxidase activity / peroxidase activity /  oxygen carrier activity / oxygen carrier activity /  oxygen binding ...Intracellular oxygen transport / oxygen binding ...Intracellular oxygen transport /  nitrite reductase activity / nitrite reductase activity /  酸化還元酵素; 他の含窒素化合物が電子供与する / 酸化還元酵素; 他の含窒素化合物が電子供与する /  血液 / 血液 /  筋形質 / 筋形質 /  酸化還元酵素; 過酸化物を電子受容体にする; ペルオキシダーゼ / removal of superoxide radicals / 酸化還元酵素; 過酸化物を電子受容体にする; ペルオキシダーゼ / removal of superoxide radicals /  peroxidase activity / peroxidase activity /  oxygen carrier activity / oxygen carrier activity /  oxygen binding / oxygen binding /  heme binding / heme binding /  metal ion binding metal ion binding類似検索 - 分子機能 | ||||||
| 生物種 |   Sus scrofa (ブタ) Sus scrofa (ブタ) | ||||||
| 手法 |  X線回折 / 解像度: 1.9 Å X線回折 / 解像度: 1.9 Å | ||||||
 データ登録者 データ登録者 | Smerdon, S.J. / Oldfield, T.J. / Wilkinson, A.J. / Dauter, Z. / Petratos, K. / Wilson, K.S. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 1992 ジャーナル: Biochemistry / 年: 1992タイトル: High-resolution X-ray structures of pig metmyoglobin and two CD3 mutants: Mb(Lys45----Arg) and Mb(Lys45----Ser). 著者: Oldfield, T.J. / Smerdon, S.J. / Dauter, Z. / Petratos, K. / Wilson, K.S. / Wilkinson, A.J. #1:  ジャーナル: Acta Crystallogr.,Sect.B / 年: 1990 ジャーナル: Acta Crystallogr.,Sect.B / 年: 1990タイトル: Determination of the Crystal Structure of Recombinant Pig Myoglobin by Molecular Replacement and its Refinement 著者: Smerdon, S.J. / Oldfield, T.J. / Dodson, E.J. / Dodson, G.G. / Hubbard, R.E. / Wilkinson, A.J. #2:  ジャーナル: Protein Eng. / 年: 1988 ジャーナル: Protein Eng. / 年: 1988タイトル: Apomyoglobin as a Molecular Recognition Surface: Expression, Reconstitution and Crystallisation of Recombinant Porcine Myoglobin in Escherichia Coli 著者: Dodson, G.G. / Hubbard, R.E. / Oldfield, T.J. / Smerdon, S.J. / Wilkinson, A.J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1myh.cif.gz 1myh.cif.gz | 79.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1myh.ent.gz pdb1myh.ent.gz | 58.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1myh.json.gz 1myh.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/my/1myh https://data.pdbj.org/pub/pdb/validation_reports/my/1myh ftp://data.pdbj.org/pub/pdb/validation_reports/my/1myh ftp://data.pdbj.org/pub/pdb/validation_reports/my/1myh | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
| ||||||||
| Atom site foot note | 1: RESIDUES 152 AND 153 ARE DISORDERED AND NOT DEFINED IN DENSITY MAPS. 2: HOH 48 AND HOH 58 ARE WATER MOLECULES IN THE HEME POCKET LIGATED TO THE HEME IRON AT DISTANCES OF 2.49 AND 2.33 ANGSTROMS, RESPECTIVELY. |
- 要素
要素
| #1: タンパク質 |  ミオグロビン ミオグロビン分子量: 17011.461 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Sus scrofa (ブタ) / 参照: UniProt: P02189 Sus scrofa (ブタ) / 参照: UniProt: P02189#2: 化合物 |  硫酸塩 硫酸塩#3: 化合物 |  Heme B Heme B#4: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.64 Å3/Da / 溶媒含有率: 66.22 % | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
結晶化 | *PLUS 温度: 15 ℃ / pH: 7.1 / 手法: 蒸気拡散法, ハンギングドロップ法詳細: taken from Dodson, G. et al(1975). Nature, 257, 758. | ||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 放射 | 散乱光タイプ: x-ray |
|---|---|
| 放射波長 | 相対比: 1 |
| 反射 | *PLUS 最高解像度: 1.9 Å / Num. obs: 27123 / Num. measured all: 37845 / Rmerge(I) obs: 0.114 |
- 解析
解析
| ソフトウェア | 名称: PROLSQ / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 1.9→10 Å /
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.9→10 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: PROLSQ / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS Rfactor obs: 0.227 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS |
 ムービー
ムービー コントローラー
コントローラー























 PDBj
PDBj








