+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1jqt | ||||||
|---|---|---|---|---|---|---|---|
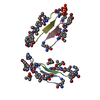
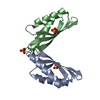
| タイトル | Fitting of L11 protein in the low resolution cryo-EM map of E.coli 70S ribosome | ||||||
 要素 要素 | 50S Ribosomal protein L11 | ||||||
 キーワード キーワード |  RIBOSOME (リボソーム) / L11 / RIBOSOME (リボソーム) / L11 /  cryo-EM (低温電子顕微鏡法) / 70S E.coli ribosome cryo-EM (低温電子顕微鏡法) / 70S E.coli ribosome | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報large ribosomal subunit rRNA binding / cytosolic large ribosomal subunit / structural constituent of ribosome /  翻訳 (生物学) 翻訳 (生物学)類似検索 - 分子機能 | ||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | ||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / Molecular Modeling based on crystal structures / 単粒子再構成法 / Molecular Modeling based on crystal structures /  クライオ電子顕微鏡法 / 解像度: 18 Å クライオ電子顕微鏡法 / 解像度: 18 Å | ||||||
 データ登録者 データ登録者 | Agrawal, R.K. / Linde, J. / Segupta, J. / Nierhaus, K.H. / Frank, J. | ||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2001 ジャーナル: J Mol Biol / 年: 2001タイトル: Localization of L11 protein on the ribosome and elucidation of its involvement in EF-G-dependent translocation. 著者: R K Agrawal / J Linde / J Sengupta / K H Nierhaus / J Frank /  要旨: L11 protein is located at the base of the L7/L12 stalk of the 50 S subunit of the Escherichia coli ribosome. Because of the flexible nature of the region, recent X-ray crystallographic studies of the ...L11 protein is located at the base of the L7/L12 stalk of the 50 S subunit of the Escherichia coli ribosome. Because of the flexible nature of the region, recent X-ray crystallographic studies of the 50 S subunit failed to locate the N-terminal domain of the protein. We have determined the position of the complete L11 protein by comparing a three-dimensional cryo-EM reconstruction of the 70 S ribosome, isolated from a mutant lacking ribosomal protein L11, with the three-dimensional map of the wild-type ribosome. Fitting of the X-ray coordinates of L11-23 S RNA complex and EF-G into the cryo-EM maps combined with molecular modeling, reveals that, following EF-G-dependent GTP hydrolysis, domain V of EF-G intrudes into the cleft between the 23 S ribosomal RNA and the N-terminal domain of L11 (where the antibiotic thiostrepton binds), causing the N-terminal domain to move and thereby inducing the formation of the arc-like connection with the G' domain of EF-G. The results provide a new insight into the mechanism of EF-G-dependent translocation. #1:  ジャーナル: Cell(Cambridge,Mass.) / 年: 1999 ジャーナル: Cell(Cambridge,Mass.) / 年: 1999タイトル: A Detailed View of a Ribosomal Active Site: The Structure of the L11-RNA Complex 著者: Wimberly, B.T. / Guymon, R. / McCutcheon, J.P. / White, S.W. / Ramakrishnan, V. #2:  ジャーナル: Cell(Cambridge,Mass.) / 年: 2000 ジャーナル: Cell(Cambridge,Mass.) / 年: 2000タイトル: EF-G-dependent GTP hydrolysis induces solution structure of the E. coli 70S ribosome at 11.5A resolution 著者: Gabashvili, I.S. / Agrawal, R.K. / Spahn, C.M.T. / Grassucci, R.A. / Svergun, D.I. / Frank, J. / Penczek, P. #3:  ジャーナル: Nat.Struct.Biol. / 年: 1999 ジャーナル: Nat.Struct.Biol. / 年: 1999タイトル: EF-G-dependent GTP hydrolysis induces translocation accompanied by large conformational changes in the 70S ribosome 著者: Agrawal, R.K. / Heagle, A.B. / Penczek, P. / Grassucci, R.A. / Frank, J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1jqt.cif.gz 1jqt.cif.gz | 15 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1jqt.ent.gz pdb1jqt.ent.gz | 6.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1jqt.json.gz 1jqt.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/jq/1jqt https://data.pdbj.org/pub/pdb/validation_reports/jq/1jqt ftp://data.pdbj.org/pub/pdb/validation_reports/jq/1jqt ftp://data.pdbj.org/pub/pdb/validation_reports/jq/1jqt | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 |  / 座標モデル: Cα原子のみ / 座標モデル: Cα原子のみ分子量: 14865.637 Da / 分子数: 1 / 由来タイプ: 天然 詳細: L11 from E. coli 70S ribosome modeled by crystal structure of L11 from Thermatogoma maritima 由来: (天然)   Escherichia coli (大腸菌) / 参照: UniProt: P29395 Escherichia coli (大腸菌) / 参照: UniProt: P29395 |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
| NMR実験の詳細 | Text: This structure was generated by fitting the X-ray crystal structure of L11 into the 70S E. coli ribosome map. L11 (linker region between N and C terminal) was modeled to accommodate the observed densities. |
- 試料調製
試料調製
| 構成要素 | 名称: 70S ribosome リボソーム / タイプ: RIBOSOME リボソーム / タイプ: RIBOSOME |
|---|---|
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
結晶化 | *PLUS 手法:  電子顕微鏡法 電子顕微鏡法 |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 顕微鏡 | モデル: FEI/PHILIPS EM420 |
|---|---|
| 電子銃 | 電子線源 : その他 / 照射モード: FLOOD BEAM : その他 / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 52000 X Bright-field microscopy / 倍率(公称値): 52000 X |
| 試料ホルダ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 撮影 | 電子線照射量: 10 e/Å2 / フィルム・検出器のモデル: GENERIC FILM |
- 解析
解析
| 対称性 | 点対称性 : C1 (非対称) : C1 (非対称) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
3次元再構成 | 解像度: 18 Å / 解像度の算出法: FSC 0.5 CUT-OFF / 粒子像の数: 36113 / 対称性のタイプ: POINT | ||||||||||||||||||||
| 原子モデル構築 | プロトコル: OTHER / 空間: REAL / Target criteria: VISUAL AGREEMENT 詳細: REFINEMENT PROTOCOL--MANUAL DETAILS--This structure was generated by fitting the X-ray crystal structure of L11 into the 70S E. coli ribosome map. L11 (linker region between N and C terminal) ...詳細: REFINEMENT PROTOCOL--MANUAL DETAILS--This structure was generated by fitting the X-ray crystal structure of L11 into the 70S E. coli ribosome map. L11 (linker region between N and C terminal) was modeled to accommodate the observed densities. Conformational changes occur in protein L11 and EF-G due to the binding of EF-G to the 70S ribosome. These changed conformations were modeled based on the fitting of the crystal coordinates to the low resolution ribosome map (factor-bound) and energy minimizing the fitted structures. | ||||||||||||||||||||
| 精密化ステップ | サイクル: LAST
| ||||||||||||||||||||
| NMR software |
| ||||||||||||||||||||
| 精密化 | 手法: Molecular Modeling based on crystal structures / ソフトェア番号: 1 詳細: Conformational changes occur in protein L11 when it is bound to 70S ribosome. This changed conformation was modeled based on the fitting of the crystal coordinates to the low resolution ...詳細: Conformational changes occur in protein L11 when it is bound to 70S ribosome. This changed conformation was modeled based on the fitting of the crystal coordinates to the low resolution ribosome map and energy minimizing the fitted structures. |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj





