| 登録情報 | データベース: PDB / ID: 1a0b
|
|---|
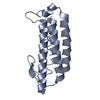
| タイトル | HISTIDINE-CONTAINING PHOSPHOTRANSFER DOMAIN OF ARCB FROM ESCHERICHIA COLI |
|---|
 要素 要素 | AEROBIC RESPIRATION CONTROL SENSOR PROTEIN ARCB |
|---|
 キーワード キーワード |  HISTIDINE KINASE / PHOSPHOTRANSFER / HISTIDINE KINASE / PHOSPHOTRANSFER /  TWO-COMPONENT SYSTEM / TWO-COMPONENT SYSTEM /  FOUR-HELIX BUNDLE (ヘリックスバンドル) FOUR-HELIX BUNDLE (ヘリックスバンドル) |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
peptidyl-histidine phosphorylation / response to oxygen levels /  histidine kinase / phosphorelay sensor kinase activity / plasma membrane => GO:0005886 / histidine kinase / phosphorelay sensor kinase activity / plasma membrane => GO:0005886 /  phosphoprotein phosphatase activity / protein autophosphorylation / regulation of DNA-templated transcription / phosphoprotein phosphatase activity / protein autophosphorylation / regulation of DNA-templated transcription /  シグナル伝達 / シグナル伝達 /  ATP binding ...peptidyl-histidine phosphorylation / response to oxygen levels / ATP binding ...peptidyl-histidine phosphorylation / response to oxygen levels /  histidine kinase / phosphorelay sensor kinase activity / plasma membrane => GO:0005886 / histidine kinase / phosphorelay sensor kinase activity / plasma membrane => GO:0005886 /  phosphoprotein phosphatase activity / protein autophosphorylation / regulation of DNA-templated transcription / phosphoprotein phosphatase activity / protein autophosphorylation / regulation of DNA-templated transcription /  シグナル伝達 / シグナル伝達 /  ATP binding / ATP binding /  細胞膜 / 細胞膜 /  細胞質基質類似検索 - 分子機能 細胞質基質類似検索 - 分子機能  Signal transduction histidine kinase, hybrid-type, aerobic respiration control ArcB / Aerobic respiration control sensor protein ArcB, transmembrane domain superfamily / Histidine kinase receptor ArcB, transmembrane domain / Histidine kinase receptor ArcB trans-membrane domain / HPT domain / Histidine Phosphotransfer domain / Hpt domain / Histidine-containing phosphotransfer (HPt) domain profile. / Signal transduction histidine kinase, hybrid-type, aerobic respiration control ArcB / Aerobic respiration control sensor protein ArcB, transmembrane domain superfamily / Histidine kinase receptor ArcB, transmembrane domain / Histidine kinase receptor ArcB trans-membrane domain / HPT domain / Histidine Phosphotransfer domain / Hpt domain / Histidine-containing phosphotransfer (HPt) domain profile. /  Signal transduction histidine kinase, phosphotransfer (Hpt) domain / HPT domain superfamily ... Signal transduction histidine kinase, phosphotransfer (Hpt) domain / HPT domain superfamily ... Signal transduction histidine kinase, hybrid-type, aerobic respiration control ArcB / Aerobic respiration control sensor protein ArcB, transmembrane domain superfamily / Histidine kinase receptor ArcB, transmembrane domain / Histidine kinase receptor ArcB trans-membrane domain / HPT domain / Histidine Phosphotransfer domain / Hpt domain / Histidine-containing phosphotransfer (HPt) domain profile. / Signal transduction histidine kinase, hybrid-type, aerobic respiration control ArcB / Aerobic respiration control sensor protein ArcB, transmembrane domain superfamily / Histidine kinase receptor ArcB, transmembrane domain / Histidine kinase receptor ArcB trans-membrane domain / HPT domain / Histidine Phosphotransfer domain / Hpt domain / Histidine-containing phosphotransfer (HPt) domain profile. /  Signal transduction histidine kinase, phosphotransfer (Hpt) domain / HPT domain superfamily / PAS-associated, C-terminal / PAC domain profile. / His Kinase A (phospho-acceptor) domain / His Kinase A (phosphoacceptor) domain / Signal transduction histidine kinase, phosphotransfer (Hpt) domain / HPT domain superfamily / PAS-associated, C-terminal / PAC domain profile. / His Kinase A (phospho-acceptor) domain / His Kinase A (phosphoacceptor) domain /  Signal transduction histidine kinase, dimerisation/phosphoacceptor domain / Signal transduction histidine kinase, dimerisation/phosphoacceptor domain /  Signal transduction histidine kinase, dimerisation/phosphoacceptor domain superfamily / Signal transduction histidine kinase-related protein, C-terminal / Signal transduction histidine kinase, dimerisation/phosphoacceptor domain superfamily / Signal transduction histidine kinase-related protein, C-terminal /  Histidine kinase domain / Histidine kinase domain profile. / Histidine kinase domain / Histidine kinase domain profile. /  Response regulator receiver domain / cheY-homologous receiver domain / Signal transduction response regulator, receiver domain / Response regulatory domain profile. / CheY-like superfamily / PAS fold / PAS fold / Response regulator receiver domain / cheY-homologous receiver domain / Signal transduction response regulator, receiver domain / Response regulatory domain profile. / CheY-like superfamily / PAS fold / PAS fold /  PAS domain / PAS repeat profile. / Four Helix Bundle (Hemerythrin (Met), subunit A) / Histidine kinase-, DNA gyrase B-, and HSP90-like ATPase / PAS domain / PAS repeat profile. / Four Helix Bundle (Hemerythrin (Met), subunit A) / Histidine kinase-, DNA gyrase B-, and HSP90-like ATPase /  PAS domain / PAS domain superfamily / Histidine kinase-like ATPases / Histidine kinase/HSP90-like ATPase / Histidine kinase/HSP90-like ATPase superfamily / Up-down Bundle / Mainly Alpha類似検索 - ドメイン・相同性 PAS domain / PAS domain superfamily / Histidine kinase-like ATPases / Histidine kinase/HSP90-like ATPase / Histidine kinase/HSP90-like ATPase superfamily / Up-down Bundle / Mainly Alpha類似検索 - ドメイン・相同性Aerobic respiration control sensor protein ArcB / Aerobic respiration control sensor protein ArcB類似検索 - 構成要素 |
|---|
| 生物種 |    Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
|---|
| 手法 |  X線回折 / X線回折 /  多重同系置換 / 解像度: 2.06 Å 多重同系置換 / 解像度: 2.06 Å |
|---|
 データ登録者 データ登録者 | Kato, M. / Mizuno, T. / Shimizu, T. / Hakoshima, T. |
|---|
 引用 引用 | |
|---|
| 履歴 | | 登録 | 1997年11月27日 | 処理サイト: BNL |
|---|
| 改定 1.0 | 1998年3月18日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年3月24日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2024年2月7日 | Group: Data collection / Database references / Derived calculations
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_struct_conn_angle / struct_conn / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr1_symmetry / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.ptnr3_symmetry / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr1_symmetry / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_conn.ptnr2_symmetry / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード HISTIDINE KINASE / PHOSPHOTRANSFER /
HISTIDINE KINASE / PHOSPHOTRANSFER /  TWO-COMPONENT SYSTEM /
TWO-COMPONENT SYSTEM /  FOUR-HELIX BUNDLE (ヘリックスバンドル)
FOUR-HELIX BUNDLE (ヘリックスバンドル) 機能・相同性情報
機能・相同性情報 histidine kinase / phosphorelay sensor kinase activity / plasma membrane => GO:0005886 /
histidine kinase / phosphorelay sensor kinase activity / plasma membrane => GO:0005886 /  phosphoprotein phosphatase activity / protein autophosphorylation / regulation of DNA-templated transcription /
phosphoprotein phosphatase activity / protein autophosphorylation / regulation of DNA-templated transcription /  シグナル伝達 /
シグナル伝達 /  ATP binding ...peptidyl-histidine phosphorylation / response to oxygen levels /
ATP binding ...peptidyl-histidine phosphorylation / response to oxygen levels /  histidine kinase / phosphorelay sensor kinase activity / plasma membrane => GO:0005886 /
histidine kinase / phosphorelay sensor kinase activity / plasma membrane => GO:0005886 /  phosphoprotein phosphatase activity / protein autophosphorylation / regulation of DNA-templated transcription /
phosphoprotein phosphatase activity / protein autophosphorylation / regulation of DNA-templated transcription /  シグナル伝達 /
シグナル伝達 /  ATP binding /
ATP binding /  細胞膜 /
細胞膜 /  細胞質基質
細胞質基質
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌) X線回折 /
X線回折 /  多重同系置換 / 解像度: 2.06 Å
多重同系置換 / 解像度: 2.06 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Cell(Cambridge,Mass.) / 年: 1997
ジャーナル: Cell(Cambridge,Mass.) / 年: 1997 ジャーナル: Acta Crystallogr.,Sect.D / 年: 1996
ジャーナル: Acta Crystallogr.,Sect.D / 年: 1996 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1a0b.cif.gz
1a0b.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1a0b.ent.gz
pdb1a0b.ent.gz PDB形式
PDB形式 1a0b.json.gz
1a0b.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/a0/1a0b
https://data.pdbj.org/pub/pdb/validation_reports/a0/1a0b ftp://data.pdbj.org/pub/pdb/validation_reports/a0/1a0b
ftp://data.pdbj.org/pub/pdb/validation_reports/a0/1a0b リンク
リンク 集合体
集合体
 要素
要素
 Escherichia coli (大腸菌) / プラスミド: PSU2DH / 細胞内の位置 (発現宿主): CYTOPLASM / 発現宿主:
Escherichia coli (大腸菌) / プラスミド: PSU2DH / 細胞内の位置 (発現宿主): CYTOPLASM / 発現宿主: 
 Escherichia coli (大腸菌) / 株 (発現宿主): K-12 / Variant (発現宿主): DZ225
Escherichia coli (大腸菌) / 株 (発現宿主): K-12 / Variant (発現宿主): DZ225 転移酵素; リンを含む基を移すもの; 含窒素基に移すもの
転移酵素; リンを含む基を移すもの; 含窒素基に移すもの 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 回転陽極 / タイプ: RIGAKU RUH3R / 波長: 1.5418
回転陽極 / タイプ: RIGAKU RUH3R / 波長: 1.5418  : 1.5418 Å / 相対比: 1
: 1.5418 Å / 相対比: 1  解析
解析 :
:  多重同系置換 / 解像度: 2.06→10 Å / Rfactor Rfree error: 0.01 / Data cutoff high absF: 10000000 / Data cutoff low absF: 0.001 / Isotropic thermal model: RESTRAINED / 交差検証法: A POSTERIORI / σ(F): 1
多重同系置換 / 解像度: 2.06→10 Å / Rfactor Rfree error: 0.01 / Data cutoff high absF: 10000000 / Data cutoff low absF: 0.001 / Isotropic thermal model: RESTRAINED / 交差検証法: A POSTERIORI / σ(F): 1  X-PLOR / バージョン: 3.1 / 分類: refinement
X-PLOR / バージョン: 3.1 / 分類: refinement ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj









