+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9146 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of lipid droplet formation protein Seipin/BSCL2 | |||||||||||||||||||||
 マップデータ マップデータ | Cryo-EM structure of lipid droplet formation protein Seipin/BSCL2 | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of axon extension involved in regeneration / diacylglycerol metabolic process / regulation of triglyceride metabolic process / lipid droplet formation / phosphatidic acid metabolic process / : /  regulation of lipid storage / regulation of lipid biosynthetic process / positive regulation of lipid storage / regulation of lipid storage / regulation of lipid biosynthetic process / positive regulation of lipid storage /  lipid storage ...positive regulation of axon extension involved in regeneration / diacylglycerol metabolic process / regulation of triglyceride metabolic process / lipid droplet formation / phosphatidic acid metabolic process / : / lipid storage ...positive regulation of axon extension involved in regeneration / diacylglycerol metabolic process / regulation of triglyceride metabolic process / lipid droplet formation / phosphatidic acid metabolic process / : /  regulation of lipid storage / regulation of lipid biosynthetic process / positive regulation of lipid storage / regulation of lipid storage / regulation of lipid biosynthetic process / positive regulation of lipid storage /  lipid storage / lipid droplet organization / endoplasmic reticulum calcium ion homeostasis / lipid biosynthetic process / fatty acid beta-oxidation / response to starvation / lipid storage / lipid droplet organization / endoplasmic reticulum calcium ion homeostasis / lipid biosynthetic process / fatty acid beta-oxidation / response to starvation /  lipid droplet / endoplasmic reticulum membrane / lipid droplet / endoplasmic reticulum membrane /  小胞体 小胞体類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ) | |||||||||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.0 Å クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Sui X / Arlt H / Liao M / Walther CT / Farese VR | |||||||||||||||||||||
| 資金援助 |  米国, 米国,  ドイツ, 6件 ドイツ, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: J Cell Biol / 年: 2018 ジャーナル: J Cell Biol / 年: 2018タイトル: Cryo-electron microscopy structure of the lipid droplet-formation protein seipin. 著者: Xuewu Sui / Henning Arlt / Kelly P Brock / Zon Weng Lai / Frank DiMaio / Debora S Marks / Maofu Liao / Robert V Farese / Tobias C Walther /  要旨: Metabolic energy is stored in cells primarily as triacylglycerols in lipid droplets (LDs), and LD dysregulation leads to metabolic diseases. The formation of monolayer-bound LDs from the endoplasmic ...Metabolic energy is stored in cells primarily as triacylglycerols in lipid droplets (LDs), and LD dysregulation leads to metabolic diseases. The formation of monolayer-bound LDs from the endoplasmic reticulum (ER) bilayer is poorly understood, but the ER protein seipin is essential to this process. In this study, we report a cryo-electron microscopy structure and functional characterization of seipin. The structure reveals a ring-shaped dodecamer with the luminal domain of each monomer resolved at ∼4.0 Å. Each luminal domain monomer exhibits two distinctive features: a hydrophobic helix (HH) positioned toward the ER bilayer and a β-sandwich domain with structural similarity to lipid-binding proteins. This structure and our functional testing in cells suggest a model in which seipin oligomers initially detect forming LDs in the ER via HHs and subsequently act as membrane anchors to enable lipid transfer and LD growth. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9146.map.gz emd_9146.map.gz | 59 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9146-v30.xml emd-9146-v30.xml emd-9146.xml emd-9146.xml | 13.8 KB 13.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
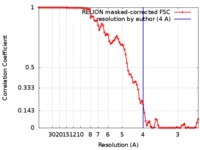
| FSC (解像度算出) |  emd_9146_fsc.xml emd_9146_fsc.xml | 8.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9146.png emd_9146.png | 101.7 KB | ||
| その他 |  emd_9146_additional.map.gz emd_9146_additional.map.gz | 45 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9146 http://ftp.pdbj.org/pub/emdb/structures/EMD-9146 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9146 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9146 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9146.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9146.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of lipid droplet formation protein Seipin/BSCL2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.31 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Cryo-EM structure of lipid droplet formation protein Seipin/BSCL2
| ファイル | emd_9146_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of lipid droplet formation protein Seipin/BSCL2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : cryo-EM protein structure of seipin/BSCL2
| 全体 | 名称: cryo-EM protein structure of seipin/BSCL2 低温電子顕微鏡法 低温電子顕微鏡法 |
|---|---|
| 要素 |
|
-超分子 #1: cryo-EM protein structure of seipin/BSCL2
| 超分子 | 名称: cryo-EM protein structure of seipin/BSCL2 / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ) |
| 分子量 | 理論値: 43 kDa/nm |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換プラスミド: pET28a Escherichia coli (大腸菌) / 組換プラスミド: pET28a |
-分子 #1: Seipin
| 分子 | 名称: Seipin / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ) |
| 分子量 | 理論値: 43.95707 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MGNILLRLIV FALDPLGLGR RFLIRPAVNL GWNVYDRVRS KADEKVGTVR ELVLRLGLIA FAVVLIIWLA VFMYAAFYYV YMPAISHTR PVHMQFKTCL ETSTPCTFPH AHVSLTKKQQ LLMVGQAYKV IVNIDMPESP QNLELGMFMV CAEMRDYDSM L RGHSCRSA ...文字列: MGNILLRLIV FALDPLGLGR RFLIRPAVNL GWNVYDRVRS KADEKVGTVR ELVLRLGLIA FAVVLIIWLA VFMYAAFYYV YMPAISHTR PVHMQFKTCL ETSTPCTFPH AHVSLTKKQQ LLMVGQAYKV IVNIDMPESP QNLELGMFMV CAEMRDYDSM L RGHSCRSA MMRYRSPLIR MISTWVLSPL YVLGWKEEFQ QVPVEIFSRY LEERQHPITD VYVEIQSQKI QFYTVTLHIV AD FTGLRYI MFNWPVLSAI VAISTNLFFI LVVFLLSWYH WSDAKWLHSV QIKYARLTKS LEPGVIHSKA SSLRDDDDDL VAY SDKSDI ADVGGDTLSD VDADDLVLVK KSRSGKRESP DALRKRPTKK TTADHAAALE HHHHHH |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / 装置: GATAN CRYOPLUNGE 3 |
| 詳細 | Protein sample was monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 58.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X