+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9071 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single-Molecule 3D Image of Human Plasma Intermediate-Density Lipoprotein (No. 03) | |||||||||||||||
 マップデータ マップデータ | primary map | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
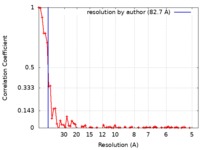
| 手法 |  電子線トモグラフィー法 / 電子線トモグラフィー法 /  クライオ電子顕微鏡法 / 解像度: 82.7 Å クライオ電子顕微鏡法 / 解像度: 82.7 Å | |||||||||||||||
 データ登録者 データ登録者 | Lei D / Yu Y / Kuang Y / Krauss R / Ren G | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Biochim Biophys Acta Mol Cell Biol Lipids / 年: 2019 ジャーナル: Biochim Biophys Acta Mol Cell Biol Lipids / 年: 2019タイトル: Single-molecule 3D imaging of human plasma intermediate-density lipoproteins reveals a polyhedral structure. 著者: Dongsheng Lei / Yadong Yu / Yu-Lin Kuang / Jianfang Liu / Ronald M Krauss / Gang Ren /  要旨: Intermediate-density lipoproteins (IDLs), the remnants of very-low-density lipoproteins via lipolysis, are rich in cholesteryl ester and are associated with cardiovascular disease. Despite ...Intermediate-density lipoproteins (IDLs), the remnants of very-low-density lipoproteins via lipolysis, are rich in cholesteryl ester and are associated with cardiovascular disease. Despite pharmacological interest in IDLs, their three-dimensional (3D) structure is still undetermined due to their variation in size, composition, and dynamic structure. To explore the 3D structure of IDLs, we reconstructed 3D density maps from individual IDL particles using cryo-electron microscopy (cryo-EM) and individual-particle electron tomography (IPET, without averaging from different molecules). 3D reconstructions of IDLs revealed an unexpected polyhedral structure that deviates from the generally assumed spherical shape model (Frias et al., 2007; Olson, 1998; Shen et al., 1977). The polyhedral-shaped IDL contains a high-density shell formed by flat surfaces that are similar to those of very-low-density lipoproteins but have sharper dihedral angles between nearby surfaces. These flat surfaces would be less hydrophobic than the curved surface of mature spherical high-density lipoprotein (HDL), leading to a lower binding affinity of IDL to hydrophobic proteins (such as cholesteryl ester transfer protein) than HDL. This is the first visualization of the IDL 3D structure, which could provide fundamental clues for delineating the role of IDL in lipid metabolism and cardiovascular disease. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9071.map.gz emd_9071.map.gz | 24.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9071-v30.xml emd-9071-v30.xml emd-9071.xml emd-9071.xml | 10.4 KB 10.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9071_fsc.xml emd_9071_fsc.xml | 6.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9071.png emd_9071.png | 23.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9071 http://ftp.pdbj.org/pub/emdb/structures/EMD-9071 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9071 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9071 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9069C  9070C  9072C  9073C  9074C  9075C  9076C  9077C  9078C  9079C  9080C  9081C  9082C  9083C  9084C  9085C  9086C  9087C  9088C  9089C  9090C  9091C 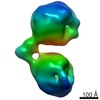 9092C  9093C  9094C 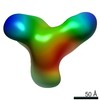 9095C  9096C 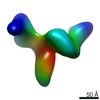 9097C 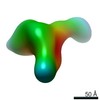 9098C  9099C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9071.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9071.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | primary map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.4 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human plasma intermediate-density lipoprotein
| 全体 | 名称: Human plasma intermediate-density lipoprotein |
|---|---|
| 要素 |
|
-超分子 #1: Human plasma intermediate-density lipoprotein
| 超分子 | 名称: Human plasma intermediate-density lipoprotein / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 組織: Blood Homo sapiens (ヒト) / 組織: Blood |
| 分子量 | 実験値: 3 MDa |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  電子線トモグラフィー法 電子線トモグラフィー法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7 / 詳細: Dulbecco's phosphate buffered saline (DPBS) |
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / 装置: LEICA EM GP |
| 詳細 | Human IDLs were isolated from the plasma of a healthy individual by non-equilibrium density gradient ultracentrifugation |
| 切片作成 | その他: NO SECTIONING |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | ZEISS LIBRA120PLUS |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.2 mm / 倍率(公称値): 50000 Bright-field microscopy / Cs: 2.2 mm / 倍率(公称値): 50000 |
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 平均電子線量: 1.4 e/Å2 |
 ムービー
ムービー コントローラー
コントローラー