+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7449 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the Gasdermin A3 membrane pore | ||||||||||||
 マップデータ マップデータ | Gasdermin A3 membrane pore | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード |  Pyroptosis / Pyroptosis /  Pore forming protein (膜孔形成毒素) / Pore forming protein (膜孔形成毒素) /  IMMUNE SYSTEM (免疫系) IMMUNE SYSTEM (免疫系) | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報sebaceous gland cell differentiation / avascular cornea development in camera-type eye / negative regulation of timing of anagen /  毛包 / wide pore channel activity / 毛包 / wide pore channel activity /  mammary gland development / phosphatidylinositol-4-phosphate binding / positive regulation of extrinsic apoptotic signaling pathway / skin development / hair follicle morphogenesis ...sebaceous gland cell differentiation / avascular cornea development in camera-type eye / negative regulation of timing of anagen / mammary gland development / phosphatidylinositol-4-phosphate binding / positive regulation of extrinsic apoptotic signaling pathway / skin development / hair follicle morphogenesis ...sebaceous gland cell differentiation / avascular cornea development in camera-type eye / negative regulation of timing of anagen /  毛包 / wide pore channel activity / 毛包 / wide pore channel activity /  mammary gland development / phosphatidylinositol-4-phosphate binding / positive regulation of extrinsic apoptotic signaling pathway / skin development / hair follicle morphogenesis / mammary gland development / phosphatidylinositol-4-phosphate binding / positive regulation of extrinsic apoptotic signaling pathway / skin development / hair follicle morphogenesis /  phosphatidylserine binding / phosphatidylserine binding /  pyroptosis / somatic stem cell population maintenance / hair follicle development / extrinsic apoptotic signaling pathway / pyroptosis / somatic stem cell population maintenance / hair follicle development / extrinsic apoptotic signaling pathway /  phosphatidylinositol-4,5-bisphosphate binding / positive regulation of interleukin-1 beta production / phosphatidylinositol-4,5-bisphosphate binding / positive regulation of interleukin-1 beta production /  ミトコンドリア / negative regulation of canonical Wnt signaling pathway / defense response to bacterium / positive regulation of apoptotic process / perinuclear region of cytoplasm / ミトコンドリア / negative regulation of canonical Wnt signaling pathway / defense response to bacterium / positive regulation of apoptotic process / perinuclear region of cytoplasm /  生体膜 / 生体膜 /  細胞核 / 細胞核 /  細胞膜 / 細胞膜 /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) | ||||||||||||
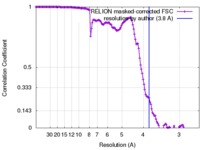
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.8 Å クライオ電子顕微鏡法 / 解像度: 3.8 Å | ||||||||||||
 データ登録者 データ登録者 | Ruan J / Wu H | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2018 ジャーナル: Nature / 年: 2018タイトル: Cryo-EM structure of the gasdermin A3 membrane pore. 著者: Jianbin Ruan / Shiyu Xia / Xing Liu / Judy Lieberman / Hao Wu /  要旨: Gasdermins mediate inflammatory cell death after cleavage by caspases or other, unknown enzymes. The cleaved N-terminal fragments bind to acidic membrane lipids to form pores, but the mechanism of ...Gasdermins mediate inflammatory cell death after cleavage by caspases or other, unknown enzymes. The cleaved N-terminal fragments bind to acidic membrane lipids to form pores, but the mechanism of pore formation remains unresolved. Here we present the cryo-electron microscopy structures of the 27-fold and 28-fold single-ring pores formed by the N-terminal fragment of mouse GSDMA3 (GSDMA3-NT) at 3.8 and 4.2 Å resolutions, and of a double-ring pore at 4.6 Å resolution. In the 27-fold pore, a 108-stranded anti-parallel β-barrel is formed by two β-hairpins from each subunit capped by a globular domain. We identify a positively charged helix that interacts with the acidic lipid cardiolipin. GSDMA3-NT undergoes radical conformational changes upon membrane insertion to form long, membrane-spanning β-strands. We also observe an unexpected additional symmetric ring of GSDMA3-NT subunits that does not insert into the membrane in the double-ring pore, which may represent a pre-pore state of GSDMA3-NT. These structures provide a basis that explains the activities of several mutant gasdermins, including defective mutants that are associated with cancer. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7449.map.gz emd_7449.map.gz | 16 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7449-v30.xml emd-7449-v30.xml emd-7449.xml emd-7449.xml | 14.5 KB 14.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7449_fsc.xml emd_7449_fsc.xml | 13.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7449.png emd_7449.png | 95.7 KB | ||
| Filedesc metadata |  emd-7449.cif.gz emd-7449.cif.gz | 5.2 KB | ||
| その他 |  emd_7449_additional_1.map.gz emd_7449_additional_1.map.gz emd_7449_additional_2.map.gz emd_7449_additional_2.map.gz | 165.4 MB 165.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7449 http://ftp.pdbj.org/pub/emdb/structures/EMD-7449 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7449 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7449 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7449.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7449.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Gasdermin A3 membrane pore | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.32 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Gasdermin A3 membrane pore
| ファイル | emd_7449_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Gasdermin A3 membrane pore | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Gasdermin A3 membrane pore
| ファイル | emd_7449_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Gasdermin A3 membrane pore | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Gasdermin A3 N domain
| 全体 | 名称: Gasdermin A3 N domain |
|---|---|
| 要素 |
|
-超分子 #1: Gasdermin A3 N domain
| 超分子 | 名称: Gasdermin A3 N domain / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 810 KDa |
-分子 #1: Gasdermin-A3
| 分子 | 名称: Gasdermin-A3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 30.401035 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: HMPVFEDVTR ALVRELNPRG DLTPLDSLID FKHFRPFCLV LRKRKSTLFW GARYVRTDYT LLDLLEPGSS PSDLTDSGNF SFKNMLDVQ VQGLVEVPKT VKVKGTAGLS QSSTLEVQTL SVAPSALENL KKERKLSADH SFLNEMRYHE KNLYVVMEAV E AKQEVTVE ...文字列: HMPVFEDVTR ALVRELNPRG DLTPLDSLID FKHFRPFCLV LRKRKSTLFW GARYVRTDYT LLDLLEPGSS PSDLTDSGNF SFKNMLDVQ VQGLVEVPKT VKVKGTAGLS QSSTLEVQTL SVAPSALENL KKERKLSADH SFLNEMRYHE KNLYVVMEAV E AKQEVTVE QTGNANAIFS LPSLALLGLQ GSLNNNKAVT IPKGCVLAYR VRLLRVFLFN LWDIPYICND SMQTFPKIRR VP CSAFISP TQMISEEPEE EKLIGELEVL FQ UniProtKB: Gasdermin-A3 |
-分子 #2: CARDIOLIPIN
| 分子 | 名称: CARDIOLIPIN / タイプ: ligand / ID: 2 / コピー数: 1 / 式: CDL |
|---|---|
| 分子量 | 理論値: 1.464043 KDa |
| Chemical component information |  ChemComp-CDL: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X