+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6247 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Thermoplasma acidophilum 20S proteasome | |||||||||
 マップデータ マップデータ | Thermoplasma acidophilum 20S proteasome. Reconstruction determined from the first 1,200 particles of a dataset used to calculate map EMD-5623. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | T. acidophilum 20S proteasome | |||||||||
| 生物種 |    Thermoplasma acidophilum (好酸性) Thermoplasma acidophilum (好酸性) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 5.0 Å クライオ電子顕微鏡法 / 解像度: 5.0 Å | |||||||||
 データ登録者 データ登録者 | Li X / Cheng Y | |||||||||
 引用 引用 |  ジャーナル: Nat Methods / 年: 2015 ジャーナル: Nat Methods / 年: 2015タイトル: Atomic-accuracy models from 4.5-Å cryo-electron microscopy data with density-guided iterative local refinement. 著者: Frank DiMaio / Yifan Song / Xueming Li / Matthias J Brunner / Chunfu Xu / Vincent Conticello / Edward Egelman / Thomas Marlovits / Yifan Cheng / David Baker /    要旨: We describe a general approach for refining protein structure models on the basis of cryo-electron microscopy maps with near-atomic resolution. The method integrates Monte Carlo sampling with local ...We describe a general approach for refining protein structure models on the basis of cryo-electron microscopy maps with near-atomic resolution. The method integrates Monte Carlo sampling with local density-guided optimization, Rosetta all-atom refinement and real-space B-factor fitting. In tests on experimental maps of three different systems with 4.5-Å resolution or better, the method consistently produced models with atomic-level accuracy largely independently of starting-model quality, and it outperformed the molecular dynamics-based MDFF method. Cross-validated model quality statistics correlated with model accuracy over the three test systems. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6247.map.gz emd_6247.map.gz | 56.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6247-v30.xml emd-6247-v30.xml emd-6247.xml emd-6247.xml | 8.6 KB 8.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  400_6247.gif 400_6247.gif 80_6247.gif 80_6247.gif | 60.7 KB 5.3 KB | ||
| その他 |  Half1_AmpCorr5.0A.map.gz Half1_AmpCorr5.0A.map.gz Half2_AmpCorr5.0A.map.gz Half2_AmpCorr5.0A.map.gz | 56.2 MB 56.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6247 http://ftp.pdbj.org/pub/emdb/structures/EMD-6247 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6247 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6247 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6247.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6247.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Thermoplasma acidophilum 20S proteasome. Reconstruction determined from the first 1,200 particles of a dataset used to calculate map EMD-5623. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.2156 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-添付マップデータ: Half1 AmpCorr5.0A.map
| ファイル | Half1_AmpCorr5.0A.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
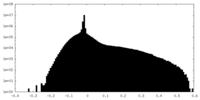
| 密度ヒストグラム |
-添付マップデータ: Half2 AmpCorr5.0A.map
| ファイル | Half2_AmpCorr5.0A.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
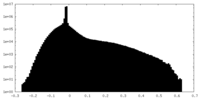
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Thermoplasma acidophilum 20S proteasome
| 全体 | 名称: Thermoplasma acidophilum 20S proteasome |
|---|---|
| 要素 |
|
-超分子 #1000: Thermoplasma acidophilum 20S proteasome
| 超分子 | 名称: Thermoplasma acidophilum 20S proteasome / タイプ: sample / ID: 1000 / 詳細: The sample was monodisperse. / 集合状態: 28-mer / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 700 KDa |
-分子 #1: Thermoplasma acidophilum 20S proteasome
| 分子 | 名称: Thermoplasma acidophilum 20S proteasome / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 集合状態: 28-mer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:    Thermoplasma acidophilum (好酸性) Thermoplasma acidophilum (好酸性) |
| 分子量 | 実験値: 700 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK III |
|---|
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.0 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 31000 Bright-field microscopy / Cs: 2.0 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 31000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 日付 | 2012年1月1日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GATAN K2 (4k x 4k) / 平均電子線量: 20 e/Å2 |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 5.0 Å / 解像度の算出法: OTHER / 使用した粒子像数: 1200 |
|---|---|
| 詳細 | FREALIGN |
 ムービー
ムービー コントローラー
コントローラー
















 Z
Z Y
Y X
X