+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4347 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the herpes-simplex virus portal-vertex | |||||||||
 マップデータ マップデータ | Reconstruction of the herpes simplex virus type 1 virion, with relaxed (C5) symmetry. The map is sharpened. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Herpes simplex virus (type 1 / strain 17) (ヘルペスウイルス) Herpes simplex virus (type 1 / strain 17) (ヘルペスウイルス) | |||||||||
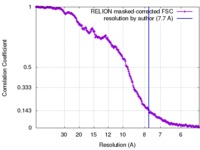
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 7.7 Å クライオ電子顕微鏡法 / 解像度: 7.7 Å | |||||||||
 データ登録者 データ登録者 | McElwee M / Vijayakrishnan S / Rixon FJ / Bhella D | |||||||||
 引用 引用 |  ジャーナル: PLoS Biol / 年: 2018 ジャーナル: PLoS Biol / 年: 2018タイトル: Structure of the herpes simplex virus portal-vertex. 著者: Marion McElwee / Swetha Vijayakrishnan / Frazer Rixon / David Bhella /  要旨: Herpesviruses include many important human pathogens such as herpes simplex virus, cytomegalovirus, varicella-zoster virus, and the oncogenic Epstein-Barr virus and Kaposi sarcoma-associated ...Herpesviruses include many important human pathogens such as herpes simplex virus, cytomegalovirus, varicella-zoster virus, and the oncogenic Epstein-Barr virus and Kaposi sarcoma-associated herpesvirus. Herpes virions contain a large icosahedral capsid that has a portal at a unique 5-fold vertex, similar to that seen in the tailed bacteriophages. The portal is a molecular motor through which the viral genome enters the capsid during virion morphogenesis. The genome also exits the capsid through the portal-vertex when it is injected through the nuclear pore into the nucleus of a new host cell to initiate infection. Structural investigations of the herpesvirus portal-vertex have proven challenging, owing to the small size of the tail-like portal-vertex-associated tegument (PVAT) and the presence of the tegument layer that lays between the nucleocapsid and the viral envelope, obscuring the view of the portal-vertex. Here, we show the structure of the herpes simplex virus portal-vertex at subnanometer resolution, solved by electron cryomicroscopy (cryoEM) and single-particle 3D reconstruction. This led to a number of new discoveries, including the presence of two previously unknown portal-associated structures that occupy the sites normally taken by the penton and the Ta triplex. Our data revealed that the PVAT is composed of 10 copies of the C-terminal domain of pUL25, which are uniquely arranged as two tiers of star-shaped density. Our 3D reconstruction of the portal-vertex also shows that one end of the viral genome extends outside the portal in the manner described for some bacteriophages but not previously seen in any eukaryote viruses. Finally, we show that the viral genome is consistently packed in a highly ordered left-handed spool to form concentric shells of DNA. Our data provide new insights into the structure of a molecular machine critical to the biology of an important class of human pathogens. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4347.map.gz emd_4347.map.gz | 747.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4347-v30.xml emd-4347-v30.xml emd-4347.xml emd-4347.xml | 16 KB 16 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4347_fsc.xml emd_4347_fsc.xml | 21.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4347_1.png emd_4347_1.png emd_4347_2.png emd_4347_2.png | 267 KB 253.4 KB | ||
| その他 |  emd_4347_additional.map.gz emd_4347_additional.map.gz | 734.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4347 http://ftp.pdbj.org/pub/emdb/structures/EMD-4347 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4347 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4347 | HTTPS FTP |
-関連構造データ
| 類似構造データ | |
|---|---|
| 電子顕微鏡画像生データ |  EMPIAR-10189 (タイトル: Structure of the herpes-simplex virus portal-vertex EMPIAR-10189 (タイトル: Structure of the herpes-simplex virus portal-vertexData size: 238.6 Data #1: Motion corrected micrographs of herpes simplex virus type 1 virions [micrographs - single frame]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4347.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4347.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of the herpes simplex virus type 1 virion, with relaxed (C5) symmetry. The map is sharpened. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.67 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Reconstruction of the herpes simplex virus type 1...
| ファイル | emd_4347_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of the herpes simplex virus type 1 virion, with relaxed (C5) symmetry. The map is NOT sharpened. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Herpes simplex virus (type 1 / strain 17)
| 全体 | 名称:   Herpes simplex virus (type 1 / strain 17) (ヘルペスウイルス) Herpes simplex virus (type 1 / strain 17) (ヘルペスウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Herpes simplex virus (type 1 / strain 17)
| 超分子 | 名称: Herpes simplex virus (type 1 / strain 17) / タイプ: virus / ID: 1 / 親要素: 0 / 詳細: Virus was propagated in BHK cells / NCBI-ID: 10299 / 生物種: Herpes simplex virus (type 1 / strain 17) / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| ウイルス殻 | Shell ID: 1 / 名称: Capsid / 直径: 1250.0 Å / T番号(三角分割数): 16 |
-超分子 #2: Portal
| 超分子 | 名称: Portal / タイプ: complex / ID: 2 / 親要素: 1 / 詳細: Dodecameric portal comprised of the pUL6 protein |
|---|---|
| 由来(天然) | 生物種:   Herpes simplex virus (type 1 / strain 17) (ヘルペスウイルス) Herpes simplex virus (type 1 / strain 17) (ヘルペスウイルス) |
-超分子 #3: Portal-vertex associated tegument protein
| 超分子 | 名称: Portal-vertex associated tegument protein / タイプ: complex / ID: 3 / 親要素: 1 詳細: portal associated tail-like density comprised of ten copies of the pUL25 protein |
|---|---|
| 由来(天然) | 生物種:   Herpes simplex virus (type 1 / strain 17) (ヘルペスウイルス) Herpes simplex virus (type 1 / strain 17) (ヘルペスウイルス) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.2 / 詳細: Phosphate buffered saline |
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | Purified enveloped virions |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 81000 Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 81000 |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 撮影したグリッド数: 1 / 実像数: 3702 / 平均露光時間: 12.0 sec. / 平均電子線量: 78.0 e/Å2 詳細: Images were acquired as 40 fractions per micrograph at 1.78 angstroms/pixel sampling. |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X