+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3568 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
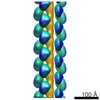
| タイトル | 3D reconstruction for the thin CcdA-CcdB-DNA filaments based on the negative stain electron microscopy dataset. | |||||||||
 マップデータ マップデータ | 3D reconstruction for the thin CcdA-CcdB-DNA filaments based on the negative stain electron microscopy dataset. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   | |||||||||
| 手法 | らせん対称体再構成法 / ネガティブ染色法 / 解像度: 23.0 Å | |||||||||
 データ登録者 データ登録者 | Vandervelde A / Efremov R / Loris R | |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2017 ジャーナル: Nucleic Acids Res / 年: 2017タイトル: Molecular mechanism governing ratio-dependent transcription regulation in the ccdAB operon. 著者: Alexandra Vandervelde / Igor Drobnak / San Hadži / Yann G-J Sterckx / Thomas Welte / Henri De Greve / Daniel Charlier / Rouslan Efremov / Remy Loris / Jurij Lah /    要旨: Bacteria can become transiently tolerant to several classes of antibiotics. This phenomenon known as persistence is regulated by small genetic elements called toxin-antitoxin modules with intricate ...Bacteria can become transiently tolerant to several classes of antibiotics. This phenomenon known as persistence is regulated by small genetic elements called toxin-antitoxin modules with intricate yet often poorly understood self-regulatory features. Here, we describe the structures of molecular complexes and interactions that drive the transcription regulation of the ccdAB toxin-antitoxin module. Low specificity and affinity of the antitoxin CcdA2 for individual binding sites on the operator are enhanced by the toxin CcdB2, which bridges the CcdA2 dimers. This results in a unique extended repressing complex that spirals around the operator and presents equally spaced DNA binding sites. The multivalency of binding sites induces a digital on-off switch for transcription, regulated by the toxin:antitoxin ratio. The ratio at which this switch occurs is modulated by non-specific interactions with the excess chromosomal DNA. Altogether, we present the molecular mechanisms underlying the ratio-dependent transcriptional regulation of the ccdAB operon. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3568.map.gz emd_3568.map.gz | 2.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3568-v30.xml emd-3568-v30.xml emd-3568.xml emd-3568.xml | 16.5 KB 16.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3568_fsc.xml emd_3568_fsc.xml | 2.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3568.png emd_3568.png | 26.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3568 http://ftp.pdbj.org/pub/emdb/structures/EMD-3568 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3568 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3568 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3568_validation.pdf.gz emd_3568_validation.pdf.gz | 232.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3568_full_validation.pdf.gz emd_3568_full_validation.pdf.gz | 231.5 KB | 表示 | |
| XML形式データ |  emd_3568_validation.xml.gz emd_3568_validation.xml.gz | 7.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3568 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3568 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3568 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3568 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3568.map.gz / 形式: CCP4 / 大きさ: 2.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3568.map.gz / 形式: CCP4 / 大きさ: 2.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D reconstruction for the thin CcdA-CcdB-DNA filaments based on the negative stain electron microscopy dataset. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.58 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Thin filaments formed as the complex between DNA, antitoxin CcdA ...
| 全体 | 名称: Thin filaments formed as the complex between DNA, antitoxin CcdA and toxin CcdB. |
|---|---|
| 要素 |
|
-超分子 #1: Thin filaments formed as the complex between DNA, antitoxin CcdA ...
| 超分子 | 名称: Thin filaments formed as the complex between DNA, antitoxin CcdA and toxin CcdB. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: antitoxin CcdA and toxin CcdB
| 超分子 | 名称: antitoxin CcdA and toxin CcdB / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: DNA
| 超分子 | 名称: DNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Antitoxin CcdA
| 分子 | 名称: Antitoxin CcdA / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKQRITVTVD SDSYQLLKAY DVNISGLVST TMQNEARRLR AERWKAENQE GMAEVARFIE MNGSFADENR DW |
-分子 #2: Toxin CcdB
| 分子 | 名称: Toxin CcdB / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MQFKVYTYKR ESRYRLFVDV QSDIIDTPGR RMVIPLASAR LLSDKVSREL YPVVHIGDES WRMMTTDMAS VPVSVIGEEV ADLSHREND IKNAINLMFW GI |
-分子 #3: DNA
| 分子 | 名称: DNA / タイプ: dna / ID: 3 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: AATTGT GAT GCTTCTAAAA TTACTAAAAT TGAGGATTTT AATGCTACAA CAATGCC TG CCTCTTCTTA TTTCTCCGGA GATCCGAAAA CCCCAAGTTA CGGATCTT C CTCTCCCTCC GCACAGCGTT ACATCCCGTC AGCACAGCAT GTAGTGCCT CATACAGTTG ...文字列: AATTGT GAT GCTTCTAAAA TTACTAAAAT TGAGGATTTT AATGCTACAA CAATGCC TG CCTCTTCTTA TTTCTCCGGA GATCCGAAAA CCCCAAGTTA CGGATCTT C CTCTCCCTCC GCACAGCGTT ACATCCCGTC AGCACAGCAT GTAGTGCCT CATACAGTTG CCCATGGCAC TATATGTTGT GTTGTATCTC TGGACTGTGA TGCGCCGCG CAGGGGCGGA AAACAGCGAT ATGATGATTT TCTCAGCGTT G TACACTTC CGGAAAGTCG TTTATTCAAA TAAAGTCGGA TCCATACGAA AC GGGAATG CGGTAATTAC GCTTTGTTTT TATAAGTCAG ATTTTAATTT TTA TTGGTT AACATAACGA AAGGTAAAAT ACATAAGGCT TACTAAAAGC CAGA TAACA GTATGCGTAT TTGCGCGCTG ATTTTTGCGG TATAAGAATA TATAC TGAT ATGTATACCC GAAGTATGTC AAAAAGAGGT GTGCTATGAA GCAGCG TAT TACAGTGACA GTTGACAGCG ACAGCTATCA GTTGCTCAAG GCATATG AT GTCAATATCT CCGGTCTGGT AAGCACAACC ATGCAGAATG AAGCCCGT C GTCTGCGTGC CGAACGCTGG AAAGCGGAAA ATCAGGAAGG GATGGCTGA GGTCGCCCGG TTTATTGAAA TGAACGGCTC TTTTGCTGAC GAGAACAGGG ACTGGTGAA ATGCAGTTTA AGGTTTACAC CTATAAAAGA GAGAGCCGTT A TCGTCTGT TTGTGGATGT ACAGAGTGAT ATTATTGACA CGCCCGGGCG AC GGATGGT GATCCCCCTG GCCAGTGCAC GTCTGCTGTC AGATAAAGTC TCC CGTGAA CTTTACCCGG TGGTGCATAT CGGGGATGAA AGCTGGCGCA TGAT GACCA CCGATATGGC CAGTGTGCCG GTCTCCGTTA TCGGGGAAGA AGTGG CTGA TCTCAGCCAC CGCGAAAATG ACATCAAAAA CGCCATTAAC C |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 構成要素:
詳細: Just before staining, the sample was supplemented with 5 mM MgAc2 (final concentration). | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 染色 | タイプ: NEGATIVE / 材質: Uranyl formate 詳細: The sample was applied to a freshly glow-discharged grid for five minutes after which the grid was washed with 5 mM MgAc2 and adsorbed protein stained with 1% uranyl formate. | ||||||||
| グリッド | モデル: Electron Microscopy Sciences, continuous carbon/formvar 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.045 kPa / 詳細: ELMO glow discharger | ||||||||
| 詳細 | The binding reactions occurred at 20degC. DNA: 15 nM CcdA: 2.5 uM (dimer concentration) CcdB: 2.5 uM (dimer concentration) DNA was first incubated with CcdA for 15 minutes. After adding CcdB, the resulting mixture was incubated for another 15 minutes before preparing the EM sample. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 1400 |
|---|---|
| 撮影 | フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) 撮影したグリッド数: 1 / 実像数: 358 / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 最大 デフォーカス(補正後): 3.0 µm / 最小 デフォーカス(補正後): 1.2 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 3.4 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.8 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダーモデル: JEOL |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera