+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Filament structure of GAC with phosphate | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Glutaminase (グルタミナーゼ) / Glutaminase (グルタミナーゼ) /  phosphate (リン酸塩) / phosphate (リン酸塩) /  filament / PROTEIN FIBRIL / filament / PROTEIN FIBRIL /  HYDROLASE (加水分解酵素) HYDROLASE (加水分解酵素) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報glutamine catabolic process / glutamate biosynthetic process / regulation of respiratory gaseous exchange by nervous system process / Glutamate and glutamine metabolism / intracellular glutamate homeostasis / Glutamate Neurotransmitter Release Cycle /  グルタミナーゼ / グルタミナーゼ /  glutaminase activity / suckling behavior / TP53 Regulates Metabolic Genes ...glutamine catabolic process / glutamate biosynthetic process / regulation of respiratory gaseous exchange by nervous system process / Glutamate and glutamine metabolism / intracellular glutamate homeostasis / Glutamate Neurotransmitter Release Cycle / glutaminase activity / suckling behavior / TP53 Regulates Metabolic Genes ...glutamine catabolic process / glutamate biosynthetic process / regulation of respiratory gaseous exchange by nervous system process / Glutamate and glutamine metabolism / intracellular glutamate homeostasis / Glutamate Neurotransmitter Release Cycle /  グルタミナーゼ / グルタミナーゼ /  glutaminase activity / suckling behavior / TP53 Regulates Metabolic Genes / chemical synaptic transmission / protein homotetramerization / glutaminase activity / suckling behavior / TP53 Regulates Metabolic Genes / chemical synaptic transmission / protein homotetramerization /  ミトコンドリアマトリックス / ミトコンドリアマトリックス /  シナプス / シナプス /  ミトコンドリア / ミトコンドリア /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
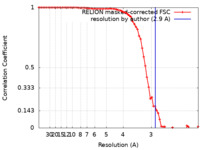
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.9 Å クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Guo CJ / Liu JL | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Res / 年: 2024 ジャーナル: Cell Res / 年: 2024タイトル: Structural basis for activation and filamentation of glutaminase. 著者: Chen-Jun Guo / Zi-Xuan Wang / Ji-Long Liu /   | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35573.map.gz emd_35573.map.gz | 20.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35573-v30.xml emd-35573-v30.xml emd-35573.xml emd-35573.xml | 13.5 KB 13.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_35573_fsc.xml emd_35573_fsc.xml | 7.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_35573.png emd_35573.png | 34.6 KB | ||
| マスクデータ |  emd_35573_msk_1.map emd_35573_msk_1.map | 30.5 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-35573.cif.gz emd-35573.cif.gz | 5.3 KB | ||
| その他 |  emd_35573_half_map_1.map.gz emd_35573_half_map_1.map.gz emd_35573_half_map_2.map.gz emd_35573_half_map_2.map.gz | 22.5 MB 22.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35573 http://ftp.pdbj.org/pub/emdb/structures/EMD-35573 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35573 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35573 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8imaMC  8imbC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35573.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35573.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_35573_msk_1.map emd_35573_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_35573_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_35573_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : GAC filament with phosphate
| 全体 | 名称: GAC filament with phosphate |
|---|---|
| 要素 |
|
-超分子 #1: GAC filament with phosphate
| 超分子 | 名称: GAC filament with phosphate / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Glutaminase kidney isoform, mitochondrial
| 分子 | 名称: Glutaminase kidney isoform, mitochondrial / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号:  グルタミナーゼ グルタミナーゼ |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 53.083738 KDa |
| 組換発現 | 生物種:   Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) |
| 配列 | 文字列: LVASGENKIK QGLLPSLEDL LFYTIAEGQE KIPVHKFITA LKSTGLRTSD PRLKECMDML RLTLQTTSDG VMLDKDLFKK CVQSNIVLL TQAFRRKFVI PDFMSFTSHI DELYESAKKQ SGGKVADYIP QLAKFSPDLW GVSVCTVDGQ RHSTGDTKVP F CLQSCVKP ...文字列: LVASGENKIK QGLLPSLEDL LFYTIAEGQE KIPVHKFITA LKSTGLRTSD PRLKECMDML RLTLQTTSDG VMLDKDLFKK CVQSNIVLL TQAFRRKFVI PDFMSFTSHI DELYESAKKQ SGGKVADYIP QLAKFSPDLW GVSVCTVDGQ RHSTGDTKVP F CLQSCVKP LKYAIAVNDL GTEYVHRYVG KEPSGLRFNK LFLNEDDKPH NPMVNAGAIV VTSLIKQGVN NAEKFDYVMQ FL NKMAGNE YVGFSNATFQ SERESGDRNF AIGYYLKEKK CFPEGTDMVG ILDFYFQLCS IEVTCESASV MAATLANGGF CPI TGERVL SPEAVRNTLS LMHSCGMYDF SGQFAFHVGL PAKSGVAGGI LLVVPNVMGM MCWSPPLDKM GNSVKGIHFC HDLV SLCNF HNYDNLRHFA KKLDPRREGG DQRHSFGPLD YESLQQELAL KETVWKKVSP ESNEDISTTV VYRMESLGEK S UniProtKB: Glutaminase kidney isoform, mitochondrial |
-分子 #2: PHOSPHATE ION
| 分子 | 名称: PHOSPHATE ION / タイプ: ligand / ID: 2 / コピー数: 4 / 式: PO4 |
|---|---|
| 分子量 | 理論値: 94.971 Da |
| Chemical component information |  ChemComp-PO4: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 8.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.2 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X