+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | AtSLAC1 6D mutant in closed state | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Stomatal closure /  anion channel (イオンチャネル) / phosphorylation-dependent activation / anion channel (イオンチャネル) / phosphorylation-dependent activation /  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報response to humidity / stomatal closure / regulation of stomatal opening / inorganic anion transport / voltage-gated monoatomic anion channel activity / regulation of stomatal closure / stomatal movement / response to ozone / intracellular monoatomic ion homeostasis / organic anion transport ...response to humidity / stomatal closure / regulation of stomatal opening / inorganic anion transport / voltage-gated monoatomic anion channel activity / regulation of stomatal closure / stomatal movement / response to ozone / intracellular monoatomic ion homeostasis / organic anion transport / monoatomic anion transmembrane transporter activity / response to carbon dioxide / response to abscisic acid / multicellular organismal-level water homeostasis / monoatomic anion transport / abscisic acid-activated signaling pathway / response to light stimulus /  生物発光 / generation of precursor metabolites and energy / 生物発光 / generation of precursor metabolites and energy /  protein phosphatase binding / protein phosphatase binding /  protein kinase binding / protein kinase binding /  生体膜 / 生体膜 /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Arabidopsis thaliana (シロイヌナズナ) Arabidopsis thaliana (シロイヌナズナ) | |||||||||||||||
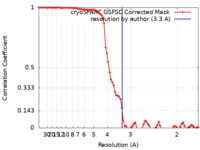
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.3 Å クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||||||||
 データ登録者 データ登録者 | Lee Y / Lee S | |||||||||||||||
| 資金援助 |  韓国, 4件 韓国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Cryo-EM structures of the plant anion channel SLAC1 from Arabidopsis thaliana suggest a combined activation model. 著者: Yeongmok Lee / Hyeon Seong Jeong / Seoyeon Jung / Junmo Hwang / Chi Truc Han Le / Sung-Hoon Jun / Eun Jo Du / KyeongJin Kang / Beom-Gi Kim / Hyun-Ho Lim / Sangho Lee /  要旨: The anion channel SLAC1 functions as a crucial effector in the ABA signaling, leading to stomata closure. SLAC1 is activated by phosphorylation in its intracellular domains. Both a binding-activation ...The anion channel SLAC1 functions as a crucial effector in the ABA signaling, leading to stomata closure. SLAC1 is activated by phosphorylation in its intracellular domains. Both a binding-activation model and an inhibition-release model for activation have been proposed based on only the closed structures of SLAC1, rendering the structure-based activation mechanism controversial. Here we report cryo-EM structures of Arabidopsis SLAC1 WT and its phosphomimetic mutants in open and closed states. Comparison of the open structure with the closed ones reveals the structural basis for opening of the conductance pore. Multiple phosphorylation of an intracellular domain (ICD) causes dissociation of ICD from the transmembrane domain. A conserved, positively-charged sequence motif in the intracellular loop 2 (ICL2) seems to be capable of sensing of the negatively charged phosphorylated ICD. Interactions between ICL2 and ICD drive drastic conformational changes, thereby widening the pore. From our results we propose that SLAC1 operates by a mechanism combining the binding-activation and inhibition-release models. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34303.map.gz emd_34303.map.gz | 2.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34303-v30.xml emd-34303-v30.xml emd-34303.xml emd-34303.xml | 19.2 KB 19.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_34303_fsc.xml emd_34303_fsc.xml | 7.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_34303.png emd_34303.png | 50.5 KB | ||
| マスクデータ |  emd_34303_msk_1.map emd_34303_msk_1.map | 52.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-34303.cif.gz emd-34303.cif.gz | 6.6 KB | ||
| その他 |  emd_34303_half_map_1.map.gz emd_34303_half_map_1.map.gz emd_34303_half_map_2.map.gz emd_34303_half_map_2.map.gz | 48.9 MB 48.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34303 http://ftp.pdbj.org/pub/emdb/structures/EMD-34303 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34303 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34303 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34303.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34303.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_34303_msk_1.map emd_34303_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_34303_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_34303_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Trimer of closed state AtSLAC1 6D mutant with sfGFP tag in GDN an...
| 全体 | 名称: Trimer of closed state AtSLAC1 6D mutant with sfGFP tag in GDN and CHS micelle |
|---|---|
| 要素 |
|
-超分子 #1: Trimer of closed state AtSLAC1 6D mutant with sfGFP tag in GDN an...
| 超分子 | 名称: Trimer of closed state AtSLAC1 6D mutant with sfGFP tag in GDN and CHS micelle タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Arabidopsis thaliana (シロイヌナズナ) Arabidopsis thaliana (シロイヌナズナ) |
| 分子量 | 理論値: 280 KDa |
-分子 #1: Guard cell S-type anion channel SLAC1,Green fluorescent protein (...
| 分子 | 名称: Guard cell S-type anion channel SLAC1,Green fluorescent protein (Fragment) タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Arabidopsis thaliana (シロイヌナズナ) Arabidopsis thaliana (シロイヌナズナ) |
| 分子量 | 理論値: 94.016984 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MERKQSNAHS TFADINEVED EAEQELQQQE NNNNKRFSGN RGPNRGKQRP FRGFSRQVSL EDGFDVLNRE SRERDDKKSL PRSGRSFGG FESGGIINGG DGRKTDFDMF RTKSTLSKQK SLLPDIIRER DIENSLRTED GETKDDDINE NVDAGRYFAA L RGPELDEV ...文字列: MERKQSNAHS TFADINEVED EAEQELQQQE NNNNKRFSGN RGPNRGKQRP FRGFSRQVSL EDGFDVLNRE SRERDDKKSL PRSGRSFGG FESGGIINGG DGRKTDFDMF RTKSTLSKQK SLLPDIIRER DIENSLRTED GETKDDDINE NVDAGRYFAA L RGPELDEV KDNEDILLPK EEQWPFLLRF PIGCFGICLG LSSQAVLWLA LAKSPATNFL HITPLINLVV WLFSLVVLVS VS FTYILKC IFYFEAVKRE YFHPVRVNFF FAPWVVCMFL AISVPPMFSP NRKYLHPAIW CVFMGPYFFL ELKIYGQWLS GGK RRLCKV ANPSSHLSVV GNFVGAILAS KVGWDEVAKF LWAVGFAHYL VVFVTLYQRL PTSEALPKEL HPVYSMFIAA PSAA SIAWN TIYGQFDGCS RTCFFIALFL YISLVARINF FTGFKFSVAW WSYTFPMTTA SVATIKYAEA VPGYPSRALA LTLSF ISTA MVCVLFVSTL LHAFVWQTLF PNDLAIAITK RKLTREKKPF KRAYDLKRWT KQALAKKISA EKDFEAEEES HHGSEN LYF QSMSKGEELF TGVVPILVEL DGDVNGHKFS VRGEGEGDAT NGKLTLKFIC TTGKLPVPWP TLVTTLTYGV QCFSRYP DH MKRHDFFKSA MPEGYVQERT ISFKDDGTYK TRAEVKFEGD TLVNRIELKG IDFKEDGNIL GHKLEYNFNS HNVYITAD K QKNGIKANFK IRHNVEDGSV QLADHYQQNT PIGDGPVLLP DNHYLSTQSV LSKDPNEKRD HMVLLEFVTA AGITHGMDE LYKYPYDVPD YAGGGSHHHH HHHHHH UniProtKB: Guard cell S-type anion channel SLAC1,  緑色蛍光タンパク質 緑色蛍光タンパク質 |
-分子 #2: CHLORIDE ION
| 分子 | 名称: CHLORIDE ION / タイプ: ligand / ID: 2 / コピー数: 3 / 式: CL |
|---|---|
| 分子量 | 理論値: 35.453 Da |
-分子 #3: CHOLESTEROL HEMISUCCINATE
| 分子 | 名称: CHOLESTEROL HEMISUCCINATE / タイプ: ligand / ID: 3 / コピー数: 3 / 式: Y01 |
|---|---|
| 分子量 | 理論値: 486.726 Da |
| Chemical component information |  ChemComp-Y01: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.3 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm最大 デフォーカス(公称値): 1.9000000000000001 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 14 eV |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X