[日本語] English
 万見
万見- EMDB-33943: Cryo-EM structure of MERS-CoV spike protein, Two RBD-up conformation 1 -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of MERS-CoV spike protein, Two RBD-up conformation 1 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  MERS-CoV (MERSコロナウイルス) / Spike / MERS-CoV (MERSコロナウイルス) / Spike /  Glycoprotein (糖タンパク質) / Glycoprotein (糖タンパク質) /  Viral protein (ウイルスタンパク質) Viral protein (ウイルスタンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell endoplasmic reticulum-Golgi intermediate compartment membrane / receptor-mediated endocytosis of virus by host cell /  membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / symbiont entry into host cell / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / symbiont entry into host cell / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / host cell plasma membrane ...host cell endoplasmic reticulum-Golgi intermediate compartment membrane / receptor-mediated endocytosis of virus by host cell / エンベロープ (ウイルス) / host cell plasma membrane ...host cell endoplasmic reticulum-Golgi intermediate compartment membrane / receptor-mediated endocytosis of virus by host cell /  membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / symbiont entry into host cell / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / symbiont entry into host cell / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / host cell plasma membrane / virion membrane / エンベロープ (ウイルス) / host cell plasma membrane / virion membrane /  生体膜 生体膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Human betacoronavirus 2c EMC/2012 (MERSコロナウイルス) Human betacoronavirus 2c EMC/2012 (MERSコロナウイルス) | |||||||||
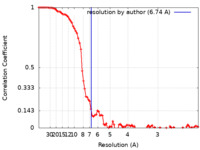
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 6.74 Å クライオ電子顕微鏡法 / 解像度: 6.74 Å | |||||||||
 データ登録者 データ登録者 | Hsu STD / Chang NE / Weng ZW / Yang TJ / Draczkowski P | |||||||||
| 資金援助 |  台湾, 1件 台湾, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: GlycoSHIELD: a versatile pipeline to assess glycan impact on protein structures 著者: Gecht M / von Bulow S / Penet C / Hummer G / Hanus C / Sikora M | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33943.map.gz emd_33943.map.gz | 30.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33943-v30.xml emd-33943-v30.xml emd-33943.xml emd-33943.xml | 18.5 KB 18.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_33943_fsc.xml emd_33943_fsc.xml | 8.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_33943.png emd_33943.png | 24 KB | ||
| その他 |  emd_33943_additional_1.map.gz emd_33943_additional_1.map.gz emd_33943_half_map_1.map.gz emd_33943_half_map_1.map.gz emd_33943_half_map_2.map.gz emd_33943_half_map_2.map.gz | 59.7 MB 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33943 http://ftp.pdbj.org/pub/emdb/structures/EMD-33943 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33943 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33943 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7ymvMC  7ymtC  7ymwC  7ymxC  7ymyC  7ymzC  7yn0C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33943.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33943.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||
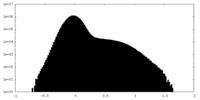
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_33943_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
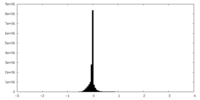
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_33943_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
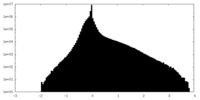
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33943_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
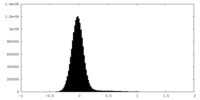
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : recombinant MERS-CoV (betacoronavirus 2c EMC 2012) fm2P Spike
| 全体 | 名称: recombinant MERS-CoV (betacoronavirus 2c EMC 2012) fm2P Spike |
|---|---|
| 要素 |
|
-超分子 #1: recombinant MERS-CoV (betacoronavirus 2c EMC 2012) fm2P Spike
| 超分子 | 名称: recombinant MERS-CoV (betacoronavirus 2c EMC 2012) fm2P Spike タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Human betacoronavirus 2c EMC/2012 (MERSコロナウイルス) Human betacoronavirus 2c EMC/2012 (MERSコロナウイルス) |
-分子 #1: Spike glycoprotein
| 分子 | 名称: Spike glycoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human betacoronavirus 2c EMC/2012 (MERSコロナウイルス) Human betacoronavirus 2c EMC/2012 (MERSコロナウイルス) |
| 分子量 | 理論値: 150.654438 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDSWFILVLL GSGLICVSAS YVDVGPDSVK SACIEVDIQQ TFFDKTWPRP IDVSKADGII YPQGRTYSNI TITYQGLFPY QGDHGDMYV YSAGHATGTT PQKLFVANYS QDVKQFANGF VVRIGAAANS TGTVIISPST SATIRKIYPA FMLGSSVGNF S DGKMGRFF ...文字列: MDSWFILVLL GSGLICVSAS YVDVGPDSVK SACIEVDIQQ TFFDKTWPRP IDVSKADGII YPQGRTYSNI TITYQGLFPY QGDHGDMYV YSAGHATGTT PQKLFVANYS QDVKQFANGF VVRIGAAANS TGTVIISPST SATIRKIYPA FMLGSSVGNF S DGKMGRFF NHTLVLLPDG CGTLLRAFYC ILEPRSGNHC PAGNSYTSFA TYHTPATDCS DGNYNRNASL NSFKEYFNLR NC TFMYTYN ITEDEILEWF GITQTAQGVH LFSSRYVDLY GGNMFQFATL PVYDTIKYYS IIPHSIRSIQ SDRKAWAAFY VYK LQPLTF LLDFSVDGYI RRAIDCGFND LSQLHCSYES FDVESGVYSV SSFEAKPSGS VVEQAEGVEC DFSPLLSGTP PQVY NFKRL VFTNCNYNLT KLLSLFSVND FTCSQISPAA IASNCYSSLI LDYFSYPLSM KSDLSVSSAG PISQFNYKQS FSNPT CLIL ATVPHNLTTI TKPLKYSYIN KCSRLLSDDR TEVPQLVNAN QYSPCVSIVP STVWEDGDYY RKQLSPLEGG GWLVAS GST VAMTEQLQMG FGITVQYGTD TNSVCPKLEF ANDTKIASQL GNCVEYSLYG VSGRGVFQNC TAVGVRQQRF VYDAYQN LV GYYSDDGNYY CLRACVSVPV SVIYDKETKT HATLFGSVAC EHISSTMSQY SRSTRSMLKR RDSTYGPLQT PVGCVLGL V NSSLFVEDCK LPLGQSLCAL PDTPSTLTPA SVGSVPGEMR LASIAFNHPI QVDQLNSSYF KLSIPTNFSF GVTQEYIQT TIQKVTVDCK QYVCNGFQKC EQLLREYGQF CSKINQALHG ANLRQDDSVR NLFASVKSSQ SSPIIPGFGG DFNLTLLEPV SISTGSRSA RSAIEDLLFD KVTIADPGYM QGYDDCMQQG PASARDLICA QYVAGYKVLP PLMDVNMEAA YTSSLLGSIA G VGWTAGLS SFAAIPFAQS IFYRLNGVGI TQQVLSENQK LIANKFNQAL GAMQTGFTTT NEAFQKVQDA VNNNAQALSK LA SELSNTF GAISASIGDI IQRLDPPEQD AQIDRLINGR LTTLNAFVAQ QLVRSESAAL SAQLAKDKVN ECVKAQSKRS GFC GQGTHI VSFVVNAPNG LYFMHVGYYP SNHIEVVSAY GLCDAANPTN CIAPVNGYFI KTNNTRIVDE WSYTGSSFYA PEPI TSLNT KYVAPQVTYQ NISTNLPPPL LGNSTGIDFQ DELDEFFKNV STSIPNFGSL TQINTTLLDL TYEMLSLQQV VKALN ESYI DLKELGNYTY EFGSGGYIPE APRDGQAYVR KDGEWVLLST FLKGQDNSAD IQHSGRPLES RGPFEQKLIS EEDLNM HTG HHHHHH UniProtKB:  スパイクタンパク質 スパイクタンパク質 |
-分子 #8: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 8 / コピー数: 14 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.6 構成要素:
| ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: blot for 2.5 seconds before plunging; blot force: -1; waiting time: 30s.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.7 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 92000 Bright-field microscopy / 最大 デフォーカス(公称値): 1.7 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 92000 |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 撮影したグリッド数: 1 / 実像数: 2886 / 平均電子線量: 40.6 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-7ymv: |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X