+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3074 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Novel inter-subunit contacts in Barley Stripe Mosaic Virus revealed by cryo-EM | |||||||||
 マップデータ マップデータ | Reconstruction of the narrow Capsid of BSMV | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | BSMV /  virus (ウイルス) / helical / virus (ウイルス) / helical /  cryo-EM (低温電子顕微鏡法) / cryo-EM (低温電子顕微鏡法) /  image processing (デジタル画像処理) / MSA image processing (デジタル画像処理) / MSA | |||||||||
| 機能・相同性 | Tobacco mosaic virus-like, coat protein / Tobacco mosaic virus-like, coat protein superfamily / Virus coat protein (TMV like) / helical viral capsid / structural molecule activity / identical protein binding /  カプシド カプシド 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |    Tobacco mosaic virus (タバコモザイクウイルス) / Tobacco mosaic virus (タバコモザイクウイルス) /   Barley stripe mosaic virus (ウイルス) Barley stripe mosaic virus (ウイルス) | |||||||||
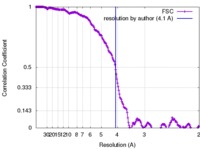
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.1 Å クライオ電子顕微鏡法 / 解像度: 4.1 Å | |||||||||
 データ登録者 データ登録者 | Clare DK / Pechnikova E / Skurat E / Makarov V / Sokolova OS / Solovyev AG / Orlova EV | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2015 ジャーナル: Structure / 年: 2015タイトル: Novel Inter-Subunit Contacts in Barley Stripe Mosaic Virus Revealed by Cryo-Electron Microscopy. 著者: Daniel Kofi Clare / Eugenia V Pechnikova / Eugene V Skurat / Valentin V Makarov / Olga S Sokolova / Andrey G Solovyev / Elena V Orlova /   要旨: Barley stripe mosaic virus (BSMV, genus Hordeivirus) is a rod-shaped single-stranded RNA virus similar to viruses of the structurally characterized and well-studied genus Tobamovirus. Here we report ...Barley stripe mosaic virus (BSMV, genus Hordeivirus) is a rod-shaped single-stranded RNA virus similar to viruses of the structurally characterized and well-studied genus Tobamovirus. Here we report the first high-resolution structure of BSMV at 4.1 Å obtained by cryo-electron microscopy. We discovered that BSMV forms two types of virion that differ in the number of coat protein (CP) subunits per turn and interactions between the CP subunits. While BSMV and tobacco mosaic virus CP subunits have a similar fold and interact with RNA using conserved residues, the axial contacts between the CP of these two viral groups are considerably different. BSMV CP subunits lack substantial axial contacts and are held together by a previously unobserved lateral contact formed at the virion surface via an interacting loop, which protrudes from the CP hydrophobic core to the adjacent CP subunit. These data provide an insight into diversity in structural organization of helical viruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3074.map.gz emd_3074.map.gz | 97.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3074-v30.xml emd-3074-v30.xml emd-3074.xml emd-3074.xml | 12.6 KB 12.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3074_fsc.xml emd_3074_fsc.xml | 20 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3074.jpg emd_3074.jpg | 99.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3074 http://ftp.pdbj.org/pub/emdb/structures/EMD-3074 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3074 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3074 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3074.map.gz / 形式: CCP4 / 大きさ: 100.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3074.map.gz / 形式: CCP4 / 大きさ: 100.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of the narrow Capsid of BSMV | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : chimeric Barley Stripe Mosaic Virus narrow virion
| 全体 | 名称: chimeric Barley Stripe Mosaic Virus narrow virion |
|---|---|
| 要素 |
|
-超分子 #1000: chimeric Barley Stripe Mosaic Virus narrow virion
| 超分子 | 名称: chimeric Barley Stripe Mosaic Virus narrow virion / タイプ: sample / ID: 1000 詳細: The sample is a helical virus with 106 copies of the capsid protein per helical repeat 集合状態: Helical, with 106 copies of the capsid protein per helical repeat Number unique components: 2 |
|---|---|
| 分子量 | 実験値: 22.5 KDa / 理論値: 22.5 KDa |
-超分子 #1: Barley stripe mosaic virus
| 超分子 | 名称: Barley stripe mosaic virus / タイプ: virus / ID: 1 / Name.synonym: BSMV / 詳細: Has RNA bound / NCBI-ID: 12327 / 生物種: Barley stripe mosaic virus / Sci species strain: ND18 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No / Syn species name: BSMV |
|---|---|
| 宿主 | 生物種:   Hordeum vulgare (オオムギ) / 別称: PLANTAE(HIGHER PLANTS) Hordeum vulgare (オオムギ) / 別称: PLANTAE(HIGHER PLANTS) |
| Host system | 生物種:   Nicotiana benthamiana (ベンサミアナタバコ) Nicotiana benthamiana (ベンサミアナタバコ) |
| 分子量 | 実験値: 22.5 KDa / 理論値: 22.5 KDa |
| ウイルス殻 | Shell ID: 1 / 名称: narrow virion / 直径: 216 Å |
-分子 #1: RNA
| 分子 | 名称: RNA / タイプ: rna / ID: 1 / Name.synonym: RNA / 分類: OTHER / Structure: SINGLE STRANDED / Synthetic?: No |
|---|---|
| 由来(天然) | 生物種:    Tobacco mosaic virus (タバコモザイクウイルス) Tobacco mosaic virus (タバコモザイクウイルス) |
| 配列 | 文字列: GAA |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 50mM Tris-HCL, 50mM KCl, 10mM MgCl2 |
| グリッド | 詳細: c-flats r2/2 or home made continuous carbon grids |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 40 % / チャンバー内温度: 100 K / 装置: HOMEMADE PLUNGER 手法: 3.5ul of sample was added to continuous carbon coated or c-flat grids, blotted for 2 seconds and then plunged in liquid ethane |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 150000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.3 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 0.7 µm / 倍率(公称値): 150000 Bright-field microscopy / Cs: 2.3 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 0.7 µm / 倍率(公称値): 150000 |
| 試料ステージ | 試料ホルダーモデル: GATAN HELIUM |
| 温度 | 最低: 80 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 150,000 times magnification |
| 日付 | 2012年6月1日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) デジタル化 - サンプリング間隔: 15 µm / 実像数: 297 / 平均電子線量: 25 e/Å2 / 詳細: each image was a 1 second low dose exposure / ビット/ピクセル: 16 |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー