+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Nodavirus RNA replication protein A polymerase domain, local refinement | |||||||||
 マップデータ マップデータ | Nodavirus RNA replication protein A polymerase domain, local refinement. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Nodavirus methyltransferase domain / Nodavirus Vmethyltransferase / host cell mitochondrial outer membrane /  RNA依存性RNAポリメラーゼ / RNA依存性RNAポリメラーゼ /  RNA-dependent RNA polymerase activity / DNA/RNA polymerase superfamily / RNA-dependent RNA polymerase activity / DNA/RNA polymerase superfamily /  生体膜 / 生体膜 /  RNA依存性RNAポリメラーゼ RNA依存性RNAポリメラーゼ 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Flock House virus (ウイルス) Flock House virus (ウイルス) | |||||||||
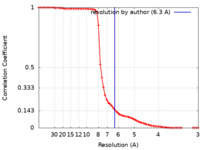
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 6.3 Å クライオ電子顕微鏡法 / 解像度: 6.3 Å | |||||||||
 データ登録者 データ登録者 | Zhan H / Unchwaniwala N / Rebolledo Viveros A / Pennington J / Horswill M / Broadberry R / Myers J / den Boon J / Grant T / Ahlquist P | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: Nodavirus RNA replication crown architecture reveals proto-crown precursor and viral protein A conformational switching. 著者: Hong Zhan / Nuruddin Unchwaniwala / Andrea Rebolledo-Viveros / Janice Pennington / Mark Horswill / Roma Broadberry / Jonathan Myers / Johan A den Boon / Timothy Grant / Paul Ahlquist /  要旨: Positive-strand RNA viruses replicate their genomes in virus-induced membrane vesicles, and the resulting RNA replication complexes are a major target for virus control. Nodavirus studies first ...Positive-strand RNA viruses replicate their genomes in virus-induced membrane vesicles, and the resulting RNA replication complexes are a major target for virus control. Nodavirus studies first revealed viral RNA replication proteins forming a 12-fold symmetric "crown" at the vesicle opening to the cytosol, an arrangement recently confirmed to extend to distantly related alphaviruses. Using cryoelectron microscopy (cryo-EM), we show that mature nodavirus crowns comprise two stacked 12-mer rings of multidomain viral RNA replication protein A. Each ring contains an ~19 nm circle of C-proximal polymerase domains, differentiated by strikingly diverged positions of N-proximal RNA capping/membrane binding domains. The lower ring is a "proto-crown" precursor that assembles prior to RNA template recruitment, RNA synthesis, and replication vesicle formation. In this proto-crown, the N-proximal segments interact to form a toroidal central floor, whose 3.1 Å resolution structure reveals many mechanistic details of the RNA capping/membrane binding domains. In the upper ring, cryo-EM fitting indicates that the N-proximal domains extend radially outside the polymerases, forming separated, membrane-binding "legs." The polymerase and N-proximal domains are connected by a long linker accommodating the conformational switch between the two rings and possibly also polymerase movements associated with RNA synthesis and nonsymmetric electron density in the lower center of mature crowns. The results reveal remarkable viral protein multifunctionality, conformational flexibility, and evolutionary plasticity and insights into (+)RNA virus replication and control. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29291.map.gz emd_29291.map.gz | 25.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29291-v30.xml emd-29291-v30.xml emd-29291.xml emd-29291.xml | 18.1 KB 18.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29291_fsc.xml emd_29291_fsc.xml | 6.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29291.png emd_29291.png | 71 KB | ||
| その他 |  emd_29291_half_map_1.map.gz emd_29291_half_map_1.map.gz emd_29291_half_map_2.map.gz emd_29291_half_map_2.map.gz | 25 MB 25 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29291 http://ftp.pdbj.org/pub/emdb/structures/EMD-29291 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29291 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29291 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8fmbMC  8fm9C  8fmaC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29291.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29291.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Nodavirus RNA replication protein A polymerase domain, local refinement. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.5 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Nodavirus RNA replication protein A polymerase domain, local...
| ファイル | emd_29291_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
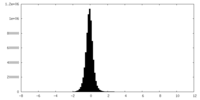
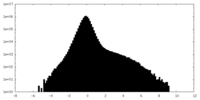
| 注釈 | Nodavirus RNA replication protein A polymerase domain, local refinement, half map1. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Nodavirus RNA replication protein A polymerase domain, local...
| ファイル | emd_29291_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
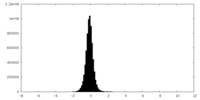
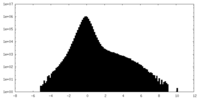
| 注釈 | Nodavirus RNA replication protein A polymerase domain, local refinement, half map2. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Nodavirus RNA replication protein A polymerase domain, local refi...
| 全体 | 名称: Nodavirus RNA replication protein A polymerase domain, local refinement |
|---|---|
| 要素 |
|
-超分子 #1: Nodavirus RNA replication protein A polymerase domain, local refi...
| 超分子 | 名称: Nodavirus RNA replication protein A polymerase domain, local refinement タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Flock House virus (ウイルス) Flock House virus (ウイルス) |
-分子 #1: RNA-directed RNA polymerase
| 分子 | 名称: RNA-directed RNA polymerase / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号:  RNA依存性RNAポリメラーゼ RNA依存性RNAポリメラーゼ |
|---|---|
| 由来(天然) | 生物種:   Flock House virus (ウイルス) Flock House virus (ウイルス) |
| 分子量 | 理論値: 113.996586 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MTLKVILGEH QITRTELLVG IATVSGCGAV VYCISKFWGY GAIAPYPQSG GNRVTRALQR AVIDKTKTPI ETRFYPLDSL RTVTPKRVA DNGHAVSGAV RDAARRLIDE SITAVGGSKF EVNPNPNSST GLRNHFHFAV GDLAQDFRND TPADDAFIVG V DVDYYVTE ...文字列: MTLKVILGEH QITRTELLVG IATVSGCGAV VYCISKFWGY GAIAPYPQSG GNRVTRALQR AVIDKTKTPI ETRFYPLDSL RTVTPKRVA DNGHAVSGAV RDAARRLIDE SITAVGGSKF EVNPNPNSST GLRNHFHFAV GDLAQDFRND TPADDAFIVG V DVDYYVTE PDVLLEHMRP VVLHTFNPKK VSGFDADSPF TIKNNLVEYK VSGGAAWVHP VWDWCEAGEF IASRVRTSWK EW FLQLPLR MIGLEKVGYH KIHHCRPWTD CPDRALVYTI PQYVIWRFNW IDTELHVRKL KRIEYQDETK PGWNRLEYVT DKN ELLVSI GREGEHAQIT IEKEKLDMLS GLSATQSVNA RLIGMGHKDP QYTSMIVQYY TGKKVVSPIS PTVYKPTMPR VHWP VTSDA DVPEVSARQY TLPIVSDCMM MPMIKRWETM SESIERRVTF VANDKKPSDR IAKIAETFVK LMNGPFKDLD PLSIE ETIE RLNKPSQQLQ LRAVFEMIGV KPRQLIESFN KNEPGMKSSR IISGFPDILF ILKVSRYTLA YSDIVLHAEH NEHWYY PGR NPTEIADGVC EFVSDCDAEV IETDFSNLDG RVSSWMQRNI AQKAMVQAFR PEYRDEIISF MDTIINCPAK AKRFGFR YE PGVGVKSGSP TTTPHNTQYN GCVEFTALTF EHPDAEPEDL FRLIGPKCGD DGLSRAIIQK SINRAAKCFG LELKVERY N PEIGLCFLSR VFVDPLATTT TIQDPLRTLR KLHLTTRDPT IPLADAACDR VEGYLCTDAL TPLISDYCKM VLRLYGPTA STEQVRNQRR SRNKEKPYWL TCDGSWPQHP QDAHLMKQVL IKRTAIDEDQ VDALIGRFAA MKDVWEKITH DSEESAAACT FDEDGVAPN SVDESLPLLN DAKQTRANPG TSRPHSNGGG SSHGNELPRR TEQRAQGPRQ PARLPKQGKT NGKSDGNITA G ETQRGGIP RGKGPRGGKT NTRRTPPKAG AQPQPSNNRK SRLEEELRRR LTE |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 2.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 平均電子線量: 100.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-8fmb: |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X