+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human PRPS1-E307A engineered mutation with ADP; Symmetry-expanded Hexamer 2 | |||||||||
 マップデータ マップデータ | Human PRPS1-E307A engineered mutation with ADP, C-terminus, Hexamer Centered | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  phosphoribosyl pyrophosphate synthetase (リボースリン酸ジホスホキナーゼ) / ADP inhibitor / Filament-breaking mutation / phosphoribosyl pyrophosphate synthetase (リボースリン酸ジホスホキナーゼ) / ADP inhibitor / Filament-breaking mutation /  TRANSFERASE (転移酵素) TRANSFERASE (転移酵素) | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.6 Å クライオ電子顕微鏡法 / 解像度: 2.6 Å | |||||||||
 データ登録者 データ登録者 | Hvorecny KL / Kollman JM | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Human PRPS1 filaments stabilize allosteric sites to regulate activity. 著者: Kelli L Hvorecny / Kenzee Hargett / Joel D Quispe / Justin M Kollman /  要旨: The universally conserved enzyme phosphoribosyl pyrophosphate synthetase (PRPS) assembles filaments in evolutionarily diverse organisms. PRPS is a key regulator of nucleotide metabolism, and ...The universally conserved enzyme phosphoribosyl pyrophosphate synthetase (PRPS) assembles filaments in evolutionarily diverse organisms. PRPS is a key regulator of nucleotide metabolism, and mutations in the human enzyme PRPS1 lead to a spectrum of diseases. Here we determine structures of human PRPS1 filaments in active and inhibited states, with fixed assembly contacts accommodating both conformations. The conserved assembly interface stabilizes the binding site for the essential activator phosphate, increasing activity in the filament. Some disease mutations alter assembly, supporting the link between filament stability and activity. Structures of active PRPS1 filaments turning over substrate also reveal coupling of catalysis in one active site with product release in an adjacent site. PRPS1 filaments therefore provide an additional layer of allosteric control, conserved throughout evolution, with likely impact on metabolic homeostasis. Stabilization of allosteric binding sites by polymerization adds to the growing diversity of assembly-based enzyme regulatory mechanisms. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27295.map.gz emd_27295.map.gz | 14.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27295-v30.xml emd-27295-v30.xml emd-27295.xml emd-27295.xml | 15.1 KB 15.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_27295.png emd_27295.png | 119.8 KB | ||
| Filedesc metadata |  emd-27295.cif.gz emd-27295.cif.gz | 5 KB | ||
| その他 |  emd_27295_half_map_1.map.gz emd_27295_half_map_1.map.gz emd_27295_half_map_2.map.gz emd_27295_half_map_2.map.gz | 98.4 MB 98.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27295 http://ftp.pdbj.org/pub/emdb/structures/EMD-27295 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27295 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27295 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27295.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27295.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human PRPS1-E307A engineered mutation with ADP, C-terminus, Hexamer Centered | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||
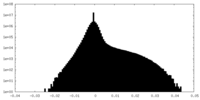
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Human PRPS1-E307A engineered mutation with ADP, C-terminus, Hexamer...
| ファイル | emd_27295_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human PRPS1-E307A engineered mutation with ADP, C-terminus, Hexamer Centered half 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
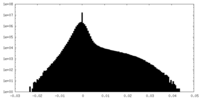
| 密度ヒストグラム |
-ハーフマップ: Human PRPS1-E307A engineered mutation with ADP, C-terminus, Hexamer...
| ファイル | emd_27295_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human PRPS1-E307A engineered mutation with ADP, C-terminus, Hexamer Centered half 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
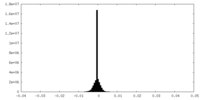
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Hexamer of phosphoribosyl pyrophosphate synthetase 1 E307A engine...
| 全体 | 名称: Hexamer of phosphoribosyl pyrophosphate synthetase 1 E307A engineered mutation with ADP |
|---|---|
| 要素 |
|
-超分子 #1: Hexamer of phosphoribosyl pyrophosphate synthetase 1 E307A engine...
| 超分子 | 名称: Hexamer of phosphoribosyl pyrophosphate synthetase 1 E307A engineered mutation with ADP タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Six copies of PRPS1-E307A assemble to form a hexamer. |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Human Phosphoribosyl Pyrophosphate Synthetase 1 E307A Mutation
| 分子 | 名称: Human Phosphoribosyl Pyrophosphate Synthetase 1 E307A Mutation タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MPNIKIFSGS SHQDLSQKIA DRLGLELGKV VTKKFSNQET CVEIGESVRG EDVYIVQSGC GEINDNLME LLIMINACKI ASASRVTAVI PCFPYARQDK KDKSRAPISA KLVANMLSVA G ADHIITMD LHASQIQGFF DIPVDNLYAE PAVLKWIREN ISEWRNCTIV ...文字列: MPNIKIFSGS SHQDLSQKIA DRLGLELGKV VTKKFSNQET CVEIGESVRG EDVYIVQSGC GEINDNLME LLIMINACKI ASASRVTAVI PCFPYARQDK KDKSRAPISA KLVANMLSVA G ADHIITMD LHASQIQGFF DIPVDNLYAE PAVLKWIREN ISEWRNCTIV SPDAGGAKRV TS IADRLNV DFALIHKERK KANEVDRMVL VGDVKDRVAI LVDDMADTCG TICHAADKLL SAG ATRVYA ILTHGIFSGP AISRINNACF EAVVVTNTIP QEDKMKHCSK IQVIDISMIL AEAI RRTHN GASVSYLFSH VPL |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 |
|---|---|
| グリッド | モデル: C-flat / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.75 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.75 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 90.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3.1) |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 2.6 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: PHENIX (ver. 1.18) / 使用した粒子像数: 851443 |
-原子モデル構築 1
| 精密化 | プロトコル: FLEXIBLE FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー

































 Z
Z Y
Y X
X