+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 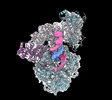 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human DNA polymerase-alpha/primase elongation complex II bound to primer/template | |||||||||
 マップデータ マップデータ | Unsharpened and flipped map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  DNA replication (DNA複製) / human DNA polymerase alpha/primase / DNA replication (DNA複製) / human DNA polymerase alpha/primase /  human primosome / elongation complex / human primosome / elongation complex /  REPLICATION (DNA複製) REPLICATION (DNA複製) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DNA primase AEP / positive regulation of DNA primase activity /  ribonucleotide binding / DNA replication initiation / DNA/RNA hybrid binding / Telomere C-strand synthesis initiation / Inhibition of replication initiation of damaged DNA by RB1/E2F1 / Polymerase switching / alpha DNA polymerase:primase complex / regulation of type I interferon production ...DNA primase AEP / positive regulation of DNA primase activity / ribonucleotide binding / DNA replication initiation / DNA/RNA hybrid binding / Telomere C-strand synthesis initiation / Inhibition of replication initiation of damaged DNA by RB1/E2F1 / Polymerase switching / alpha DNA polymerase:primase complex / regulation of type I interferon production ...DNA primase AEP / positive regulation of DNA primase activity /  ribonucleotide binding / DNA replication initiation / DNA/RNA hybrid binding / Telomere C-strand synthesis initiation / Inhibition of replication initiation of damaged DNA by RB1/E2F1 / Polymerase switching / alpha DNA polymerase:primase complex / regulation of type I interferon production / Processive synthesis on the lagging strand / Removal of the Flap Intermediate / ribonucleotide binding / DNA replication initiation / DNA/RNA hybrid binding / Telomere C-strand synthesis initiation / Inhibition of replication initiation of damaged DNA by RB1/E2F1 / Polymerase switching / alpha DNA polymerase:primase complex / regulation of type I interferon production / Processive synthesis on the lagging strand / Removal of the Flap Intermediate /  DNA primase activity / Polymerase switching on the C-strand of the telomere / lagging strand elongation / mitotic DNA replication initiation / DNA primase activity / Polymerase switching on the C-strand of the telomere / lagging strand elongation / mitotic DNA replication initiation /  DNA replication, synthesis of primer / DNA strand elongation involved in DNA replication / leading strand elongation / DNA synthesis involved in DNA repair / G1/S-Specific Transcription / DNA replication, synthesis of primer / DNA strand elongation involved in DNA replication / leading strand elongation / DNA synthesis involved in DNA repair / G1/S-Specific Transcription /  DNA replication origin binding / DNA replication initiation / Activation of the pre-replicative complex / Defective pyroptosis / DNA replication origin binding / DNA replication initiation / Activation of the pre-replicative complex / Defective pyroptosis /  nuclear matrix / double-strand break repair via nonhomologous end joining / protein import into nucleus / nuclear matrix / double-strand break repair via nonhomologous end joining / protein import into nucleus /  single-stranded DNA binding / single-stranded DNA binding /  核膜 / 4 iron, 4 sulfur cluster binding / 核膜 / 4 iron, 4 sulfur cluster binding /  DNA複製 / DNA複製 /  DNAポリメラーゼ / DNAポリメラーゼ /  DNA-directed DNA polymerase activity / DNA-directed DNA polymerase activity /  nucleotide binding / nucleotide binding /  DNA修復 / DNA修復 /  chromatin binding / chromatin binding /  クロマチン / クロマチン /  核小体 / 核小体 /  protein kinase binding / magnesium ion binding / protein kinase binding / magnesium ion binding /  DNA binding / zinc ion binding / DNA binding / zinc ion binding /  核質 / 核質 /  生体膜 / 生体膜 /  metal ion binding / metal ion binding /  細胞核 / 細胞核 /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
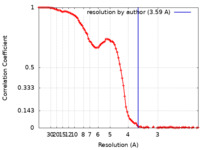
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.59 Å クライオ電子顕微鏡法 / 解像度: 3.59 Å | |||||||||
 データ登録者 データ登録者 | He Q / Baranovskiy A / Lim C / Tahirov T | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Structures of human primosome elongation complexes. 著者: Qixiang He / Andrey G Baranovskiy / Lucia M Morstadt / Alisa E Lisova / Nigar D Babayeva / Benjamin L Lusk / Ci Ji Lim / Tahir H Tahirov /  要旨: The synthesis of RNA-DNA primer by primosome requires coordination between primase and DNA polymerase α subunits, which is accompanied by unknown architectural rearrangements of multiple domains. ...The synthesis of RNA-DNA primer by primosome requires coordination between primase and DNA polymerase α subunits, which is accompanied by unknown architectural rearrangements of multiple domains. Using cryogenic electron microscopy, we solved a 3.6 Å human primosome structure caught at an early stage of RNA primer elongation with deoxynucleotides. The structure confirms a long-standing role of primase large subunit and reveals new insights into how primosome is limited to synthesizing short RNA-DNA primers. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27258.map.gz emd_27258.map.gz | 32.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27258-v30.xml emd-27258-v30.xml emd-27258.xml emd-27258.xml | 26.7 KB 26.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27258_fsc.xml emd_27258_fsc.xml | 11.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27258.png emd_27258.png | 120.2 KB | ||
| その他 |  emd_27258_half_map_1.map.gz emd_27258_half_map_1.map.gz emd_27258_half_map_2.map.gz emd_27258_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27258 http://ftp.pdbj.org/pub/emdb/structures/EMD-27258 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27258 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27258 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8d9dMC  8d96C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27258.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27258.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened and flipped map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.12 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map B
| ファイル | emd_27258_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_27258_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Elongation complex II of human DNA polymerase alpha/primase bound...
+超分子 #1: Elongation complex II of human DNA polymerase alpha/primase bound...
+分子 #1: DNA primase small subunit
+分子 #2: DNA primase large subunit
+分子 #3: DNA polymerase alpha catalytic subunit
+分子 #4: DNA polymerase alpha subunit B
+分子 #5: DNA/RNA (5'-D(*(GTP))-R(P*GP*CP*GP*GP*CP*AP*CP*G)-D(P*AP*CP*C)-3')
+分子 #6: DNA (5'-D(*AP*TP*GP*GP*TP*CP*GP*TP*GP*CP*CP*GP*CP*CP*AP*AP*TP*AP*...
+分子 #7: ZINC ION
+分子 #8: IRON/SULFUR CLUSTER
+分子 #9: MAGNESIUM ION
+分子 #10: 2'-DEOXYADENOSINE 5'-TRIPHOSPHATE
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: CHAPSO is made fresh at 80 mM before added to the sample at a final concentration of 4-8 mM immediately before vitrification. | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV | ||||||||||||||||||
| 詳細 | This sample was monodisperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 2 / 実像数: 13243 / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X