+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 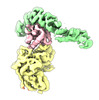 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Yeast TRAPPII-Rab11/Ypt32 complex in the closed/open state (focused refinement of Trs130 N-terminal region, Tca17, Trs33 and Bet3 in the closed monomer) | |||||||||
 マップデータ マップデータ | Map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  complex / complex /  GTPase (GTPアーゼ) / Guanosine Exchange Factor / GEF / GTPase (GTPアーゼ) / Guanosine Exchange Factor / GEF /  PROTEIN TRANSPORT PROTEIN TRANSPORT | |||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.2 Å クライオ電子顕微鏡法 / 解像度: 4.2 Å | |||||||||
 データ登録者 データ登録者 | Bagde SR / Fromme JC | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Structure of a TRAPPII-Rab11 activation intermediate reveals GTPase substrate selection mechanisms. 著者: Saket R Bagde / J Christopher Fromme /  要旨: Rab1 and Rab11 are essential regulators of the eukaryotic secretory and endocytic recycling pathways. The transport protein particle (TRAPP) complexes activate these guanosine triphosphatases via ...Rab1 and Rab11 are essential regulators of the eukaryotic secretory and endocytic recycling pathways. The transport protein particle (TRAPP) complexes activate these guanosine triphosphatases via nucleotide exchange using a shared set of core subunits. The basal specificity of the TRAPP core is toward Rab1, yet the TRAPPII complex is specific for Rab11. A steric gating mechanism has been proposed to explain TRAPPII counterselection against Rab1. Here, we present cryo-electron microscopy structures of the 22-subunit TRAPPII complex from budding yeast, including a TRAPPII-Rab11 nucleotide exchange intermediate. The Trs130 subunit provides a "leg" that positions the active site distal to the membrane surface, and this leg is required for steric gating. The related TRAPPIII complex is unable to activate Rab11 because of a repulsive interaction, which TRAPPII surmounts using the Trs120 subunit as a "lid" to enclose the active site. TRAPPII also adopts an open conformation enabling Rab11 to access and exit from the active site chamber. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26252.map.gz emd_26252.map.gz | 192.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26252-v30.xml emd-26252-v30.xml emd-26252.xml emd-26252.xml | 19.2 KB 19.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26252_fsc.xml emd_26252_fsc.xml | 14.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26252.png emd_26252.png | 103.5 KB | ||
| マスクデータ |  emd_26252_msk_1.map emd_26252_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-26252.cif.gz emd-26252.cif.gz | 4.9 KB | ||
| その他 |  emd_26252_additional_1.map.gz emd_26252_additional_1.map.gz emd_26252_half_map_1.map.gz emd_26252_half_map_1.map.gz emd_26252_half_map_2.map.gz emd_26252_half_map_2.map.gz | 10.3 MB 194.3 MB 193.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26252 http://ftp.pdbj.org/pub/emdb/structures/EMD-26252 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26252 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26252 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26252.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26252.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.432 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_26252_msk_1.map emd_26252_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: PostProcessed (sharpened) masked map
| ファイル | emd_26252_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PostProcessed (sharpened) masked map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_26252_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_26252_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : TRAPPII complex bound to Rab11/Ypt32
| 全体 | 名称: TRAPPII complex bound to Rab11/Ypt32 |
|---|---|
| 要素 |
|
-超分子 #1: TRAPPII complex bound to Rab11/Ypt32
| 超分子 | 名称: TRAPPII complex bound to Rab11/Ypt32 / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 分子量 | 理論値: 1.1 MDa |
-超分子 #2: Rab11/Ypt32
| 超分子 | 名称: Rab11/Ypt32 / タイプ: complex / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
-超分子 #3: TRAPPII
| 超分子 | 名称: TRAPPII / タイプ: complex / ID: 3 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5.6 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 50 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: The sample was incubated on the grid for 10 seconds followed by blotting for 5 seconds before plunging in liquid ethane.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 63000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 63000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 3 / 実像数: 4998 / 平均露光時間: 3.5 sec. / 平均電子線量: 53.0 e/Å2 / 詳細: Images were collected as 50 frame movies. |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



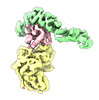






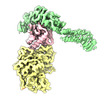

















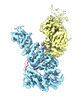

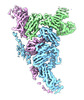











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)