+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
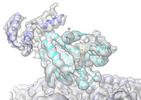
| タイトル | Complex of AMPPNP-Kif7 and Gli2 Zinc-Finger domain bound to microtubules | |||||||||
 マップデータ マップデータ | Complex of Kif7_AMPPNP and Gli2 Zinc-finger domain bound to microtubules | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ciliary tip / positive regulation of smoothened signaling pathway /  kinesin complex / kinesin complex /  microtubule motor activity / microtubule-based movement / Hedgehog 'off' state / negative regulation of smoothened signaling pathway / ciliary basal body / microtubule motor activity / microtubule-based movement / Hedgehog 'off' state / negative regulation of smoothened signaling pathway / ciliary basal body /  加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / Hedgehog 'on' state ...ciliary tip / positive regulation of smoothened signaling pathway / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / Hedgehog 'on' state ...ciliary tip / positive regulation of smoothened signaling pathway /  kinesin complex / kinesin complex /  microtubule motor activity / microtubule-based movement / Hedgehog 'off' state / negative regulation of smoothened signaling pathway / ciliary basal body / microtubule motor activity / microtubule-based movement / Hedgehog 'off' state / negative regulation of smoothened signaling pathway / ciliary basal body /  加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / Hedgehog 'on' state / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / Hedgehog 'on' state /  繊毛 / structural constituent of cytoskeleton / microtubule cytoskeleton organization / mitotic cell cycle / 繊毛 / structural constituent of cytoskeleton / microtubule cytoskeleton organization / mitotic cell cycle /  microtubule binding / microtubule binding /  微小管 / 微小管 /  hydrolase activity / hydrolase activity /  GTPase activity / GTP binding / GTPase activity / GTP binding /  ATP hydrolysis activity / ATP hydrolysis activity /  ATP binding / ATP binding /  metal ion binding / metal ion binding /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Sus scrofa (ブタ) / Sus scrofa (ブタ) /   Pig (ブタ) Pig (ブタ) | |||||||||
| 手法 | らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.89 Å クライオ電子顕微鏡法 / 解像度: 3.89 Å | |||||||||
 データ登録者 データ登録者 | Wilson-Kubalek EM / Mani N / Haque F / Freniere C / Milligan RA / Subramanian R | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Cell Biol / 年: 2022 ジャーナル: Nat Cell Biol / 年: 2022タイトル: Cytoskeletal regulation of a transcription factor by DNA mimicry via coiled-coil interactions. 著者: Farah Haque / Christian Freniere / Qiong Ye / Nandini Mani / Elizabeth M Wilson-Kubalek / Pei-I Ku / Ronald A Milligan / Radhika Subramanian /  要旨: A long-established strategy for transcription regulation is the tethering of transcription factors to cellular membranes. By contrast, the principal effectors of Hedgehog signalling, the GLI ...A long-established strategy for transcription regulation is the tethering of transcription factors to cellular membranes. By contrast, the principal effectors of Hedgehog signalling, the GLI transcription factors, are regulated by microtubules in the primary cilium and the cytoplasm. How GLI is tethered to microtubules remains unclear. Here, we uncover DNA mimicry by the ciliary kinesin KIF7 as a mechanism for the recruitment of GLI to microtubules, wherein the coiled-coil dimerization domain of KIF7, characterized by its striking shape, size and charge similarity to DNA, forms a complex with the DNA-binding zinc fingers in GLI, thus revealing a mode of tethering a DNA-binding protein to the cytoskeleton. GLI increases KIF7 microtubule affinity and consequently modulates the localization of both proteins to microtubules and the cilium tip. Thus, the kinesin-microtubule system is not a passive GLI tether but a regulatable platform tuned by the kinesin-transcription factor interaction. We retooled this coiled-coil-based GLI-KIF7 interaction to inhibit the nuclear and cilium localization of GLI. This strategy can potentially be exploited to downregulate erroneously activated GLI in human cancers. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24721.map.gz emd_24721.map.gz | 5.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24721-v30.xml emd-24721-v30.xml emd-24721.xml emd-24721.xml | 21.3 KB 21.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_24721.png emd_24721.png | 110.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24721 http://ftp.pdbj.org/pub/emdb/structures/EMD-24721 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24721 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24721 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7rx0MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24721.map.gz / 形式: CCP4 / 大きさ: 6.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24721.map.gz / 形式: CCP4 / 大きさ: 6.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Complex of Kif7_AMPPNP and Gli2 Zinc-finger domain bound to microtubules | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.3 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : Microtubule-bound complex of Kif7 and Gli2 zinc finger domain
+超分子 #1: Microtubule-bound complex of Kif7 and Gli2 zinc finger domain
+超分子 #2: Kinesin Kif7
+超分子 #3: Zinc-finger protein Gli2
+超分子 #4: Microtubules
+分子 #1: Kinesin-like protein KIF7
+分子 #2: Zinc finger protein GLI2
+分子 #3: Tubulin alpha-1A chain
+分子 #4: Tubulin beta chain
+分子 #5: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER
+分子 #6: GUANOSINE-5'-TRIPHOSPHATE
+分子 #7: PHOSPHOMETHYLPHOSPHONIC ACID GUANYLATE ESTER
+分子 #8: MAGNESIUM ION
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6.8 構成要素:
| |||||||||||||||
| グリッド | モデル: C-flat-1.2/1.3 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 280 K / 装置: HOMEMADE PLUNGER / 詳細: Blotted from behind the grid for 2 seconds. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 0-40 / 平均露光時間: 9.0 sec. / 平均電子線量: 36.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| Segment selection | 選択した数: 50639 詳細: Segments were picked along helical segments manually using Appion |
|---|---|
| CTF補正 | ソフトウェア - 名称: CTFFIND (ver. 4) / 詳細: CTF correction using CTFFIND4 |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 8.956 Å 想定した対称性 - らせんパラメータ - ΔΦ: -25.747 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) アルゴリズム: BACK PROJECTION / 解像度のタイプ: BY AUTHOR / 解像度: 3.89 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: FREALIX / 使用した粒子像数: 25730 |
| 詳細 | The images were corrected by motionCorr2 |
 ムービー
ムービー コントローラー
コントローラー