+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22732 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the SARS-CoV-2 S 2P trimer in complex with the human neutralizing antibody Fab fragment, C110 | |||||||||
 マップデータ マップデータ | Sharpened, non-uniform refined map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell ...Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / receptor-mediated endocytosis of virus by host cell / Attachment and Entry / membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / receptor ligand activity / symbiont-mediated suppression of host innate immune response / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Dam KA / Barnes CO / Bjorkman PJ | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2020 ジャーナル: Nature / 年: 2020タイトル: SARS-CoV-2 neutralizing antibody structures inform therapeutic strategies. 著者: Christopher O Barnes / Claudia A Jette / Morgan E Abernathy / Kim-Marie A Dam / Shannon R Esswein / Harry B Gristick / Andrey G Malyutin / Naima G Sharaf / Kathryn E Huey-Tubman / Yu E Lee / ...著者: Christopher O Barnes / Claudia A Jette / Morgan E Abernathy / Kim-Marie A Dam / Shannon R Esswein / Harry B Gristick / Andrey G Malyutin / Naima G Sharaf / Kathryn E Huey-Tubman / Yu E Lee / Davide F Robbiani / Michel C Nussenzweig / Anthony P West / Pamela J Bjorkman /   要旨: The coronavirus disease 2019 (COVID-19) pandemic presents an urgent health crisis. Human neutralizing antibodies that target the host ACE2 receptor-binding domain (RBD) of the severe acute ...The coronavirus disease 2019 (COVID-19) pandemic presents an urgent health crisis. Human neutralizing antibodies that target the host ACE2 receptor-binding domain (RBD) of the severe acute respiratory syndrome coronavirus-2 (SARS-CoV-2) spike protein show promise therapeutically and are being evaluated clinically. Here, to identify the structural correlates of SARS-CoV-2 neutralization, we solved eight new structures of distinct COVID-19 human neutralizing antibodies in complex with the SARS-CoV-2 spike trimer or RBD. Structural comparisons allowed us to classify the antibodies into categories: (1) neutralizing antibodies encoded by the VH3-53 gene segment with short CDRH3 loops that block ACE2 and bind only to 'up' RBDs; (2) ACE2-blocking neutralizing antibodies that bind both up and 'down' RBDs and can contact adjacent RBDs; (3) neutralizing antibodies that bind outside the ACE2 site and recognize both up and down RBDs; and (4) previously described antibodies that do not block ACE2 and bind only to up RBDs. Class 2 contained four neutralizing antibodies with epitopes that bridged RBDs, including a VH3-53 antibody that used a long CDRH3 with a hydrophobic tip to bridge between adjacent down RBDs, thereby locking the spike into a closed conformation. Epitope and paratope mapping revealed few interactions with host-derived N-glycans and minor contributions of antibody somatic hypermutations to epitope contacts. Affinity measurements and mapping of naturally occurring and in vitro-selected spike mutants in 3D provided insight into the potential for SARS-CoV-2 to escape from antibodies elicited during infection or delivered therapeutically. These classifications and structural analyses provide rules for assigning current and future human RBD-targeting antibodies into classes, evaluating avidity effects and suggesting combinations for clinical use, and provide insight into immune responses against SARS-CoV-2. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22732.map.gz emd_22732.map.gz | 157.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22732-v30.xml emd-22732-v30.xml emd-22732.xml emd-22732.xml | 27.8 KB 27.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
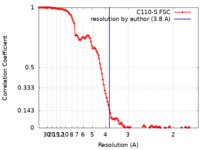
| FSC (解像度算出) |  emd_22732_fsc.xml emd_22732_fsc.xml | 12.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22732.png emd_22732.png | 18.6 KB | ||
| その他 |  emd_22732_additional_1.map.gz emd_22732_additional_1.map.gz emd_22732_additional_2.map.gz emd_22732_additional_2.map.gz emd_22732_additional_3.map.gz emd_22732_additional_3.map.gz | 82.8 MB 136.7 MB 157.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22732 http://ftp.pdbj.org/pub/emdb/structures/EMD-22732 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22732 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22732 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22732_validation.pdf.gz emd_22732_validation.pdf.gz | 447.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22732_full_validation.pdf.gz emd_22732_full_validation.pdf.gz | 446.9 KB | 表示 | |
| XML形式データ |  emd_22732_validation.xml.gz emd_22732_validation.xml.gz | 13 KB | 表示 | |
| CIF形式データ |  emd_22732_validation.cif.gz emd_22732_validation.cif.gz | 17.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22732 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22732 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22732 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22732 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7k8vMC  7k8mC  7k8nC  7k8oC  7k8pC  7k8qC  7k8rC  7k8sC  7k8tC  7k8uC  7k8wC  7k8xC  7k8yC  7k8zC  7k90C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22732.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22732.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened, non-uniform refined map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.836 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
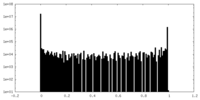
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Unsharpened, non-uniform refined map
| ファイル | emd_22732_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened, non-uniform refined map | ||||||||||||
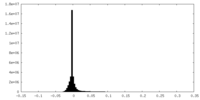
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Mask used for local refinement
| ファイル | emd_22732_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Mask used for local refinement | ||||||||||||
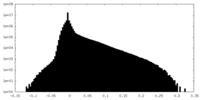
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Sharpened, local refinement map
| ファイル | emd_22732_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened, local refinement map | ||||||||||||
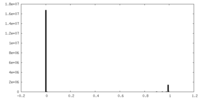
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of SARS-CoV-2 S 2P trimer with human neutralizing...
| 全体 | 名称: Ternary complex of SARS-CoV-2 S 2P trimer with human neutralizing antibody Fab fragment C110 |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of SARS-CoV-2 S 2P trimer with human neutralizing...
| 超分子 | 名称: Ternary complex of SARS-CoV-2 S 2P trimer with human neutralizing antibody Fab fragment C110 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 分子量 | 実験値: 720 KDa |
-超分子 #2: SARS-CoV-2 S 2P glycoprotein
| 超分子 | 名称: SARS-CoV-2 S 2P glycoprotein / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: neutralizing antibody Fab fragment C110
| 超分子 | 名称: neutralizing antibody Fab fragment C110 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Spike glycoprotein
| 分子 | 名称: Spike glycoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 139.788953 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDG VYFASTEKSN IIRGWIFGTT LDSKTQSLLI VNNATNVVIK VCEFQFCNDP FLGVYYHKNN KSWMESEFRV Y SSANNCTF ...文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDG VYFASTEKSN IIRGWIFGTT LDSKTQSLLI VNNATNVVIK VCEFQFCNDP FLGVYYHKNN KSWMESEFRV Y SSANNCTF EYVSQPFLMD LEGKQGNFKN LREFVFKNID GYFKIYSKHT PINLVRDLPQ GFSALEPLVD LPIGINITRF QT LLALHRS YLTPGDSSSG WTAGAAAYYV GYLQPRTFLL KYNENGTITD AVDCALDPLS ETKCTLKSFT VEKGIYQTSN FRV QPTESI VRFPNITNLC PFGEVFNATR FASVYAWNRK RISNCVADYS VLYNSASFST FKCYGVSPTK LNDLCFTNVY ADSF VIRGD EVRQIAPGQT GKIADYNYKL PDDFTGCVIA WNSNNLDSKV GGNYNYLYRL FRKSNLKPFE RDISTEIYQA GSTPC NGVE GFNCYFPLQS YGFQPTNGVG YQPYRVVVLS FELLHAPATV CGPKKSTNLV KNKCVNFNFN GLTGTGVLTE SNKKFL PFQ QFGRDIADTT DAVRDPQTLE ILDITPCSFG GVSVITPGTN TSNEVAVLYQ DVNCTEVPVA IHADQLTPTW RVYSTGS NV FQTRAGCLIG AEHVNNSYEC DIPIGAGICA SYQTQTNSPR RARSVASQSI IAYTMSLGAE NSVAYSNNSI AIPTNFTI S VTTEILPVSM TKTSVDCTMY ICGDSTECSN LLLQYGSFCT QLNRALTGIA VEQDKNTQEV FAQVKQIYKT PPIKDFGGF NFSQILPDPS KPSKRSFIED LLFNKVTLAD AGFIKQYGDC LGDIAARDLI CAQKFNGLTV LPPLLTDEMI AQYTSALLAG TITSGWTFG AGAALQIPFA MQMAYRFNGI GVTQNVLYEN QKLIANQFNS AIGKIQDSLS STASALGKLQ DVVNQNAQAL N TLVKQLSS NFGAISSVLN DILSRLDPPE AEVQIDRLIT GRLQSLQTYV TQQLIRAAEI RASANLAATK MSECVLGQSK RV DFCGKGY HLMSFPQSAP HGVVFLHVTY VPAQEKNFTT APAICHDGKA HFPREGVFVS NGTHWFVTQR NFYEPQIITT DNT FVSGNC DVVIGIVNNT VYDPLQPELD SFKEELDKYF KNHTSPDVDL GDISGINASV VNIQKEIDRL NEVAKNLNES LIDL QELGK YEQYIKWPSG RLVPRGSPGS GYIPEAPRDG QAYVRKDGEW VLLSTFLGHH HHHH |
-分子 #2: C110 Fab Heavy Chain
| 分子 | 名称: C110 Fab Heavy Chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.825955 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLQQSGAE VKKPGESLKI SCKGSGYSFT SYWIGWVRQM PGKGLEWMGI IYPGDSDTRY SPSFQGQVTI SADKSISTAY MQWSSLKAS DTAMYYCARS FRDDPRIAVA GPADAFDIWG QGTMVTVSSA STKGPSVFPL APSSKSTSGG TAALGCLVKD Y FPEPVTVS ...文字列: QVQLQQSGAE VKKPGESLKI SCKGSGYSFT SYWIGWVRQM PGKGLEWMGI IYPGDSDTRY SPSFQGQVTI SADKSISTAY MQWSSLKAS DTAMYYCARS FRDDPRIAVA GPADAFDIWG QGTMVTVSSA STKGPSVFPL APSSKSTSGG TAALGCLVKD Y FPEPVTVS WNSGALTSGV HTFPAVLQSS GLYSLSSVVT VPSSSLGTQT YICNVNHKPS NTKVDKRVEP KSCDKTHHHH HH |
-分子 #3: C110 Fab Light Chain
| 分子 | 名称: C110 Fab Light Chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.65318 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIQMTQSPST LSASVGDRVT ITCRASQSIS YWLAWYQQKP GKAPKLLIYQ ASSLESGVPS RFSGSESGTE FTLTISSLQP DDFATYYCQ QYNSYPYTFG QGTKLEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS ...文字列: DIQMTQSPST LSASVGDRVT ITCRASQSIS YWLAWYQQKP GKAPKLLIYQ ASSLESGVPS RFSGSESGTE FTLTISSLQP DDFATYYCQ QYNSYPYTFG QGTKLEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS KDSTYSLSST LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRGEC |
-分子 #5: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 5 / コピー数: 21 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 詳細: 0.2 mA | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV / 詳細: 3s blot. | |||||||||
| 詳細 | Monodisperse sample |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 1 / 実像数: 2097 / 平均露光時間: 2.6 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

























 Z
Z Y
Y X
X