+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22376 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | cryo-EM structure of human ATG9A in nanodiscs | |||||||||
 マップデータ マップデータ | primary map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 phospholipid scramblase activity / programmed necrotic cell death / nucleophagy / protein localization to phagophore assembly site / phagophore assembly site membrane / phagophore assembly site / bone morphogenesis / phospholipid scramblase activity / programmed necrotic cell death / nucleophagy / protein localization to phagophore assembly site / phagophore assembly site membrane / phagophore assembly site / bone morphogenesis /  オートファジー / オートファジー /  autophagosome assembly / autophagosome assembly /  オートファゴソーム ... オートファゴソーム ... phospholipid scramblase activity / programmed necrotic cell death / nucleophagy / protein localization to phagophore assembly site / phagophore assembly site membrane / phagophore assembly site / bone morphogenesis / phospholipid scramblase activity / programmed necrotic cell death / nucleophagy / protein localization to phagophore assembly site / phagophore assembly site membrane / phagophore assembly site / bone morphogenesis /  オートファジー / オートファジー /  autophagosome assembly / autophagosome assembly /  オートファゴソーム / オートファゴソーム /  ミトコンドリア / ミトコンドリア /  ゴルジ体 / recycling endosome / recycling endosome membrane / late endosome / late endosome membrane / ゴルジ体 / recycling endosome / recycling endosome membrane / late endosome / late endosome membrane /  エンドソーム / エンドソーム /  ゴルジ体 / intracellular membrane-bounded organelle / endoplasmic reticulum membrane / ゴルジ体 / intracellular membrane-bounded organelle / endoplasmic reticulum membrane /  ゴルジ体 / ゴルジ体 /  ミトコンドリア / ミトコンドリア /  生体膜 生体膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
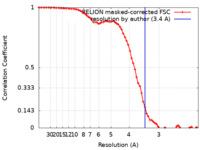
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.4 Å クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Maeda S / Otomo T | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2020 ジャーナル: Nat Struct Mol Biol / 年: 2020タイトル: Structure, lipid scrambling activity and role in autophagosome formation of ATG9A. 著者: Shintaro Maeda / Hayashi Yamamoto / Lisa N Kinch / Christina M Garza / Satoru Takahashi / Chinatsu Otomo / Nick V Grishin / Stefano Forli / Noboru Mizushima / Takanori Otomo /   要旨: De novo formation of the double-membrane compartment autophagosome is seeded by small vesicles carrying membrane protein autophagy-related 9 (ATG9), the function of which remains unknown. Here we ...De novo formation of the double-membrane compartment autophagosome is seeded by small vesicles carrying membrane protein autophagy-related 9 (ATG9), the function of which remains unknown. Here we find that ATG9A scrambles phospholipids of membranes in vitro. Cryo-EM structures of human ATG9A reveal a trimer with a solvated central pore, which is connected laterally to the cytosol through the cavity within each protomer. Similarities to ABC exporters suggest that ATG9A could be a transporter that uses the central pore to function. Moreover, molecular dynamics simulation suggests that the central pore opens laterally to accommodate lipid headgroups, thereby enabling lipids to flip. Mutations in the pore reduce scrambling activity and yield markedly smaller autophagosomes, indicating that lipid scrambling by ATG9A is essential for membrane expansion. We propose ATG9A acts as a membrane-embedded funnel to facilitate lipid flipping and to redistribute lipids added to the outer leaflet of ATG9 vesicles, thereby enabling growth into autophagosomes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22376.map.gz emd_22376.map.gz | 25.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22376-v30.xml emd-22376-v30.xml emd-22376.xml emd-22376.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22376_fsc.xml emd_22376_fsc.xml | 6.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22376.png emd_22376.png | 57.3 KB | ||
| その他 |  emd_22376_additional_1.map.gz emd_22376_additional_1.map.gz emd_22376_half_map_1.map.gz emd_22376_half_map_1.map.gz emd_22376_half_map_2.map.gz emd_22376_half_map_2.map.gz | 20.5 MB 20.6 MB 20.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22376 http://ftp.pdbj.org/pub/emdb/structures/EMD-22376 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22376 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22376 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22376.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22376.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | primary map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.134 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: raw map
| ファイル | emd_22376_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | raw map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
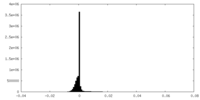
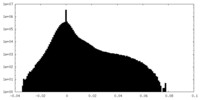
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_22376_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
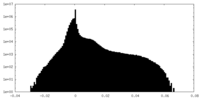
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_22376_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
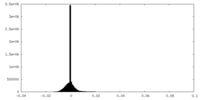
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human ATG9A in MSP2N2 nanodiscs
| 全体 | 名称: Human ATG9A in MSP2N2 nanodiscs |
|---|---|
| 要素 |
|
-超分子 #1: Human ATG9A in MSP2N2 nanodiscs
| 超分子 | 名称: Human ATG9A in MSP2N2 nanodiscs / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
-分子 #1: Autophagy-related protein 9A
| 分子 | 名称: Autophagy-related protein 9A / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 66.797344 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MAQFDTEYQR LEASYSDSPP GEEDLLVHVA EGSKSPWHHI ENLDLFFSRV YNLHQKNGFT CMLIGEIFEL MQFLFVVAFT TFLVSCVDY DILFANKMVN HSLHPTEPVK VTLPDAFLPA QVCSARIQEN GSLITILVIA GVFWIHRLIK FIYNICCYWE I HSFYLHAL ...文字列: MAQFDTEYQR LEASYSDSPP GEEDLLVHVA EGSKSPWHHI ENLDLFFSRV YNLHQKNGFT CMLIGEIFEL MQFLFVVAFT TFLVSCVDY DILFANKMVN HSLHPTEPVK VTLPDAFLPA QVCSARIQEN GSLITILVIA GVFWIHRLIK FIYNICCYWE I HSFYLHAL RIPMSALPYC TWQEVQARIV QTQKEHQICI HKRELTELDI YHRILRFQNY MVALVNKSLL PLRFRLPGLG EA VFFTRGL KYNFELILFW GPGSLFLNEW SLKAEYKRGG QRLELAQRLS NRILWIGIAN FLLCPLILIW QILYAFFSYA EVL KREPGA LGARCWSLYG RCYLRHFNEL EHELQSRLNR GYKPASKYMN CFLSPLLTLL AKNGAFFAGS ILAVLIALTI YDED VLAVE HVLTTVTLLG VTVTVCRSFI PDQHMVFCPE QLLRVILAHI HYMPDHWQGN AHRSQTRDEF AQLFQYKAVF ILEEL LSPI VTPLILIFCL RPRALEIIDF FRNFTVEVVG VGDTCSFAQM DVRQHGHPQW LSAGQTEASV YQQAEDGKTE LSLMHF AIT NPGWQPPRES TAFLG |
-分子 #2: (2S)-3-(hexadecanoyloxy)-2-[(9Z)-octadec-9-enoyloxy]propyl 2-(tri...
| 分子 | 名称: (2S)-3-(hexadecanoyloxy)-2-[(9Z)-octadec-9-enoyloxy]propyl 2-(trimethylammonio)ethyl phosphate タイプ: ligand / ID: 2 / コピー数: 45 / 式: POV |
|---|---|
| 分子量 | 理論値: 760.076 Da |
| Chemical component information |  ChemComp-POV: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 9 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 88 % / チャンバー内温度: 277 K / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 73000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 73000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 66.0 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z
Z Y
Y X
X