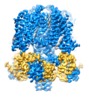
登録情報 データベース : EMDB / ID : EMD-20965タイトル structure of human KCNQ1-CaM complex human KCNQ1 複合体 : KCNQ1-CaM complexタンパク質・ペプチド : Potassium voltage-gated channel subfamily KQT member 1タンパク質・ペプチド : Calmodulin-1リガンド : CALCIUM ION / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / 解像度 : 3.1 Å Mackinnon R / Sun J 資金援助 Organization Grant number 国 National Institutes of Health/National Heart, Lung, and Blood Institute (NIH/NHLBI) 5K99HL143037 Howard Hughes Medical Institute (HHMI)
ジャーナル : Cell / 年 : 2020タイトル : Structural Basis of Human KCNQ1 Modulation and Gating.著者 : Ji Sun / Roderick MacKinnon / 要旨 : KCNQ1, also known as Kv7.1, is a voltage-dependent K channel that regulates gastric acid secretion, salt and glucose homeostasis, and heart rhythm. Its functional properties are regulated in a tissue- ... KCNQ1, also known as Kv7.1, is a voltage-dependent K channel that regulates gastric acid secretion, salt and glucose homeostasis, and heart rhythm. Its functional properties are regulated in a tissue-specific manner through co-assembly with beta subunits KCNE1-5. In non-excitable cells, KCNQ1 forms a complex with KCNE3, which suppresses channel closure at negative membrane voltages that otherwise would close it. Pore opening is regulated by the signaling lipid PIP2. Using cryoelectron microscopy (cryo-EM), we show that KCNE3 tucks its single-membrane-spanning helix against KCNQ1, at a location that appears to lock the voltage sensor in its depolarized conformation. Without PIP2, the pore remains closed. Upon addition, PIP2 occupies a site on KCNQ1 within the inner membrane leaflet, which triggers a large conformational change that leads to dilation of the pore's gate. It is likely that this mechanism of PIP2 activation is conserved among Kv7 channels. 履歴 登録 2019年11月16日 - ヘッダ(付随情報) 公開 2019年11月27日 - マップ公開 2019年12月4日 - 更新 2024年3月6日 - 現状 2024年3月6日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ KvLQT1
KvLQT1  試料
試料 キーワード
キーワード potassium channel (カリウムチャネル) /
potassium channel (カリウムチャネル) /  KCNQ1 / CaM /
KCNQ1 / CaM /  MEMBRANE PROTEIN (膜タンパク質)
MEMBRANE PROTEIN (膜タンパク質) 機能・相同性情報
機能・相同性情報 protein phosphatase 1 binding / delayed rectifier potassium channel activity / positive regulation of potassium ion transmembrane transport / Voltage gated Potassium channels / outward rectifier potassium channel activity / potassium ion homeostasis / ventricular cardiac muscle cell action potential / non-motile cilium assembly / regulation of ventricular cardiac muscle cell membrane repolarization /
protein phosphatase 1 binding / delayed rectifier potassium channel activity / positive regulation of potassium ion transmembrane transport / Voltage gated Potassium channels / outward rectifier potassium channel activity / potassium ion homeostasis / ventricular cardiac muscle cell action potential / non-motile cilium assembly / regulation of ventricular cardiac muscle cell membrane repolarization /  CAM型光合成 / Cam-PDE 1 activation / intestinal absorption / Sodium/Calcium exchangers /
CAM型光合成 / Cam-PDE 1 activation / intestinal absorption / Sodium/Calcium exchangers /  regulation of heart contraction / Calmodulin induced events / Reduction of cytosolic Ca++ levels / Activation of Ca-permeable Kainate Receptor / CREB1 phosphorylation through the activation of CaMKII/CaMKK/CaMKIV cascasde / Loss of phosphorylation of MECP2 at T308 / CREB1 phosphorylation through the activation of Adenylate Cyclase / PKA activation / negative regulation of high voltage-gated calcium channel activity / ciliary base / monoatomic ion channel complex / inner ear morphogenesis / CaMK IV-mediated phosphorylation of CREB / Glycogen breakdown (glycogenolysis) / negative regulation of calcium ion export across plasma membrane / positive regulation of heart rate / organelle localization by membrane tethering / Activation of RAC1 downstream of NMDARs / regulation of cardiac muscle cell action potential / mitochondrion-endoplasmic reticulum membrane tethering / CLEC7A (Dectin-1) induces NFAT activation / autophagosome membrane docking / cochlea development / renal absorption / adrenergic receptor signaling pathway / positive regulation of ryanodine-sensitive calcium-release channel activity / potassium ion import across plasma membrane / Negative regulation of NMDA receptor-mediated neuronal transmission / regulation of cell communication by electrical coupling involved in cardiac conduction / Unblocking of NMDA receptors, glutamate binding and activation / negative regulation of peptidyl-threonine phosphorylation / Synthesis of IP3 and IP4 in the cytosol / regulation of heart rate by cardiac conduction / Phase 0 - rapid depolarisation / protein kinase A regulatory subunit binding / protein phosphatase activator activity / RHO GTPases activate PAKs /
regulation of heart contraction / Calmodulin induced events / Reduction of cytosolic Ca++ levels / Activation of Ca-permeable Kainate Receptor / CREB1 phosphorylation through the activation of CaMKII/CaMKK/CaMKIV cascasde / Loss of phosphorylation of MECP2 at T308 / CREB1 phosphorylation through the activation of Adenylate Cyclase / PKA activation / negative regulation of high voltage-gated calcium channel activity / ciliary base / monoatomic ion channel complex / inner ear morphogenesis / CaMK IV-mediated phosphorylation of CREB / Glycogen breakdown (glycogenolysis) / negative regulation of calcium ion export across plasma membrane / positive regulation of heart rate / organelle localization by membrane tethering / Activation of RAC1 downstream of NMDARs / regulation of cardiac muscle cell action potential / mitochondrion-endoplasmic reticulum membrane tethering / CLEC7A (Dectin-1) induces NFAT activation / autophagosome membrane docking / cochlea development / renal absorption / adrenergic receptor signaling pathway / positive regulation of ryanodine-sensitive calcium-release channel activity / potassium ion import across plasma membrane / Negative regulation of NMDA receptor-mediated neuronal transmission / regulation of cell communication by electrical coupling involved in cardiac conduction / Unblocking of NMDA receptors, glutamate binding and activation / negative regulation of peptidyl-threonine phosphorylation / Synthesis of IP3 and IP4 in the cytosol / regulation of heart rate by cardiac conduction / Phase 0 - rapid depolarisation / protein kinase A regulatory subunit binding / protein phosphatase activator activity / RHO GTPases activate PAKs /  voltage-gated potassium channel activity / protein kinase A catalytic subunit binding / inner ear development /
voltage-gated potassium channel activity / protein kinase A catalytic subunit binding / inner ear development /  social behavior / positive regulation of cyclic-nucleotide phosphodiesterase activity / positive regulation of phosphoprotein phosphatase activity /
social behavior / positive regulation of cyclic-nucleotide phosphodiesterase activity / positive regulation of phosphoprotein phosphatase activity /  長期増強 / Ion transport by P-type ATPases / Uptake and function of anthrax toxins / Calcineurin activates NFAT / Regulation of MECP2 expression and activity /
長期増強 / Ion transport by P-type ATPases / Uptake and function of anthrax toxins / Calcineurin activates NFAT / Regulation of MECP2 expression and activity /  catalytic complex / DARPP-32 events / detection of calcium ion / negative regulation of ryanodine-sensitive calcium-release channel activity / Smooth Muscle Contraction / RHO GTPases activate IQGAPs / regulation of cardiac muscle contraction / calcium channel inhibitor activity / cellular response to interferon-beta / regulation of cardiac muscle contraction by regulation of the release of sequestered calcium ion /
catalytic complex / DARPP-32 events / detection of calcium ion / negative regulation of ryanodine-sensitive calcium-release channel activity / Smooth Muscle Contraction / RHO GTPases activate IQGAPs / regulation of cardiac muscle contraction / calcium channel inhibitor activity / cellular response to interferon-beta / regulation of cardiac muscle contraction by regulation of the release of sequestered calcium ion /  Protein methylation /
Protein methylation /  voltage-gated potassium channel complex
voltage-gated potassium channel complex
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 単粒子再構成法 /
単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.1 Å
クライオ電子顕微鏡法 / 解像度: 3.1 Å  データ登録者
データ登録者 米国, 2件
米国, 2件  引用
引用 ジャーナル: Cell / 年: 2020
ジャーナル: Cell / 年: 2020
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_20965.map.gz
emd_20965.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-20965-v30.xml
emd-20965-v30.xml emd-20965.xml
emd-20965.xml EMDBヘッダ
EMDBヘッダ emd_20965.png
emd_20965.png emd-20965.cif.gz
emd-20965.cif.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-20965
http://ftp.pdbj.org/pub/emdb/structures/EMD-20965 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20965
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20965 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_20965.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_20965.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Homo sapiens (ヒト)
Homo sapiens (ヒト) Calmodulin-1
Calmodulin-1 クライオ電子顕微鏡法
クライオ電子顕微鏡法 解析
解析 単粒子再構成法
単粒子再構成法 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN Bright-field microscopy
Bright-field microscopy
 画像解析
画像解析 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION / 使用した粒子像数: 66746
) / 解像度のタイプ: BY AUTHOR / 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION / 使用した粒子像数: 66746 
 ムービー
ムービー コントローラー
コントローラー