+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20015 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of filamentous SgrAI endonuclease in its activated form | |||||||||
 マップデータ マップデータ | Cryo-EM reconstruction of filamentous SgrAI in its activated form | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  restriction endonuclease (制限酵素) / restriction endonuclease (制限酵素) /  DNAse (デオキシリボヌクレアーゼ) / DNAse (デオキシリボヌクレアーゼ) /  allostery (アロステリック効果) / bacterial innate immunity / allostery (アロステリック効果) / bacterial innate immunity /  filament / hyper-activation / filament / hyper-activation /  substrate specificity / HYDROLASE-DNA complex substrate specificity / HYDROLASE-DNA complex | |||||||||
| 機能・相同性 |  Restriction endonuclease, type II, Cfr10I/Bse634I / Cfr10I/Bse634I restriction endonuclease / Restriction endonuclease type II-like / identical protein binding / Restriction endonuclease, type II, Cfr10I/Bse634I / Cfr10I/Bse634I restriction endonuclease / Restriction endonuclease type II-like / identical protein binding /  metal ion binding / SgraIR restriction enzyme metal ion binding / SgraIR restriction enzyme 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Streptomyces griseus (ストレプトマイシン生産菌) Streptomyces griseus (ストレプトマイシン生産菌) | |||||||||
| 手法 | らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.5 Å クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Polley S / Lyumkis D | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2019 ジャーナル: Structure / 年: 2019タイトル: Mechanism of Filamentation-Induced Allosteric Activation of the SgrAI Endonuclease. 著者: Smarajit Polley / Dmitry Lyumkis / Nancy C Horton /   要旨: Filament formation by enzymes is increasingly recognized as an important phenomenon with potentially unique regulatory properties and biological roles. SgrAI is an allosterically regulated type II ...Filament formation by enzymes is increasingly recognized as an important phenomenon with potentially unique regulatory properties and biological roles. SgrAI is an allosterically regulated type II restriction endonuclease that forms filaments with enhanced DNA cleavage activity and altered sequence specificity. Here, we present the cryoelectron microscopy (cryo-EM) structure of the filament of SgrAI in its activated configuration. The structural data illuminate the mechanistic origin of hyperaccelerated DNA cleavage activity and suggests how indirect DNA sequence readout within filamentous SgrAI may enable recognition of substantially more nucleotide sequences than its low-activity form, thereby altering and partially relaxing its DNA sequence specificity. Together, substrate DNA binding, indirect readout, and filamentation simultaneously enhance SgrAI's catalytic activity and modulate substrate preference. This unusual enzyme mechanism may have evolved to perform the specialized functions of bacterial innate immunity in rapid defense against invading phage DNA without causing damage to the host DNA. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20015.map.gz emd_20015.map.gz | 58.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20015-v30.xml emd-20015-v30.xml emd-20015.xml emd-20015.xml | 19 KB 19 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20015.png emd_20015.png | 195 KB | ||
| マスクデータ |  emd_20015_msk_1.map emd_20015_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-20015.cif.gz emd-20015.cif.gz | 6.6 KB | ||
| その他 |  emd_20015_half_map_1.map.gz emd_20015_half_map_1.map.gz emd_20015_half_map_2.map.gz emd_20015_half_map_2.map.gz | 9.9 MB 9.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20015 http://ftp.pdbj.org/pub/emdb/structures/EMD-20015 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20015 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20015 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20015.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20015.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM reconstruction of filamentous SgrAI in its activated form | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.31 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_20015_msk_1.map emd_20015_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
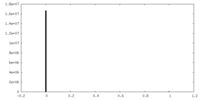
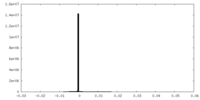
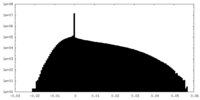
| 投影像・断面図 |
| ||||||||||||
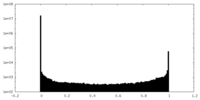
| 密度ヒストグラム |
-ハーフマップ: half-map 1 for the Cryo-EM reconstruction of filamentous...
| ファイル | emd_20015_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map 1 for the Cryo-EM reconstruction of filamentous SgrAI in its activated form | ||||||||||||
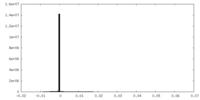
| 投影像・断面図 |
| ||||||||||||
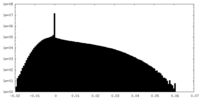
| 密度ヒストグラム |
-ハーフマップ: half-map 2 for the Cryo-EM reconstruction of filamentous...
| ファイル | emd_20015_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map 2 for the Cryo-EM reconstruction of filamentous SgrAI in its activated form | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Filamentous assembly of SgrAI protein bound to pre-cleaved DNA
| 全体 | 名称: Filamentous assembly of SgrAI protein bound to pre-cleaved DNA |
|---|---|
| 要素 |
|
-超分子 #1: Filamentous assembly of SgrAI protein bound to pre-cleaved DNA
| 超分子 | 名称: Filamentous assembly of SgrAI protein bound to pre-cleaved DNA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Streptomyces griseus (ストレプトマイシン生産菌) Streptomyces griseus (ストレプトマイシン生産菌) |
| 分子量 | 理論値: 74 kDa/nm |
-分子 #1: SgraIR restriction enzyme
| 分子 | 名称: SgraIR restriction enzyme / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO EC番号:  加水分解酵素; エステル加水分解酵素; 5'-リン酸モノエステル産生エンドデオキシリボヌクレアーゼ 加水分解酵素; エステル加水分解酵素; 5'-リン酸モノエステル産生エンドデオキシリボヌクレアーゼ |
|---|---|
| 由来(天然) | 生物種:   Streptomyces griseus (ストレプトマイシン生産菌) Streptomyces griseus (ストレプトマイシン生産菌) |
| 分子量 | 理論値: 38.073102 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MPFTYSIEAT RNLATTERCI QDIRNAPVRN RSTQFQLAQQ NMLAYTFGEV IPGFASAGIN GMDYRDVIGR PVENAVTEGT HFFRDDFRV DSNAKAKVAG DIFEIVSSAV MWNCAARWNS LMVGEGWRSQ PRYSRPTLSP SPRRQVAVLN LPRSFDWVSL L VPESQEVI ...文字列: MPFTYSIEAT RNLATTERCI QDIRNAPVRN RSTQFQLAQQ NMLAYTFGEV IPGFASAGIN GMDYRDVIGR PVENAVTEGT HFFRDDFRV DSNAKAKVAG DIFEIVSSAV MWNCAARWNS LMVGEGWRSQ PRYSRPTLSP SPRRQVAVLN LPRSFDWVSL L VPESQEVI EEFRAGLRKD GLGLPTSTPD LAVVVLPEEF QNDEMWREEI AGLTRPNQIL LSGAYQRLQG RVQPGEISLA VA FKRSLRS DRLYQPLYEA NVMQLLLEGK LGAPKVEFEV HTLAPEGTNA FVTYEAASLY GLAEGRSAVH RAIRELYVPP TAA DLARRF FAFLNERMEL VNG UniProtKB: SgraIR restriction enzyme |
-分子 #2: DNA (26-MER)
| 分子 | 名称: DNA (26-MER) / タイプ: dna / ID: 2 / 詳細: assembled from pre-cleaved DNAs / コピー数: 2 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Streptomyces griseus (ストレプトマイシン生産菌) Streptomyces griseus (ストレプトマイシン生産菌) |
| 分子量 | 理論値: 12.314889 KDa |
| 配列 | 文字列: (DG)(DA)(DT)(DG)(DC)(DG)(DT)(DG)(DG)(DG) (DT)(DC)(DT)(DT)(DC)(DA)(DC)(DA)(DC)(DC) (DG)(DG)(DT)(DG)(DT)(DG)(DA)(DA)(DG) (DA)(DC)(DC)(DC)(DA)(DC)(DG)(DC)(DA)(DT) (DC) |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||||||||
| グリッド | モデル: UltrAuFoil / 材質: GOLD / メッシュ: 400 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 6 sec. / 前処理 - 雰囲気: OTHER | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 38167 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 22500 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-60 / 実像数: 216 / 平均電子線量: 55.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: RELION (ver. 3.0) |
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 21.6 Å 想定した対称性 - らせんパラメータ - ΔΦ: -86.2 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) アルゴリズム: FOURIER SPACE / 解像度のタイプ: BY AUTHOR / 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.0) / 使用した粒子像数: 7 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 当てはまり具合の基準: Correlation coefficient |
|---|---|
| 得られたモデル |  PDB-6obj: |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X