+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Atomic structure and conformational variability of the HER2-Trastuzumab-Pertuzumab complex | |||||||||
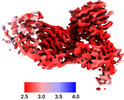
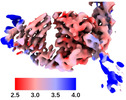
 マップデータ マップデータ | Composite map visible in figure 1 d | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  ErbB-2 (HER2) / Pertuzumab / ErbB-2 (HER2) / Pertuzumab /  Trastuzumab (トラスツズマブ) / Trastuzumab (トラスツズマブ) /  Ternary complex / Ternary complex /  Protein (タンパク質) / Protein (タンパク質) /  Flexibility (剛性) / Continuous conformation / Flexibility (剛性) / Continuous conformation /  Cryo-EM (低温電子顕微鏡法) / Cryo-EM (低温電子顕微鏡法) /  Single particle analysis (単粒子解析法) / Single particle analysis (単粒子解析法) /  STRUCTURAL PROTEIN (タンパク質) STRUCTURAL PROTEIN (タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of immature T cell proliferation in thymus / ERBB3:ERBB2 complex / ERBB2-ERBB4 signaling pathway / GRB7 events in ERBB2 signaling / immature T cell proliferation in thymus / RNA polymerase I core binding / semaphorin receptor complex / regulation of microtubule-based process / ErbB-3 class receptor binding / Sema4D induced cell migration and growth-cone collapse ...negative regulation of immature T cell proliferation in thymus / ERBB3:ERBB2 complex / ERBB2-ERBB4 signaling pathway / GRB7 events in ERBB2 signaling / immature T cell proliferation in thymus / RNA polymerase I core binding / semaphorin receptor complex / regulation of microtubule-based process / ErbB-3 class receptor binding / Sema4D induced cell migration and growth-cone collapse / motor neuron axon guidance / neurotransmitter receptor localization to postsynaptic specialization membrane / PLCG1 events in ERBB2 signaling / ERBB2-EGFR signaling pathway / positive regulation of Rho protein signal transduction / ERBB2 Activates PTK6 Signaling / neuromuscular junction development / Drug-mediated inhibition of ERBB2 signaling / Resistance of ERBB2 KD mutants to trastuzumab / Resistance of ERBB2 KD mutants to sapitinib / Resistance of ERBB2 KD mutants to tesevatinib / Resistance of ERBB2 KD mutants to neratinib / Resistance of ERBB2 KD mutants to osimertinib / Resistance of ERBB2 KD mutants to afatinib / Resistance of ERBB2 KD mutants to AEE788 / Resistance of ERBB2 KD mutants to lapatinib /  Drug resistance in ERBB2 TMD/JMD mutants / Drug resistance in ERBB2 TMD/JMD mutants /  enzyme-linked receptor protein signaling pathway / ERBB2-ERBB3 signaling pathway / positive regulation of transcription by RNA polymerase I / oligodendrocyte differentiation / ERBB2 Regulates Cell Motility / semaphorin-plexin signaling pathway / PI3K events in ERBB2 signaling / positive regulation of protein targeting to membrane / positive regulation of cell adhesion / enzyme-linked receptor protein signaling pathway / ERBB2-ERBB3 signaling pathway / positive regulation of transcription by RNA polymerase I / oligodendrocyte differentiation / ERBB2 Regulates Cell Motility / semaphorin-plexin signaling pathway / PI3K events in ERBB2 signaling / positive regulation of protein targeting to membrane / positive regulation of cell adhesion /  regulation of angiogenesis / regulation of angiogenesis /  coreceptor activity / Schwann cell development / Signaling by ERBB2 / cellular response to epidermal growth factor stimulus / coreceptor activity / Schwann cell development / Signaling by ERBB2 / cellular response to epidermal growth factor stimulus /  髄鞘 / TFAP2 (AP-2) family regulates transcription of growth factors and their receptors / GRB2 events in ERBB2 signaling / Downregulation of ERBB2:ERBB3 signaling / 髄鞘 / TFAP2 (AP-2) family regulates transcription of growth factors and their receptors / GRB2 events in ERBB2 signaling / Downregulation of ERBB2:ERBB3 signaling /  transmembrane receptor protein tyrosine kinase activity / transmembrane receptor protein tyrosine kinase activity /  神経発生 / SHC1 events in ERBB2 signaling / Constitutive Signaling by Overexpressed ERBB2 / regulation of ERK1 and ERK2 cascade / basal plasma membrane / positive regulation of translation / phosphatidylinositol 3-kinase/protein kinase B signal transduction / positive regulation of epithelial cell proliferation / Signaling by ERBB2 TMD/JMD mutants / 神経発生 / SHC1 events in ERBB2 signaling / Constitutive Signaling by Overexpressed ERBB2 / regulation of ERK1 and ERK2 cascade / basal plasma membrane / positive regulation of translation / phosphatidylinositol 3-kinase/protein kinase B signal transduction / positive regulation of epithelial cell proliferation / Signaling by ERBB2 TMD/JMD mutants /  neuromuscular junction / positive regulation of MAP kinase activity / neuromuscular junction / positive regulation of MAP kinase activity /  wound healing / Signaling by ERBB2 ECD mutants / Signaling by ERBB2 KD Mutants / wound healing / Signaling by ERBB2 ECD mutants / Signaling by ERBB2 KD Mutants /  受容体型チロシンキナーゼ / neuron differentiation / 受容体型チロシンキナーゼ / neuron differentiation /  receptor tyrosine kinase binding / cellular response to growth factor stimulus / Downregulation of ERBB2 signaling / ruffle membrane / peptidyl-tyrosine phosphorylation / receptor tyrosine kinase binding / cellular response to growth factor stimulus / Downregulation of ERBB2 signaling / ruffle membrane / peptidyl-tyrosine phosphorylation /  cell surface receptor protein tyrosine kinase signaling pathway / Constitutive Signaling by Aberrant PI3K in Cancer / transmembrane signaling receptor activity / PIP3 activates AKT signaling / cell surface receptor protein tyrosine kinase signaling pathway / Constitutive Signaling by Aberrant PI3K in Cancer / transmembrane signaling receptor activity / PIP3 activates AKT signaling /  presynaptic membrane / presynaptic membrane /  髄鞘 / 髄鞘 /  heart development / PI5P, PP2A and IER3 Regulate PI3K/AKT Signaling / RAF/MAP kinase cascade / positive regulation of cell growth / basolateral plasma membrane / heart development / PI5P, PP2A and IER3 Regulate PI3K/AKT Signaling / RAF/MAP kinase cascade / positive regulation of cell growth / basolateral plasma membrane /  protein tyrosine kinase activity / positive regulation of MAPK cascade / cell surface receptor signaling pathway / protein tyrosine kinase activity / positive regulation of MAPK cascade / cell surface receptor signaling pathway /  receptor complex / receptor complex /  エンドソーム / endosome membrane / intracellular signal transduction / positive regulation of protein phosphorylation / apical plasma membrane / protein heterodimerization activity / エンドソーム / endosome membrane / intracellular signal transduction / positive regulation of protein phosphorylation / apical plasma membrane / protein heterodimerization activity /  protein phosphorylation / protein phosphorylation /  signaling receptor binding / positive regulation of cell population proliferation / negative regulation of apoptotic process / perinuclear region of cytoplasm / signaling receptor binding / positive regulation of cell population proliferation / negative regulation of apoptotic process / perinuclear region of cytoplasm /  シグナル伝達 / シグナル伝達 /  核質 / 核質 /  ATP binding / ATP binding /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞核 / 細胞核 /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
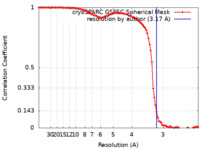
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.17 Å クライオ電子顕微鏡法 / 解像度: 3.17 Å | |||||||||
 データ登録者 データ登録者 | Ruedas R / Vuillemot R / Tubiana T / Winter JM / Pieri L / Arteni AA / Samson C / Jonic J / Mathieu M / Bressanelli S | |||||||||
| 資金援助 |  フランス, 2件 フランス, 2件
| |||||||||
 引用 引用 |  ジャーナル: J Struct Biol / 年: 2024 ジャーナル: J Struct Biol / 年: 2024タイトル: Structure and conformational variability of the HER2-trastuzumab-pertuzumab complex. 著者: Rémi Ruedas / Rémi Vuillemot / Thibault Tubiana / Jean-Marie Winter / Laura Pieri / Ana-Andreea Arteni / Camille Samson / Slavica Jonic / Magali Mathieu / Stéphane Bressanelli /  要旨: Single particle analysis from cryogenic transmission electron microscopy (cryo-EM) is particularly attractive for complexes for which structure prediction remains intractable, such as antibody- ...Single particle analysis from cryogenic transmission electron microscopy (cryo-EM) is particularly attractive for complexes for which structure prediction remains intractable, such as antibody-antigen complexes. Here we obtain the detailed structure of a particularly difficult complex between human epidermal growth factor receptor 2 (HER2) and the antigen-binding fragments from two distinct therapeutic antibodies binding to distant parts of the flexible HER2, pertuzumab and trastuzumab (HTP). We highlight the strengths and limitations of current data processing software in dealing with various kinds of heterogeneities, particularly continuous conformational heterogeneity, and in describing the motions that can be extracted from our dataset. Our HTP structure provides a more detailed view than the one previously available for this ternary complex. This allowed us to pinpoint a previously overlooked loop in domain IV that may be involved both in binding of trastuzumab and in HER2 dimerization. This finding may contribute to explain the synergistic anticancer effect of the two antibodies. We further propose that the flexibility of the HTP complex, beyond the difficulties it causes for cryo-EM analysis, actually reflects regulation of HER2 signaling and its inhibition by therapeutic antibodies. Notably we obtain our best data with ultra-thin continuous carbon grids, showing that with current cameras their use to alleviate particle misdistribution is compatible with a protein complex of only 162 kDa. Perhaps most importantly, we provide here a dataset for such a smallish protein complex for further development of software accounting for continuous conformational heterogeneity in cryo-EM images. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17993.map.gz emd_17993.map.gz | 40 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17993-v30.xml emd-17993-v30.xml emd-17993.xml emd-17993.xml | 21.6 KB 21.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17993_fsc.xml emd_17993_fsc.xml | 10.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17993.png emd_17993.png | 89.2 KB | ||
| Filedesc metadata |  emd-17993.cif.gz emd-17993.cif.gz | 7.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17993 http://ftp.pdbj.org/pub/emdb/structures/EMD-17993 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17993 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17993 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17993.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17993.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Composite map visible in figure 1 d | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.16 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of her2-trastuzumab-pertuzumab Fabs
| 全体 | 名称: Ternary complex of her2-trastuzumab-pertuzumab Fabs |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of her2-trastuzumab-pertuzumab Fabs
| 超分子 | 名称: Ternary complex of her2-trastuzumab-pertuzumab Fabs / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 詳細: Fab fragment from trastuzumab and pertuzumab linked to the extracellular domain of erbb2 (her2) |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 171.364 KDa |
-分子 #1: Trastuzumab Fab light chain
| 分子 | 名称: Trastuzumab Fab light chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.466031 KDa |
| 組換発現 | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ) |
| 配列 | 文字列: DIQMTQSPSS LSASVGDRVT ITCRASQDVN TAVAWYQQKP GKAPKLLIYS ASFLYSGVPS RFSGSRSGTD FTLTISSLQP EDFATYYCQ QHYTTPPTFG QGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS ...文字列: DIQMTQSPSS LSASVGDRVT ITCRASQDVN TAVAWYQQKP GKAPKLLIYS ASFLYSGVPS RFSGSRSGTD FTLTISSLQP EDFATYYCQ QHYTTPPTFG QGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS KDSTYSLSST LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRGEC |
-分子 #2: Trastuzumab Fab heavy chain
| 分子 | 名称: Trastuzumab Fab heavy chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.42518 KDa |
| 組換発現 | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ) |
| 配列 | 文字列: EVQLVESGGG LVQPGGSLRL SCAASGFNIK DTYIHWVRQA PGKGLEWVAR IYPTNGYTRY ADSVKGRFTI SADTSKNTAY LQMNSLRAE DTAVYYCSRW GGDGFYAMDY WGQGTLVTVS SASTKGPSVF PLAPSSKSTS GGTAALGCLV KDYFPEPVTV S WNSGALTS ...文字列: EVQLVESGGG LVQPGGSLRL SCAASGFNIK DTYIHWVRQA PGKGLEWVAR IYPTNGYTRY ADSVKGRFTI SADTSKNTAY LQMNSLRAE DTAVYYCSRW GGDGFYAMDY WGQGTLVTVS SASTKGPSVF PLAPSSKSTS GGTAALGCLV KDYFPEPVTV S WNSGALTS GVHTFPAVLQ SSGLYSLSSV VTVPSSSLGT QTYICNVNHK PSNTKVDKKV EP |
-分子 #3: Pertuzumab Fab light chain
| 分子 | 名称: Pertuzumab Fab light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.548152 KDa |
| 組換発現 | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ) |
| 配列 | 文字列: DIQMTQSPSS LSASVGDRVT ITCKASQDVS IGVAWYQQKP GKAPKLLIYS ASYRYTGVPS RFSGSGSGTD FTLTISSLQP EDFATYYCQ QYYIYPYTFG QGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS ...文字列: DIQMTQSPSS LSASVGDRVT ITCKASQDVS IGVAWYQQKP GKAPKLLIYS ASYRYTGVPS RFSGSGSGTD FTLTISSLQP EDFATYYCQ QYYIYPYTFG QGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS KDSTYSLSST LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRGEC |
-分子 #4: Pertuzumab Fab heavy chain
| 分子 | 名称: Pertuzumab Fab heavy chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.674486 KDa |
| 組換発現 | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ) |
| 配列 | 文字列: EVQLVESGGG LVQPGGSLRL SCAASGFTFT DYTMDWVRQA PGKGLEWVAD VNPNSGGSIY NQRFKGRFTL SVDRSKNTLY LQMNSLRAE DTAVYYCARN LGPSFYFDYW GQGTLVTVSS ASTKGPSVFP LAPSSKSTSG GTAALGCLVK DYFPEPVTVS W NSGALTSG ...文字列: EVQLVESGGG LVQPGGSLRL SCAASGFTFT DYTMDWVRQA PGKGLEWVAD VNPNSGGSIY NQRFKGRFTL SVDRSKNTLY LQMNSLRAE DTAVYYCARN LGPSFYFDYW GQGTLVTVSS ASTKGPSVFP LAPSSKSTSG GTAALGCLVK DYFPEPVTVS W NSGALTSG VHTFPAVLQS SGLYSLSSVV TVPSSSLGTQ TYICNVNHKP SNTKVDKKVE PKSC |
-分子 #5: Receptor tyrosine-protein kinase erbB-2
| 分子 | 名称: Receptor tyrosine-protein kinase erbB-2 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 68.794086 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: TQVCTGTDMK LRLPASPETH LDMLRHLYQG CQVVQGNLEL TYLPTNASLS FLQDIQEVQG YVLIAHNQVR QVPLQRLRIV RGTQLFEDN YALAVLDNGD PLNNTTPVTG ASPGGLRELQ LRSLTEILKG GVLIQRNPQL CYQDTILWKD IFHKNNQLAL T LIDTNRSR ...文字列: TQVCTGTDMK LRLPASPETH LDMLRHLYQG CQVVQGNLEL TYLPTNASLS FLQDIQEVQG YVLIAHNQVR QVPLQRLRIV RGTQLFEDN YALAVLDNGD PLNNTTPVTG ASPGGLRELQ LRSLTEILKG GVLIQRNPQL CYQDTILWKD IFHKNNQLAL T LIDTNRSR ACHPCSPMCK GSRCWGESSE DCQSLTRTVC AGGCARCKGP LPTDCCHEQC AAGCTGPKHS DCLACLHFNH SG ICELHCP ALVTYNTDTF ESMPNPEGRY TFGASCVTAC PYNYLSTDVG SCTLVCPLHN QEVTAEDGTQ RCEKCSKPCA RVC YGLGME HLREVRAVTS ANIQEFAGCK KIFGSLAFLP ESFDGDPASN TAPLQPEQLQ VFETLEEITG YLYISAWPDS LPDL SVFQN LQVIRGRILH NGAYSLTLQG LGISWLGLRS LRELGSGLAL IHHNTHLCFV HTVPWDQLFR NPHQALLHTA NRPED ECVG EGLACHQLCA RGHCWGPGPT QCVNCSQFLR GQECVEECRV LQGLPREYVN ARHCLPCHPE CQPQNGSVTC FGPEAD QCV ACAHYKDPPF CVARCPSGVK PDLSYMPIWK FPDEEGACQP CPINCTHSCV DLDDKGCPAE Q UniProtKB: Receptor tyrosine-protein kinase erbB-2 |
-分子 #7: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 7 / コピー数: 3 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.12 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 2 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: AIR / 詳細: 12 mAu | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 288.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.1 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 240000 Bright-field microscopy / Cs: 2.1 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 240000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: HELIUM |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 8928 / 平均露光時間: 4.71 sec. / 平均電子線量: 60.0 e/Å2 |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT | ||||||||||||
| 得られたモデル |  PDB-8pwh: |
 ムービー
ムービー コントローラー
コントローラー