+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Pyrococcus abyssi DNA polymerase D (PolD) in its editing mode bound to a primer/template substrate containing three consecutive mismatches | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Polymerase (ポリメラーゼ) / Polymerase (ポリメラーゼ) /  DNA (デオキシリボ核酸) / DNA (デオキシリボ核酸) /  Replication (DNA複製) / PolD / Replication (DNA複製) / PolD /  Archaea (古細菌) / Archaea (古細菌) /  Editing (編集) / Editing (編集) /  Proofreading (校正) / Proofreading (校正) /  Exonuclease (エキソヌクレアーゼ) / Exonuclease (エキソヌクレアーゼ) /  Nuclease (ヌクレアーゼ) / Nuclease (ヌクレアーゼ) /  DNA BINDING PROTEIN (DNA結合タンパク質) DNA BINDING PROTEIN (DNA結合タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 エキソデオキシリボヌクレアーゼI / single-stranded DNA 3'-5' DNA exonuclease activity / DNA catabolic process / DNA polymerase processivity factor activity / エキソデオキシリボヌクレアーゼI / single-stranded DNA 3'-5' DNA exonuclease activity / DNA catabolic process / DNA polymerase processivity factor activity /  regulation of DNA replication / DNA-templated DNA replication / regulation of DNA replication / DNA-templated DNA replication /  DNA複製 / DNA複製 /  DNAポリメラーゼ / DNAポリメラーゼ /  DNA-directed DNA polymerase activity / DNA-directed DNA polymerase activity /  DNA binding DNA binding類似検索 - 分子機能 | |||||||||
| 生物種 |    Pyrococcus abyssi GE5 (古細菌) Pyrococcus abyssi GE5 (古細菌) | |||||||||
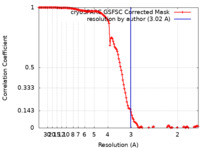
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.02 Å クライオ電子顕微鏡法 / 解像度: 3.02 Å | |||||||||
 データ登録者 データ登録者 | Betancurt-Anzola L / Martinez-Carranza M / Zatopek KM / Gardner AF / Sauguet L | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Molecular basis for proofreading by the unique exonuclease domain of Family-D DNA polymerases. 著者: Leonardo Betancurt-Anzola / Markel Martínez-Carranza / Marc Delarue / Kelly M Zatopek / Andrew F Gardner / Ludovic Sauguet /   要旨: Replicative DNA polymerases duplicate entire genomes at high fidelity. This feature is shared among the three domains of life and is facilitated by their dual polymerase and exonuclease activities. ...Replicative DNA polymerases duplicate entire genomes at high fidelity. This feature is shared among the three domains of life and is facilitated by their dual polymerase and exonuclease activities. Family D replicative DNA polymerases (PolD), found exclusively in Archaea, contain an unusual RNA polymerase-like catalytic core, and a unique Mre11-like proofreading active site. Here, we present cryo-EM structures of PolD trapped in a proofreading mode, revealing an unanticipated correction mechanism that extends the repertoire of protein domains known to be involved in DNA proofreading. Based on our experimental structures, mutants of PolD were designed and their contribution to mismatch bypass and exonuclease kinetics was determined. This study sheds light on the convergent evolution of structurally distinct families of DNA polymerases, and the domain acquisition and exchange mechanism that occurred during the evolution of the replisome in the three domains of life. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17816.map.gz emd_17816.map.gz | 230.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17816-v30.xml emd-17816-v30.xml emd-17816.xml emd-17816.xml | 20 KB 20 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17816_fsc.xml emd_17816_fsc.xml | 13.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17816.png emd_17816.png | 154 KB | ||
| マスクデータ |  emd_17816_msk_1.map emd_17816_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17816.cif.gz emd-17816.cif.gz | 7.5 KB | ||
| その他 |  emd_17816_half_map_1.map.gz emd_17816_half_map_1.map.gz emd_17816_half_map_2.map.gz emd_17816_half_map_2.map.gz | 226.9 MB 226.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17816 http://ftp.pdbj.org/pub/emdb/structures/EMD-17816 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17816 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17816 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17816.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17816.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17816_msk_1.map emd_17816_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
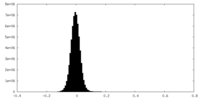
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_17816_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
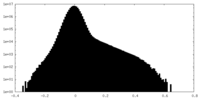
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_17816_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
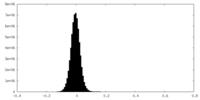
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Binary complex of PolD bound to PCNA and a primer/template duplex...
| 全体 | 名称: Binary complex of PolD bound to PCNA and a primer/template duplex containing three consecutive mismatches |
|---|---|
| 要素 |
|
-超分子 #1: Binary complex of PolD bound to PCNA and a primer/template duplex...
| 超分子 | 名称: Binary complex of PolD bound to PCNA and a primer/template duplex containing three consecutive mismatches タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #3, #5, #4, #1-#2 |
|---|---|
| 由来(天然) | 生物種:    Pyrococcus abyssi GE5 (古細菌) Pyrococcus abyssi GE5 (古細菌) |
-分子 #1: DNA (5'-D(P*CP*CP*GP*GP*GP*CP*CP*GP*AP*GP*CP*CP*GP*TP*(GS)P*(C7R)...
| 分子 | 名称: DNA (5'-D(P*CP*CP*GP*GP*GP*CP*CP*GP*AP*GP*CP*CP*GP*TP*(GS)P*(C7R)P*(PST)P*(PST)P*(PST))-3') タイプ: dna / ID: 1 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:    Pyrococcus abyssi GE5 (古細菌) Pyrococcus abyssi GE5 (古細菌) |
| 分子量 | 理論値: 6.512451 KDa |
| 配列 | 文字列: (DC)(DG)(DC)(DC)(DG)(DG)(DG)(DC)(DC)(DG) (DA)(DG)(DC)(DC)(DG)(DT)(GS)(C7R)(PST) (PST)(PST) |
-分子 #2: DNA (5'-D(P*AP*GP*CP*AP*CP*GP*GP*CP*TP*CP*GP*GP*CP*CP*CP*GP*G)-3')
| 分子 | 名称: DNA (5'-D(P*AP*GP*CP*AP*CP*GP*GP*CP*TP*CP*GP*GP*CP*CP*CP*GP*G)-3') タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:    Pyrococcus abyssi GE5 (古細菌) Pyrococcus abyssi GE5 (古細菌) |
| 分子量 | 理論値: 7.717934 KDa |
| 配列 | 文字列: (DA)(DG)(DG)(DT)(DC)(DG)(DT)(DG)(DC)(DA) (DC)(DG)(DG)(DC)(DT)(DC)(DG)(DG)(DC)(DC) (DC)(DG)(DG)(DC)(DG) |
-分子 #3: DNA polymerase II small subunit
| 分子 | 名称: DNA polymerase II small subunit / タイプ: protein_or_peptide / ID: 3 / 詳細: DP1 subunit / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Pyrococcus abyssi GE5 (古細菌) Pyrococcus abyssi GE5 (古細菌) |
| 分子量 | 理論値: 74.009742 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MGKHHHHSGH HHTGHHHHSG SHHHTSSSAS TGENLYFQGT GDGSDELVKA LERAGYLLTP SAYYLLVDHF KEGKFSLVEL VKFAKSKGV FIIDGDLAYE FLQFLGLGVP QEIKESYIST GEEAEKTVES QETRASELEE GGVSQVSSGE LQELKEESPE I STTEEEIG ...文字列: MGKHHHHSGH HHTGHHHHSG SHHHTSSSAS TGENLYFQGT GDGSDELVKA LERAGYLLTP SAYYLLVDHF KEGKFSLVEL VKFAKSKGV FIIDGDLAYE FLQFLGLGVP QEIKESYIST GEEAEKTVES QETRASELEE GGVSQVSSGE LQELKEESPE I STTEEEIG GLELVQSSIS TGSEVEYNNG ENGESVVVLD KYGYPILYAP EEIGEEKEYS KYEDVVIEWN PSVTPVQIEK NY EVKFDVR QVKLRPPKVK NGSGKEGEII VEAYASLFKS RLSKLKRILR ENPEISNVVD IGKLNYVSGD EEVTIIGLVN SKR ETNRGL IFEVEDKTGI VKVFLPKDSE DYREAFKVLP DAVVAFKGFY SKKGIFFANK FYLPDVPLYR KQKPPLEEKV YAIL ISDIH VGSREFCEKA FLKFLEWLNG HVESKEEEEI VSRVKYLIIA GDVVDGIGIY PGQYSDLVIP DIFDQYEALA NLLAN VPEH ITMFIGPGNA DAARPAIPQP EFYKEYAKPI YKLKNAIIIS NPAVIRLHGR DFLIAHGRGI EDVVSFVPGL THHKPG LPM VELLKMRHLA PTFGGKVPIA PDPEDLLVIE EVPDLVQMGH VHVYDAVVYR GVQLVNSATW QAQTEFQKMV NIVPTPA KV PVVDVESARV VKVLDFSGWC UniProtKB:  DNA polymerase II small subunit DNA polymerase II small subunit |
-分子 #4: DNA polymerase sliding clamp
| 分子 | 名称: DNA polymerase sliding clamp / タイプ: protein_or_peptide / ID: 4 / 詳細: PCNA / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Pyrococcus abyssi GE5 (古細菌) Pyrococcus abyssi GE5 (古細菌) |
| 分子量 | 理論値: 29.471764 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MRGSHHHHHH GSMPFEIVFE GAKEFAQLIE TASRLIDEAA FKVTEEGISM RAMDPSRVVL IDLNLPASIF SKYEVDGEET IGVNMDHLK KVLKRGKAKE TLILRKGEEN FLEISLQGTA TRTFKLPLID VEEIEVDLPE LPFTAKVVIL GDVIKEAVKD A SLVSDSMK ...文字列: MRGSHHHHHH GSMPFEIVFE GAKEFAQLIE TASRLIDEAA FKVTEEGISM RAMDPSRVVL IDLNLPASIF SKYEVDGEET IGVNMDHLK KVLKRGKAKE TLILRKGEEN FLEISLQGTA TRTFKLPLID VEEIEVDLPE LPFTAKVVIL GDVIKEAVKD A SLVSDSMK FIAKENEFTM RAEGETQEVE VKLTLEDEGL LDIEVQEETK SAYGISYLSD MVKGLGKADE VTIKFGNEMP MQ MEYYIRD EGRLIFLLAP RVEE UniProtKB:  DNAクランプ DNAクランプ |
-分子 #5: DP2
| 分子 | 名称: DP2 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Pyrococcus abyssi GE5 (古細菌) Pyrococcus abyssi GE5 (古細菌) |
| 分子量 | 理論値: 144.418969 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MELPKEMEEY FEMLQREIDK AYEIAKKARA QGKDPSLDVE IPQATDMAGR VESLVGPPGV AKRIRELVKE YGKEIAALKI VDEIIEGKF GDLGSREKYA EQAVRTALAI LTEGIVSAPI EGIANVKIKR NTWADNSEYL ALYYAGPIRS SGGTAQALSV L VGDYVRRK ...文字列: MELPKEMEEY FEMLQREIDK AYEIAKKARA QGKDPSLDVE IPQATDMAGR VESLVGPPGV AKRIRELVKE YGKEIAALKI VDEIIEGKF GDLGSREKYA EQAVRTALAI LTEGIVSAPI EGIANVKIKR NTWADNSEYL ALYYAGPIRS SGGTAQALSV L VGDYVRRK LGLDRFKPSE KHIERMVEEV DLYHRAVTRL QYHPSPEEVR LAMRNIPIEI TGEATDDVEV SHRDVPGVET NQ LRGGAIL VLAEGVLQKA KKLVKYIDKM GIEGWEWLKE FVEAKEKGEP KEEGKEESLA ESTLEETKVE VDMGFYYSLY QKF KEEIAP SDKYAKEVIG GRPLFSDPSK PGGFRLRYGR SRASGFATWG INPATMILVD EFLAIGTQLK TERPGKGAVV TPVT TIEGP IVKLKDGSVL RVDDYNLALK VREDVEEILY LGDAVIAFGD FVENNQTLLP ANYCEEWWIL EFVKALKEIY EVHLE PFTE NEEESIEEAS DYLEIDPEFL KEMLRDPLRV KPPVELAIHF SEVLGIPLHP YYTLYWNSVE PKDVEKLWRL LKNYAE IEW SNFRGIKFAK KIVISQEKLG DSKRTLELLG LPHTVRDGNV IVDYPWAAAL LTPLGNLNWE FMAKPLYATI DIINENN EI KLRDRGISWI GARMGRPEKA KERKMKPPVQ VLFPIGLAGG SSRDIKKAAE EGKVAEVEIA FFKCPKCGHV GPEHLCPN C GTRKELLWVC PRCNAEYPES QAEGYNYTCP KCNVKLRPYA KRKIRPSELL NRAMENVKVY GVDKLKGVMG MTSGWKMPE PLEKGLLRAK NDVYVFKDGT IRFDATDAPI THFRPREIGV SVEKLRELGY THDFEGKPLV SEDQIVELKP QDIILSKEAG RYLLKVAKF VDDLLEKFYG LPRFYNAEKM EDLIGHLVIG LAPHTSAGIV GRIIGFVDAL VGYAHPYFHA AKRRNCDGDE D AVMLLLDA LLNFSRYYLP EKRGGKMDAP LVITTRLDPR EVDSEVHNMD IVRYYPLEFY EATYELKSPK ELVGVIERVE DR LGKPEMY YGLKFTHDTD DIALGPKMSL YKQLGDMEEK VRRQLEVAKR IRAVDEHGVA EKILNSHLIP DLRGNLRSFT RQE FRCVKC NTKFRRPPLN GKCPVCGGKI VLTVSKGAIE KYLGTAKMLV TEYNVKNYTR QRICLTERDI DSLFENVFPE TQLT LIVNP NDICQRLVMA RTGEVNKSGL LENLSNGSKK TEKAEKAEKP RKKSDEKPKK KRVISLEEFF SRKSK |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 3 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #7: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 7 / コピー数: 3 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)