+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
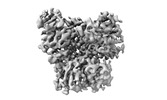
| タイトル | Cryo-EM structure of the RAF activating complex KSR-MEK-CNK-HYP | ||||||||||||
 マップデータ マップデータ | Final Map | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Kinase suppressor of Ras (KSR) / Dual specificity mitogen-activated protein kinase kinase (MEK) / Connector enhancer of KSR (CNK) / Protein Aveugle (AVE) / Hyphen protein (HYP) /  protein complex (タンパク質複合体) / protein complex (タンパク質複合体) /  SIGNALING PROTEIN SIGNALING PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報hemocyte differentiation / MAPK3 (ERK1) activation / MAPK1 (ERK2) activation / Frs2-mediated activation / Negative feedback regulation of MAPK pathway / terminal region determination / Signal transduction by L1 / RAF activation / Phosphorylation of CI / compound eye cone cell differentiation ...hemocyte differentiation / MAPK3 (ERK1) activation / MAPK1 (ERK2) activation / Frs2-mediated activation / Negative feedback regulation of MAPK pathway / terminal region determination / Signal transduction by L1 / RAF activation / Phosphorylation of CI / compound eye cone cell differentiation / Phosphorylation of SMO / terminal branching, open tracheal system / torso signaling pathway / photoreceptor cell development / tracheal outgrowth, open tracheal system / R7 cell fate commitment / compound eye photoreceptor cell differentiation / sevenless signaling pathway / Phosphorylation of PER and TIM / MAP2K and MAPK activation / eye photoreceptor cell differentiation / epithelial cell migration, open tracheal system / imaginal disc-derived wing vein specification / border follicle cell migration / imaginal disc-derived wing morphogenesis / anterior/posterior axis specification, embryo / cellular response to X-ray /  MAPキナーゼキナーゼ / MAP-kinase scaffold activity / mitogen-activated protein kinase kinase kinase binding / mitotic DNA replication checkpoint signaling / dorsal/ventral pattern formation / positive regulation of Ras protein signal transduction / mitotic G2 DNA damage checkpoint signaling / MAPキナーゼキナーゼ / MAP-kinase scaffold activity / mitogen-activated protein kinase kinase kinase binding / mitotic DNA replication checkpoint signaling / dorsal/ventral pattern formation / positive regulation of Ras protein signal transduction / mitotic G2 DNA damage checkpoint signaling /  MAP kinase kinase activity / fibroblast growth factor receptor signaling pathway / enzyme regulator activity / vascular endothelial growth factor receptor signaling pathway / condensed chromosome / ERK1 and ERK2 cascade / MAP kinase kinase activity / fibroblast growth factor receptor signaling pathway / enzyme regulator activity / vascular endothelial growth factor receptor signaling pathway / condensed chromosome / ERK1 and ERK2 cascade /  視覚 / determination of adult lifespan / epidermal growth factor receptor signaling pathway / cytoplasmic side of plasma membrane / 視覚 / determination of adult lifespan / epidermal growth factor receptor signaling pathway / cytoplasmic side of plasma membrane /  receptor tyrosine kinase binding / receptor tyrosine kinase binding /  kinase binding / kinase binding /  cell surface receptor protein tyrosine kinase signaling pathway / cell surface receptor protein tyrosine kinase signaling pathway /  分裂促進因子活性化タンパク質キナーゼ / apical part of cell / insulin receptor signaling pathway / 分裂促進因子活性化タンパク質キナーゼ / apical part of cell / insulin receptor signaling pathway /  細胞皮質 / 細胞皮質 /  scaffold protein binding / scaffold protein binding /  protein tyrosine kinase activity / defense response to virus / Ras protein signal transduction / positive regulation of ERK1 and ERK2 cascade / protein tyrosine kinase activity / defense response to virus / Ras protein signal transduction / positive regulation of ERK1 and ERK2 cascade /  non-specific serine/threonine protein kinase / non-specific serine/threonine protein kinase /  protein kinase activity / protein kinase activity /  protein phosphorylation / protein serine kinase activity / protein serine/threonine kinase activity / protein phosphorylation / protein serine kinase activity / protein serine/threonine kinase activity /  ATP binding / ATP binding /  metal ion binding / metal ion binding /  細胞膜 / 細胞膜 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ) | ||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.32 Å クライオ電子顕微鏡法 / 解像度: 3.32 Å | ||||||||||||
 データ登録者 データ登録者 | Maisonneuve P / Fronzes R / Sicheri F | ||||||||||||
| 資金援助 |  カナダ, カナダ,  フランス, 3件 フランス, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: The CNK-HYP scaffolding complex promotes RAF activation by enhancing KSR-MEK interaction. 著者: Pierre Maisonneuve / Malha Sahmi / Fanny Bergeron-Labrecque / Xianjie Iris Ma / Juliette Queguiner / Geneviève Arseneault / Martin Lefrançois / Igor Kurinov / Rémi Fronzes / Frank Sicheri / Marc Therrien /    要旨: The RAS-MAPK pathway regulates cell proliferation, differentiation and survival, and its dysregulation is associated with cancer development. The pathway minimally comprises the small GTPase RAS and ...The RAS-MAPK pathway regulates cell proliferation, differentiation and survival, and its dysregulation is associated with cancer development. The pathway minimally comprises the small GTPase RAS and the kinases RAF, MEK and ERK. Activation of RAF by RAS is notoriously intricate and remains only partially understood. There are three RAF isoforms in mammals (ARAF, BRAF and CRAF) and two related pseudokinases (KSR1 and KSR2). RAS-mediated activation of RAF depends on an allosteric mechanism driven by the dimerization of its kinase domain. Recent work on human RAFs showed that MEK binding to KSR1 promotes KSR1-BRAF heterodimerization, which leads to the phosphorylation of free MEK molecules by BRAF. Similar findings were made with the single Drosophila RAF homolog. Here we show that the fly scaffold proteins CNK and HYP stabilize the KSR-MEK interaction, which in turn enhances RAF-KSR heterodimerization and RAF activation. The cryogenic electron microscopy structure of the minimal KSR-MEK-CNK-HYP complex reveals a ring-like arrangement of the CNK-HYP complex allowing CNK to simultaneously engage KSR and MEK, thus stabilizing the binary interaction. Together, these results illuminate how CNK contributes to RAF activation by stimulating the allosteric function of KSR and highlight the diversity of mechanisms impacting RAF dimerization as well as the regulatory potential of the KSR-MEK interaction. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16281.map.gz emd_16281.map.gz | 167.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16281-v30.xml emd-16281-v30.xml emd-16281.xml emd-16281.xml | 22.6 KB 22.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_16281.png emd_16281.png | 51.3 KB | ||
| マスクデータ |  emd_16281_msk_1.map emd_16281_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-16281.cif.gz emd-16281.cif.gz | 7.3 KB | ||
| その他 |  emd_16281_half_map_1.map.gz emd_16281_half_map_1.map.gz emd_16281_half_map_2.map.gz emd_16281_half_map_2.map.gz | 165.2 MB 165.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16281 http://ftp.pdbj.org/pub/emdb/structures/EMD-16281 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16281 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16281 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8bw9MC  8bw8C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16281.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16281.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final Map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.93 Å | ||||||||||||||||||||||||||||||||||||
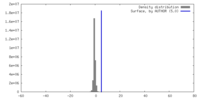
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16281_msk_1.map emd_16281_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
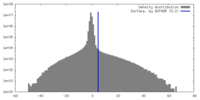
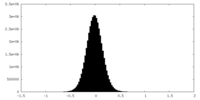
| 密度ヒストグラム |
-ハーフマップ: Half-map B
| ファイル | emd_16281_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
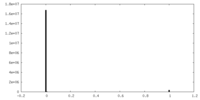
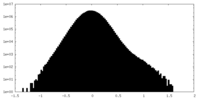
| 密度ヒストグラム |
-ハーフマップ: Half-map A
| ファイル | emd_16281_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
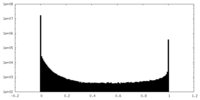
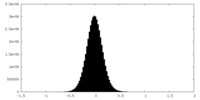
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Quaternary complex of the kinase domains of KSR and MEK bound to ...
| 全体 | 名称: Quaternary complex of the kinase domains of KSR and MEK bound to the scaffolding complex CNK-HYP |
|---|---|
| 要素 |
|
-超分子 #1: Quaternary complex of the kinase domains of KSR and MEK bound to ...
| 超分子 | 名称: Quaternary complex of the kinase domains of KSR and MEK bound to the scaffolding complex CNK-HYP タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 / 詳細: Mg2+/ANP and Trametinib ligands bound to MEK |
|---|---|
| 由来(天然) | 生物種:   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ) |
| 分子量 | 理論値: 132 KDa |
-分子 #1: Protein aveugle
| 分子 | 名称: Protein aveugle / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ) |
| 分子量 | 理論値: 13.143911 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: GAMDPGEETI NSTQNKTRTK TTRPKAVYLW TVSDVLKWYR RHCGEYTQYE QLFAQHDITG RALLRITDSS LQRMGVTDNR DREAIWREI VKQRLKTDIM EIRDMERLNI Y UniProtKB: Protein aveugle |
-分子 #2: Connector enhancer of KSR protein CNK
| 分子 | 名称: Connector enhancer of KSR protein CNK / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ) |
| 分子量 | 理論値: 38.534203 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: GAMDPMAYIN IAEWTPDQVT DWIKGLDESM KGYLYEFSKQ EIGGRALLNI RPYELENLGM LRIGHQEIVL EAVENLRNFH YHLKNDNLQ FMALHVATAA KNLHRELARN HAESTKIDTR ILHDITRTIA TLKPLVGSLE RTPFRKQEMY REYCGNVLKC G LELATIAH ...文字列: GAMDPMAYIN IAEWTPDQVT DWIKGLDESM KGYLYEFSKQ EIGGRALLNI RPYELENLGM LRIGHQEIVL EAVENLRNFH YHLKNDNLQ FMALHVATAA KNLHRELARN HAESTKIDTR ILHDITRTIA TLKPLVGSLE RTPFRKQEMY REYCGNVLKC G LELATIAH RDRFALQPVP AIRQSAERLE NLANFVIQDI SDPMVLQPAS LNLVTLKKRE SELGFNIESS YNGIHRVTDI KY NSPAHNS GKIEDGDEIV QINYQTVVGW QHRTVLEHLR EALPDVVLTV KKRPKHTKMF GQIYMQPYRL PSKKRNMAAR WAA QMPSPR AAFLTLDTE UniProtKB: Connector enhancer of KSR protein CNK |
-分子 #3: Dual specificity mitogen-activated protein kinase kinase dSOR1
| 分子 | 名称: Dual specificity mitogen-activated protein kinase kinase dSOR1 タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号:  MAPキナーゼキナーゼ MAPキナーゼキナーゼ |
|---|---|
| 由来(天然) | 生物種:   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ) |
| 分子量 | 理論値: 43.926496 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MSKNKLNLVL PPVNTEATVA AATVAPTPPF KTPSGTDTHS LLGKPKTSID ALTETLEGLD MGDTERKRIK MFLSQKEKIG ELSDEDLEK LGELGSGNGG VVMKVRHTHT HLIMARKLIH LEVKPAIKKQ ILRELKVLHE CNFPHIVGFY GAFYSDGEIS I CMEYMDGG ...文字列: MSKNKLNLVL PPVNTEATVA AATVAPTPPF KTPSGTDTHS LLGKPKTSID ALTETLEGLD MGDTERKRIK MFLSQKEKIG ELSDEDLEK LGELGSGNGG VVMKVRHTHT HLIMARKLIH LEVKPAIKKQ ILRELKVLHE CNFPHIVGFY GAFYSDGEIS I CMEYMDGG SLDLILKRAG RIPESILGRI TLAVLKGLSY LRDNHAIIHR DVKPSNILVN SSGEIKICDF GVSGQLIDSM AN SFVGTRS YMSPERLQGT HYSVQSDIWS LGLSLVEMAI GMYPIPPPNT ATLESIFADN AEESGQPTDE PRAMAIFELL DYI VNEPPP KLEHKIFSTE FKDFVDICLK KQPDERADLK TLLSHPWIRK AELEEVDISG WVCKTMDLPP STPKRNTSPN UniProtKB: Dual specificity mitogen-activated protein kinase kinase dSOR1 |
-分子 #4: KSR
| 分子 | 名称: KSR / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ) |
| 分子量 | 理論値: 36.474898 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: GGRTEDGDSG QWRQNSISLK EWDIPYGDLL LLERIGQGRF GTVHRALWHG DVAVKLLNED YLQDEHMLET FRSEVANFKN TRHENLVLF MGACMNPPYL AIVTSLCKGN TLYTYIHQRR EKFAMNRTLL IAQQIAQGMG YLHAREIIHK DLRTKNIFIE N GKVIITDF ...文字列: GGRTEDGDSG QWRQNSISLK EWDIPYGDLL LLERIGQGRF GTVHRALWHG DVAVKLLNED YLQDEHMLET FRSEVANFKN TRHENLVLF MGACMNPPYL AIVTSLCKGN TLYTYIHQRR EKFAMNRTLL IAQQIAQGMG YLHAREIIHK DLRTKNIFIE N GKVIITDF GLFSSTKLLY CDMGLGVPHN WLCYLAPELI RALQPEKPRG ECLEFTPYSD VYSFGTVWYE LICGEFTFKD QP AESIIWQ VGRGMKQSLA NLQSGRDVKD LLMLCWTYEK EHRPQFARLL SLLEHLPKKR LARSPSHPVN LSRSAESVF UniProtKB:  non-specific serine/threonine protein kinase non-specific serine/threonine protein kinase |
-分子 #5: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 5 / コピー数: 1 / 式: ANP |
|---|---|
| 分子量 | 理論値: 506.196 Da |
| Chemical component information |  ChemComp-ANP: |
-分子 #6: Trametinib
| 分子 | 名称: Trametinib / タイプ: ligand / ID: 6 / コピー数: 1 / 式: QOM |
|---|---|
| 分子量 | 理論値: 615.395 Da |
| Chemical component information |  ChemComp-QOM: |
-分子 #7: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 7 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.79 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 構成要素:
詳細: AMPPNP 25mM Trametinib 0.05mM | |||||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.00026000000000000003 kPa | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV / 詳細: 3.5 sec blot time 1 sec drain time. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 26.398 µm / 最小 デフォーカス(公称値): 5.212 µm Bright-field microscopy / 最大 デフォーカス(公称値): 26.398 µm / 最小 デフォーカス(公称値): 5.212 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 実像数: 44 / 平均電子線量: 1.15 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.32 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 141531 |
 ムービー
ムービー コントローラー
コントローラー










 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)