+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | BAM-EspP complex structure with BamA-S425C/EspP-S1299C mutations in nanodisc | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Bam protein complex / Gram-negative-bacterium-type cell outer membrane assembly / protein insertion into membrane /  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; セリンエンドペプチターゼ / cell outer membrane / protein-macromolecule adaptor activity / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; セリンエンドペプチターゼ / cell outer membrane / protein-macromolecule adaptor activity /  ペリプラズム / response to antibiotic / serine-type endopeptidase activity / ペリプラズム / response to antibiotic / serine-type endopeptidase activity /  細胞膜 ...Bam protein complex / Gram-negative-bacterium-type cell outer membrane assembly / protein insertion into membrane / 細胞膜 ...Bam protein complex / Gram-negative-bacterium-type cell outer membrane assembly / protein insertion into membrane /  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; セリンエンドペプチターゼ / cell outer membrane / protein-macromolecule adaptor activity / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; セリンエンドペプチターゼ / cell outer membrane / protein-macromolecule adaptor activity /  ペリプラズム / response to antibiotic / serine-type endopeptidase activity / ペリプラズム / response to antibiotic / serine-type endopeptidase activity /  細胞膜 / 細胞膜 /  タンパク質分解 / extracellular region / タンパク質分解 / extracellular region /  生体膜 / identical protein binding 生体膜 / identical protein binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Escherichia coli K-12 (大腸菌) / Escherichia coli K-12 (大腸菌) /   Escherichia coli O157:H7 (大腸菌) Escherichia coli O157:H7 (大腸菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.1 Å クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Shen C / Chang S / Luo Q / Zhang Z / Xie T / Luo B / Lu G / Zhu X / Wei X / Dong C ...Shen C / Chang S / Luo Q / Zhang Z / Xie T / Luo B / Lu G / Zhu X / Wei X / Dong C / Zhou R / Zhang X / Tang X / Dong H | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2023 ジャーナル: Nature / 年: 2023タイトル: Structural basis of BAM-mediated outer membrane β-barrel protein assembly. 著者: Chongrong Shen / Shenghai Chang / Qinghua Luo / Kevin Chun Chan / Zhibo Zhang / Bingnan Luo / Teng Xie / Guangwen Lu / Xiaofeng Zhu / Xiawei Wei / Changjiang Dong / Ruhong Zhou / Xing Zhang / ...著者: Chongrong Shen / Shenghai Chang / Qinghua Luo / Kevin Chun Chan / Zhibo Zhang / Bingnan Luo / Teng Xie / Guangwen Lu / Xiaofeng Zhu / Xiawei Wei / Changjiang Dong / Ruhong Zhou / Xing Zhang / Xiaodi Tang / Haohao Dong /   要旨: The outer membrane structure is common in Gram-negative bacteria, mitochondria and chloroplasts, and contains outer membrane β-barrel proteins (OMPs) that are essential interchange portals of ...The outer membrane structure is common in Gram-negative bacteria, mitochondria and chloroplasts, and contains outer membrane β-barrel proteins (OMPs) that are essential interchange portals of materials. All known OMPs share the antiparallel β-strand topology, implicating a common evolutionary origin and conserved folding mechanism. Models have been proposed for bacterial β-barrel assembly machinery (BAM) to initiate OMP folding; however, mechanisms by which BAM proceeds to complete OMP assembly remain unclear. Here we report intermediate structures of BAM assembling an OMP substrate, EspP, demonstrating sequential conformational dynamics of BAM during the late stages of OMP assembly, which is further supported by molecular dynamics simulations. Mutagenic in vitro and in vivo assembly assays reveal functional residues of BamA and EspP for barrel hybridization, closure and release. Our work provides novel insights into the common mechanism of OMP assembly. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16138.map.gz emd_16138.map.gz | 32.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16138-v30.xml emd-16138-v30.xml emd-16138.xml emd-16138.xml | 24.9 KB 24.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_16138.png emd_16138.png | 106.1 KB | ||
| その他 |  emd_16138_additional_1.map.gz emd_16138_additional_1.map.gz emd_16138_half_map_1.map.gz emd_16138_half_map_1.map.gz emd_16138_half_map_2.map.gz emd_16138_half_map_2.map.gz | 32.3 MB 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16138 http://ftp.pdbj.org/pub/emdb/structures/EMD-16138 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16138 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16138 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16138.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16138.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.93 Å | ||||||||||||||||||||||||||||||||||||
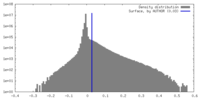
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_16138_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
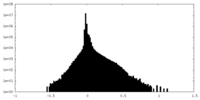
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_16138_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
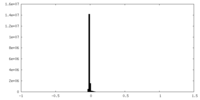
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_16138_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
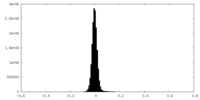
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : BAM-EspP complex with BamA-G431C/EspP-N1293C mutations in nanodisc
| 全体 | 名称: BAM-EspP complex with BamA-G431C/EspP-N1293C mutations in nanodisc |
|---|---|
| 要素 |
|
-超分子 #1: BAM-EspP complex with BamA-G431C/EspP-N1293C mutations in nanodisc
| 超分子 | 名称: BAM-EspP complex with BamA-G431C/EspP-N1293C mutations in nanodisc タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌) |
-超分子 #2: BAM
| 超分子 | 名称: BAM / タイプ: complex / ID: 2 / キメラ: Yes / 親要素: 1 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌) |
-超分子 #3: EspP
| 超分子 | 名称: EspP / タイプ: complex / ID: 3 / キメラ: Yes / 親要素: 1 / 含まれる分子: #6 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli O157:H7 (大腸菌) Escherichia coli O157:H7 (大腸菌) |
-分子 #1: Outer membrane protein assembly factor BamA
| 分子 | 名称: Outer membrane protein assembly factor BamA / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli K-12 (大腸菌) / 株: K12 / DH10B Escherichia coli K-12 (大腸菌) / 株: K12 / DH10B |
| 分子量 | 理論値: 88.273555 KDa |
| 組換発現 | 生物種:   Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) |
| 配列 | 文字列: FVVKDIHFEG LQRVAVGAAL LSMPVRTGDT VNDEDISNTI RALFATGNFE DVRVLRDGDT LLVQVKERPT IASITFSGNK SVKDDMLKQ NLEASGVRVG ESLDRTTIAD IEKGLEDFYY SVGKYSASVK AVVTPLPRNR VDLKLVFQEG VSAEIQQINI V GNHAFTTD ...文字列: FVVKDIHFEG LQRVAVGAAL LSMPVRTGDT VNDEDISNTI RALFATGNFE DVRVLRDGDT LLVQVKERPT IASITFSGNK SVKDDMLKQ NLEASGVRVG ESLDRTTIAD IEKGLEDFYY SVGKYSASVK AVVTPLPRNR VDLKLVFQEG VSAEIQQINI V GNHAFTTD ELISHFQLRD EVPWWNVVGD RKYQKQKLAG DLETLRSYYL DRGYARFNID STQVSLTPDK KGIYVTVNIT EG DQYKLSG VEVSGNLAGH SAEIEQLTKI EPGELYNGTK VTKMEDDIKK LLGRYGYAYP RVQSMPEIND ADKTVKLRVN VDA GNRFYV RKIRFEGNDT SKDAVLRREM RQMEGAWLGS DLVDQGKERL NRLGFFETVD TDTQRVPGSP DQVDVVYKVK ERNT GCFNF GIGYGTESGV SFQAGVQQDN WLGTGYAVGI NGTKNDYQTY AELSVTNPYF TVDGVSLGGR LFYNDFQADD ADLSD YTNK SYGTDVTLGF PINEYNSLRA GLGYVHNSLS NMQPQVAMWR YLYSMGEHPS TSDQDNSFKT DDFTFNYGWT YNKLDR GYF PTDGSRVNLT GKVTIPGSDN EYYKVTLDTA TYVPIDDDHK WVVLGRTRWG YGDGLGGKEM PFYENFYAGG SSTVRGF QS NTIGPKAVYF PHQASNYDPD YDYECATQDG AKDLCKSDDA VGGNAMAVAS LEFITPTPFI SDKYANSVRT SFFWDMGT V WDTNWDSSQY SGYPDYSDPS NIRMSAGIAL QWMSPLGPLV FSYAQPFKKY DGDKAEQFQF NIGKTW |
-分子 #2: Outer membrane protein assembly factor BamB
| 分子 | 名称: Outer membrane protein assembly factor BamB / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌) |
| 分子量 | 理論値: 41.918945 KDa |
| 組換発現 | 生物種:   Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) |
| 配列 | 文字列: MQLRKLLLPG LLSVTLLSGC SLFNSEEDVV KMSPLPTVEN QFTPTTAWST SVGSGIGNFY SNLHPALADN VVYAADRAGL VKALNADDG KEIWSVSLAE KDGWFSKEPA LLSGGVTVSG GHVYIGSEKA QVYALNTSDG TVAWQTKVAG EALSRPVVSD G LVLIHTSN ...文字列: MQLRKLLLPG LLSVTLLSGC SLFNSEEDVV KMSPLPTVEN QFTPTTAWST SVGSGIGNFY SNLHPALADN VVYAADRAGL VKALNADDG KEIWSVSLAE KDGWFSKEPA LLSGGVTVSG GHVYIGSEKA QVYALNTSDG TVAWQTKVAG EALSRPVVSD G LVLIHTSN GQLQALNEAD GAVKWTVNLD MPSLSLRGES APTTAFGAAV VGGDNGRVSA VLMEQGQMIW QQRISQATGS TE IDRLSDV DTTPVVVNGV VFALAYNGNL TALDLRSGQI MWKRELGSVN DFIVDGNRIY LVDQNDRVMA LTIDGGVTLW TQS DLLHRL LTSPVLYNGN LVVGDSEGYL HWINVEDGRF VAQQKVDSSG FQTEPVAADG KLLIQAKDGT VYSITR |
-分子 #3: Outer membrane protein assembly factor BamC
| 分子 | 名称: Outer membrane protein assembly factor BamC / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌) |
| 分子量 | 理論値: 36.875277 KDa |
| 組換発現 | 生物種:   Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) |
| 配列 | 文字列: MAYSVQKSRL AKVAGVSLVL LLAACSSDSR YKRQVSGDEA YLEAAPLAEL HAPAGMILPV TSGDYAIPVT NGSGAVGKAL DIRPPAQPL ALVSGARTQF TGDTASLLVE NGRGNTLWPQ VVSVLQAKNY TITQRDDAGQ TLTTDWVQWN RLDEDEQYRG R YQISVKPQ ...文字列: MAYSVQKSRL AKVAGVSLVL LLAACSSDSR YKRQVSGDEA YLEAAPLAEL HAPAGMILPV TSGDYAIPVT NGSGAVGKAL DIRPPAQPL ALVSGARTQF TGDTASLLVE NGRGNTLWPQ VVSVLQAKNY TITQRDDAGQ TLTTDWVQWN RLDEDEQYRG R YQISVKPQ GYQQAVTVKL LNLEQAGKPV ADAASMQRYS TEMMNVISAG LDKSATDAAN AAQNRASTTM DVQSAADDTG LP MLVVRGP FNVVWQRLPA ALEKVGMKVT DSTRSQGNMA VTYKPLSDSD WQELGASDPG LASGDYKLQV GDLDNRSSLQ FID PKGHTL TQSQNDALVA VFQAAFSK |
-分子 #4: Outer membrane protein assembly factor BamD
| 分子 | 名称: Outer membrane protein assembly factor BamD / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌) |
| 分子量 | 理論値: 27.85835 KDa |
| 組換発現 | 生物種:   Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) |
| 配列 | 文字列: MTRMKYLVAA ATLSLFLAGC SGSKEEVPDN PPNEIYATAQ QKLQDGNWRQ AITQLEALDN RYPFGPYSQQ VQLDLIYAYY KNADLPLAQ AAIDRFIRLN PTHPNIDYVM YMRGLTNMAL DDSALQGFFG VDRSDRDPQH ARAAFSDFSK LVRGYPNSQY T TDATKRLV ...文字列: MTRMKYLVAA ATLSLFLAGC SGSKEEVPDN PPNEIYATAQ QKLQDGNWRQ AITQLEALDN RYPFGPYSQQ VQLDLIYAYY KNADLPLAQ AAIDRFIRLN PTHPNIDYVM YMRGLTNMAL DDSALQGFFG VDRSDRDPQH ARAAFSDFSK LVRGYPNSQY T TDATKRLV FLKDRLAKYE YSVAEYYTER GAWVAVVNRV EGMLRDYPDT QATRDALPLM ENAYRQMQMN AQAEKVAKII AA NSSNT |
-分子 #5: Outer membrane protein assembly factor BamE
| 分子 | 名称: Outer membrane protein assembly factor BamE / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌) |
| 分子量 | 理論値: 13.530256 KDa |
| 組換発現 | 生物種:   Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) |
| 配列 | 文字列: MRCKTLTAAA AVLLMLTAGC STLERVVYRP DINQGNYLTA NDVSKIRVGM TQQQVAYALG TPLMSDPFGT NTWFYVFRQQ PGHEGVTQQ TLTLTFNSSG VLTNIDNKPA LSGNGGHHHH HHHH |
-分子 #6: Serine protease EspP
| 分子 | 名称: Serine protease EspP / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO EC番号:  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; セリンエンドペプチターゼ 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; セリンエンドペプチターゼ |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌) |
| 分子量 | 理論値: 38.754836 KDa |
| 組換発現 | 生物種:   Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) |
| 配列 | 文字列: NIELVSAPKD TNENVFKASK QTIGFSDVTP VITTRETDDK ITWSLTGYNT VANKEATRNA AALFSVDYKA FLNEVNNLNK RMGDLRDIN GEAGAWARIM SGTGSASGGF SDNYTHVQVG VDKKHELDGL DLFTGFTVTH TDSSASADVF SGKTKSVGAG L YASAMFDS ...文字列: NIELVSAPKD TNENVFKASK QTIGFSDVTP VITTRETDDK ITWSLTGYNT VANKEATRNA AALFSVDYKA FLNEVNNLNK RMGDLRDIN GEAGAWARIM SGTGSASGGF SDNYTHVQVG VDKKHELDGL DLFTGFTVTH TDSSASADVF SGKTKSVGAG L YASAMFDS GAYIDLIGKY VHHDNEYTAT FAGLGTRDYS THSWYAGAEA GYRYHVTEDA WIEPQAELVY GSVSGKQFAW KD QGMHLSM KDKDYNPLIG RTGVDVGKSF SGKDWKVTAR AGLGYQFDLL ANGETVLRDA SGEKRIKGEK DSRMLMSVGL NAE IRDNVR FGLEFEKSAF GKYNVDNAVN ANFRYCF |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 2.0 µm / 最小 デフォーカス(補正後): 1.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
|---|---|
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 203494 |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)