+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1503 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Initial location of the RNA-dependent RNA polymerase in the bacteriophage phi6 procapsid determined by cryo-electron microscopy | |||||||||
 マップデータ マップデータ | Map of a procapsid without the P7 protein, containing only proteins P1, P2 and P4 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Cystoviridae / P2 protein Cystoviridae / P2 protein | |||||||||
| 生物種 |   Pseudomonas phage phi6 (ファージ) Pseudomonas phage phi6 (ファージ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / クライオ電子顕微鏡法 /  ネガティブ染色法 / 解像度: 16.0 Å ネガティブ染色法 / 解像度: 16.0 Å | |||||||||
 データ登録者 データ登録者 | Sen A / Heymann JB / Cheng N / Qiao J / Mindich L / Steven AC | |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2008 ジャーナル: J Biol Chem / 年: 2008タイトル: Initial location of the RNA-dependent RNA polymerase in the bacteriophage Phi6 procapsid determined by cryo-electron microscopy. 著者: Anindito Sen / J Bernard Heymann / Naiqian Cheng / Jian Qiao / Leonard Mindich / Alasdair C Steven /  要旨: The RNA-dependent RNA polymerases (RdRPs) of Cystoviridae bacteriophages, like those of eukaryotic viruses of the Reoviridae, function inside the inner capsid shell in both replication and ...The RNA-dependent RNA polymerases (RdRPs) of Cystoviridae bacteriophages, like those of eukaryotic viruses of the Reoviridae, function inside the inner capsid shell in both replication and transcription. In bacteriophage Phi6, this inner shell is first assembled as an icosahedral procapsid with recessed 5-fold vertices that subsequently undergoes major structural changes during maturation. The tripartite genome is packaged as single-stranded RNA molecules via channels on the 5-fold vertices, and transcripts probably exit the mature capsid by the same route. The RdRP (protein P2) is assembled within the procapsid, and it was thought that it should be located on the 5-fold axes near the RNA entry and exit channels. To determine the initial location of the RdRP inside the procapsid of bacteriophage Phi6, we performed cryo-electron microscopy of wild type and mutant procapsids and complemented these data with biochemical determinations of copy numbers. We observe ring-like densities on the 3-fold axes that are strong in a mutant that has approximately 10 copies of P2 per particle; faint in wild type, reflecting the lower copy number of approximately 3; and completely absent in a P2-null mutant. The dimensions and shapes of these densities match those of the known crystal structure of the P2 monomer. We propose that, during maturation, the P2 molecules rotate to occupy positions closer to adjacent 5-fold vertices where they conduct replication and transcription. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1503.map.gz emd_1503.map.gz | 19.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1503-v30.xml emd-1503-v30.xml emd-1503.xml emd-1503.xml | 9.3 KB 9.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1503.gif 1503.gif emd_1503.png emd_1503.png emd_1503.tif emd_1503.tif emd_1503_1.png emd_1503_1.png emd_1503_1.tif emd_1503_1.tif | 84.3 KB 646.5 KB 699 KB 493 KB 520.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1503 http://ftp.pdbj.org/pub/emdb/structures/EMD-1503 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1503 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1503 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1503.map.gz / 形式: CCP4 / 大きさ: 47.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1503.map.gz / 形式: CCP4 / 大きさ: 47.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map of a procapsid without the P7 protein, containing only proteins P1, P2 and P4 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.54 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
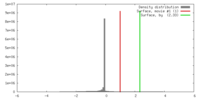
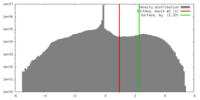
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Phi 6 procapsid without the P7 protein
| 全体 | 名称: Phi 6 procapsid without the P7 protein |
|---|---|
| 要素 |
|
-超分子 #1000: Phi 6 procapsid without the P7 protein
| 超分子 | 名称: Phi 6 procapsid without the P7 protein / タイプ: sample / ID: 1000 詳細: Produced using the Escherichia coli cell line LM1115 with the plasmid, pLM574 集合状態: Procapsid with 3 proteins P1, P2, P4 / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 14 MDa |
-超分子 #1: Pseudomonas phage phi6
| 超分子 | 名称: Pseudomonas phage phi6 / タイプ: virus / ID: 1 / NCBI-ID: 10879 / 生物種: Pseudomonas phage phi6 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: Yes |
|---|---|
| 宿主 | 生物種:   Pseudomonas syringae (シュードモナス・シリンガエ) Pseudomonas syringae (シュードモナス・シリンガエ)別称: BACTERIA(EUBACTERIA) |
| 分子量 | 理論値: 14 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: Procapsid / 直径: 470 Å / T番号(三角分割数): 1 |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法, ネガティブ染色法,  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 10mM sodium phosphate, 1mM MgCl2 |
| 染色 | タイプ: NEGATIVE / 詳細: None |
| 凍結 | 凍結剤: ETHANE / 装置: REICHERT-JUNG PLUNGER 詳細: Vitrification instrument: Reichert-Jung cryo-fixation unit |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM200FEG |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 1.3 µm / 倍率(公称値): 50000 Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 1.3 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Side entry, Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| アライメント法 | Legacy - 非点収差: Corrected at 300000 times magnification |
| 日付 | 2007年3月23日 |
| 撮影 | デジタル化 - スキャナー: NIKON SUPER COOLSCAN 9000 デジタル化 - サンプリング間隔: 6.35 µm / 実像数: 34 / 平均電子線量: 15 e/Å2 / ビット/ピクセル: 16 |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
- 画像解析
画像解析
| CTF補正 | 詳細: Each particle phase flipped |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 16.0 Å / 解像度の算出法: FSC 0.33 CUT-OFF / ソフトウェア - 名称: Bsoft, PFT3DR / 使用した粒子像数: 1238 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 16.0 Å / 解像度の算出法: FSC 0.33 CUT-OFF / ソフトウェア - 名称: Bsoft, PFT3DR / 使用した粒子像数: 1238 |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)