[日本語] English
 万見
万見- EMDB-14388: Structure of Mycobacterium tuberculosis adenylyl cyclase Rv1625c / Cya -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Mycobacterium tuberculosis adenylyl cyclase Rv1625c / Cya | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報receptor guanylyl cyclase signaling pathway /  peptide receptor activity / cGMP biosynthetic process / peptide receptor activity / cGMP biosynthetic process /  guanylate cyclase activity / guanylate cyclase activity /  アデニル酸シクラーゼ / cAMP biosynthetic process / アデニル酸シクラーゼ / cAMP biosynthetic process /  adenylate cyclase activity / manganese ion binding / intracellular signal transduction / magnesium ion binding ...receptor guanylyl cyclase signaling pathway / adenylate cyclase activity / manganese ion binding / intracellular signal transduction / magnesium ion binding ...receptor guanylyl cyclase signaling pathway /  peptide receptor activity / cGMP biosynthetic process / peptide receptor activity / cGMP biosynthetic process /  guanylate cyclase activity / guanylate cyclase activity /  アデニル酸シクラーゼ / cAMP biosynthetic process / アデニル酸シクラーゼ / cAMP biosynthetic process /  adenylate cyclase activity / manganese ion binding / intracellular signal transduction / magnesium ion binding / adenylate cyclase activity / manganese ion binding / intracellular signal transduction / magnesium ion binding /  ATP binding / ATP binding /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Mycobacterium tuberculosis '98-R604 INH-RIF-EM' (結核菌) / Mycobacterium tuberculosis '98-R604 INH-RIF-EM' (結核菌) /   Vicugna pacos (アルパカ) Vicugna pacos (アルパカ) | ||||||||||||
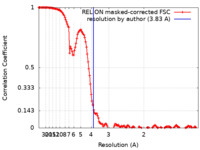
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.83 Å クライオ電子顕微鏡法 / 解像度: 3.83 Å | ||||||||||||
 データ登録者 データ登録者 | Mehta V / Khanppnavar B / Korkhov VM | ||||||||||||
| 資金援助 |  スイス, 3件 スイス, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2022 ジャーナル: Elife / 年: 2022タイトル: Structure of Cya, an evolutionary ancestor of the mammalian membrane adenylyl cyclases. 著者: Ved Mehta / Basavraj Khanppnavar / Dina Schuster / Ilayda Kantarci / Irene Vercellino / Angela Kosturanova / Tarun Iype / Sasa Stefanic / Paola Picotti / Volodymyr M Korkhov /  要旨: adenylyl cyclase (AC) Rv1625c/Cya is an evolutionary ancestor of the mammalian membrane ACs and a model system for studies of their structure and function. Although the vital role of ACs in cellular ... adenylyl cyclase (AC) Rv1625c/Cya is an evolutionary ancestor of the mammalian membrane ACs and a model system for studies of their structure and function. Although the vital role of ACs in cellular signalling is well established, the function of their transmembrane (TM) regions remains unknown. Here, we describe the cryo-EM structure of Cya bound to a stabilizing nanobody at 3.6 Å resolution. The TM helices 1-5 form a structurally conserved domain that facilitates the assembly of the helical and catalytic domains. The TM region contains discrete pockets accessible from the extracellular and cytosolic side of the membrane. Neutralization of the negatively charged extracellular pocket Ex1 destabilizes the cytosolic helical domain and reduces the catalytic activity of the enzyme. The TM domain acts as a functional component of Cya, guiding the assembly of the catalytic domain and providing the means for direct regulation of catalytic activity in response to extracellular ligands. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14388.map.gz emd_14388.map.gz | 189.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14388-v30.xml emd-14388-v30.xml emd-14388.xml emd-14388.xml | 18.3 KB 18.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14388_fsc.xml emd_14388_fsc.xml | 14.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14388.png emd_14388.png | 78.6 KB | ||
| その他 |  emd_14388_half_map_1.map.gz emd_14388_half_map_1.map.gz emd_14388_half_map_2.map.gz emd_14388_half_map_2.map.gz | 193.5 MB 193.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14388 http://ftp.pdbj.org/pub/emdb/structures/EMD-14388 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14388 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14388 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14388.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14388.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.66 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_14388_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_14388_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : A complex of Rv1625c / Cya homodimer bound to nano body Nb4
| 全体 | 名称: A complex of Rv1625c / Cya homodimer bound to nano body Nb4 |
|---|---|
| 要素 |
|
-超分子 #1: A complex of Rv1625c / Cya homodimer bound to nano body Nb4
| 超分子 | 名称: A complex of Rv1625c / Cya homodimer bound to nano body Nb4 タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|
-超分子 #2: Adenylate cyclase
| 超分子 | 名称: Adenylate cyclase / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Mycobacterium tuberculosis '98-R604 INH-RIF-EM' (結核菌) Mycobacterium tuberculosis '98-R604 INH-RIF-EM' (結核菌) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-超分子 #3: Nanobody Nb4
| 超分子 | 名称: Nanobody Nb4 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:   Vicugna pacos (アルパカ) Vicugna pacos (アルパカ) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-分子 #1: Adenylate cyclase
| 分子 | 名称: Adenylate cyclase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号:  アデニル酸シクラーゼ アデニル酸シクラーゼ |
|---|---|
| 由来(天然) | 生物種:   Mycobacterium tuberculosis '98-R604 INH-RIF-EM' (結核菌) Mycobacterium tuberculosis '98-R604 INH-RIF-EM' (結核菌) |
| 分子量 | 理論値: 50.386613 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MGSAWSHPQF EKGSGLEVLF QGPSGHMAAR KCGAPPIAAD GSTRRPDCVT AVRTQARAPT QHYAESVARR QRVLTITAWL AVVVTGSFA LMQLATGAGG WYIALINVFT AVTFAIVPLL HRFGGLVAPL TFIGTAYVAI FAIGWDVGTD AGAQFFFLVA A ALVVLLVG ...文字列: MGSAWSHPQF EKGSGLEVLF QGPSGHMAAR KCGAPPIAAD GSTRRPDCVT AVRTQARAPT QHYAESVARR QRVLTITAWL AVVVTGSFA LMQLATGAGG WYIALINVFT AVTFAIVPLL HRFGGLVAPL TFIGTAYVAI FAIGWDVGTD AGAQFFFLVA A ALVVLLVG IEHTALAVGL AAVAAGLVIA LEFLVPPDTG LQPPWAMSVS FVLTTVSACG VAVATVWFAL RDTARAEAVM EA EHDRSEA LLANMLPASI AERLKEPERN IIADKYDEAS VLFADIVGFT ERASSTAPAD LVRFLDRLYS AFDELVDQHG LEK IKVSGD SYMVVSGVPR PRPDHTQALA DFALDMTNVA AQLKDPRGNP VPLRVGLATG PVVAGVVGSR RFFYDVWGDA VNVA SRMES TDSVGQIQVP DEVYERLKDD FVLRERGHIN VKGKGVMRTW YLIGRKVAAD PGEVRGAEPR TAGVAAA |
-分子 #2: Nanobody Nb4
| 分子 | 名称: Nanobody Nb4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Vicugna pacos (アルパカ) Vicugna pacos (アルパカ) |
| 分子量 | 理論値: 13.771179 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MAQWQLVESG GGLVQAGGSL RLSCTASGII LSINSMGWYR QTAGNEREWV AFSTAGGSTT YADSVKGRFT ISRDNAKNTV YLQMNSLKP EDTAVYYCNT PAGRVGGTWG QGTPVTVSSH HHHHHEPEA |
-分子 #3: 3'-O-(N-METHYLANTHRANILOYL)-GUANOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: 3'-O-(N-METHYLANTHRANILOYL)-GUANOSINE-5'-TRIPHOSPHATE タイプ: ligand / ID: 3 / コピー数: 2 / 式: ONM |
|---|---|
| 分子量 | 理論値: 656.328 Da |
| Chemical component information |  ChemComp-ONM: |
-分子 #4: MANGANESE (II) ION
| 分子 | 名称: MANGANESE (II) ION / タイプ: ligand / ID: 4 / コピー数: 4 / 式: MN |
|---|---|
| 分子量 | 理論値: 54.938 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 詳細: 50 mM Tris pH 7.5, 200 mM NaCl, 0.1 % digitonin |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 48.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)