+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10217 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the RecBCD no Chi negative control complex | |||||||||
 マップデータ マップデータ | EM map of the RecBCD no Chi control structure | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 RecBCD / RecBCD /  exodeoxyribonuclease V activity / exodeoxyribonuclease V activity /  exodeoxyribonuclease V complex / DNA 5'-3' helicase / clearance of foreign intracellular DNA / DNA 3'-5' helicase / DNA translocase activity / single-stranded DNA helicase activity / exodeoxyribonuclease V complex / DNA 5'-3' helicase / clearance of foreign intracellular DNA / DNA 3'-5' helicase / DNA translocase activity / single-stranded DNA helicase activity /  相同組換え / 3'-5' DNA helicase activity ... 相同組換え / 3'-5' DNA helicase activity ... RecBCD / RecBCD /  exodeoxyribonuclease V activity / exodeoxyribonuclease V activity /  exodeoxyribonuclease V complex / DNA 5'-3' helicase / clearance of foreign intracellular DNA / DNA 3'-5' helicase / DNA translocase activity / single-stranded DNA helicase activity / exodeoxyribonuclease V complex / DNA 5'-3' helicase / clearance of foreign intracellular DNA / DNA 3'-5' helicase / DNA translocase activity / single-stranded DNA helicase activity /  相同組換え / 3'-5' DNA helicase activity / ATP-dependent activity, acting on DNA / 相同組換え / 3'-5' DNA helicase activity / ATP-dependent activity, acting on DNA /  DNA helicase activity / DNA endonuclease activity / DNA helicase activity / DNA endonuclease activity /  helicase activity / double-strand break repair via homologous recombination / response to radiation / DNA recombination / DNA damage response / magnesium ion binding / helicase activity / double-strand break repair via homologous recombination / response to radiation / DNA recombination / DNA damage response / magnesium ion binding /  ATP hydrolysis activity / ATP hydrolysis activity /  DNA binding / DNA binding /  ATP binding / ATP binding /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Escherichia coli (大腸菌) / synthetic construct (人工物) Escherichia coli (大腸菌) / synthetic construct (人工物) | |||||||||
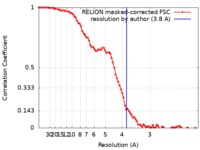
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.8 Å クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Cheng K / Wilkinson M / Wigley DB | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2020 ジャーナル: Nat Struct Mol Biol / 年: 2020タイトル: A conformational switch in response to Chi converts RecBCD from phage destruction to DNA repair. 著者: Kaiying Cheng / Martin Wilkinson / Yuriy Chaban / Dale B Wigley /  要旨: The RecBCD complex plays key roles in phage DNA degradation, CRISPR array acquisition (adaptation) and host DNA repair. The switch between these roles is regulated by a DNA sequence called Chi. We ...The RecBCD complex plays key roles in phage DNA degradation, CRISPR array acquisition (adaptation) and host DNA repair. The switch between these roles is regulated by a DNA sequence called Chi. We report cryo-EM structures of the Escherichia coli RecBCD complex bound to several different DNA forks containing a Chi sequence, including one in which Chi is recognized and others in which it is not. The Chi-recognized structure shows conformational changes in regions of the protein that contact Chi and reveals a tortuous path taken by the DNA. Sequence specificity arises from interactions with both the RecC subunit and the sequence itself. These structures provide molecular details for how Chi is recognized and insights into the changes that occur in response to Chi binding that switch RecBCD from bacteriophage destruction and CRISPR spacer acquisition to constructive host DNA repair. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10217.map.gz emd_10217.map.gz | 48.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10217-v30.xml emd-10217-v30.xml emd-10217.xml emd-10217.xml | 21.4 KB 21.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10217_fsc.xml emd_10217_fsc.xml | 8.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10217.png emd_10217.png | 90.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10217 http://ftp.pdbj.org/pub/emdb/structures/EMD-10217 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10217 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10217 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10217.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10217.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM map of the RecBCD no Chi control structure | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.048 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : RecBCD bound to a forked DNA substrate that does not contain Chi
| 全体 | 名称: RecBCD bound to a forked DNA substrate that does not contain Chi |
|---|---|
| 要素 |
|
-超分子 #1: RecBCD bound to a forked DNA substrate that does not contain Chi
| 超分子 | 名称: RecBCD bound to a forked DNA substrate that does not contain Chi タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 分子量 | 理論値: 355 KDa |
-超分子 #2: RecBCD
| 超分子 | 名称: RecBCD / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
-超分子 #3: DNA
| 超分子 | 名称: DNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #4 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 組換発現 | 生物種: synthetic construct (人工物) |
-分子 #1: RecBCD enzyme subunit RecB
| 分子 | 名称: RecBCD enzyme subunit RecB / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号:  RecBCD RecBCD |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 134.123688 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: GMSDVAETLD PLRLPLQGER LIEASAGTGK TFTIAALYLR LLLGLGGSAA FPRPLTVEEL LVVTFTEAAT AELRGRIRSN IHELRIACL RETTDNPLYE RLLEEIDDKA QAAQWLLLAE RQMDEAAVFT IHGFCQRMLN LNAFESGMLF EQQLIEDESL L RYQACADF ...文字列: GMSDVAETLD PLRLPLQGER LIEASAGTGK TFTIAALYLR LLLGLGGSAA FPRPLTVEEL LVVTFTEAAT AELRGRIRSN IHELRIACL RETTDNPLYE RLLEEIDDKA QAAQWLLLAE RQMDEAAVFT IHGFCQRMLN LNAFESGMLF EQQLIEDESL L RYQACADF WRRHCYPLPR EIAQVVFETW KGPQALLRDI NRYLQGEAPV IKAPPPDDET LASRHAQIVA RIDTVKQQWR DA VGELDAL IESSGIDRRK FNRSNQAKWI DKISAWAEEE TNSYQLPESL EKFSQRFLED RTKAGGETPR HPLFEAIDQL LAE PLSIRD LVITRALAEI RETVAREKRR RGELGFDDML SRLDSALRSE SGEVLAAAIR TRFPVAMIDE FQDTDPQQYR IFRR IWHHQ PETALLLIGD PKQAIYAFRG ADIFTYMKAR SEVHAHYTLD TNWRSAPGMV NSVNKLFSQT DDAFMFREIP FIPVK SAGK NQALRFVFKG ETQPAMKMWL MEGESCGVGD YQSTMAQVCA AQIRDWLQAG QRGEALLMNG DDARPVRASD ISVLVR SRQ EAAQVRDALT LLEIPSVYLS NRDSVFETLE AQEMLWLLQA VMTPERENTL RSALATSMMG LNALDIETLN NDEHAWD VV VEEFDGYRQI WRKRGVMPML RALMSARNIA ENLLATAGGE RRLTDILHIS ELLQEAGTQL ESEHALVRWL SQHILEPD S NASSQQMRLE SDKHLVQIVT IHKSKGLEYP LVWLPFITNF RVQEQAFYHD RHSFEAVLDL NAAPESVDLA EAERLAEDL RLLYVALTRS VWHCSLGVAP LVRRRGDKKG DTDVHQSALG RLLQKGEPQD AAGLRTCIEA LCDDDIAWQT AQTGDNQPWQ VNDVSTAEL NAKTLQRLPG DNWRVTSYSG LQQRGHGIAQ DLMPRLDVDA AGVASVVEEP TLTPHQFPRG ASPGTFLHSL F EDLDFTQP VDPNWVREKL ELGGFESQWE PVLTEWITAV LQAPLNETGV SLSQLSARNK QVEMEFYLPI SEPLIASQLD TL IRQFDPL SAGCPPLEFM QVRGMLKGFI DLVFRHEGRY YLLAYKSNWL GEDSSAYTQQ AMAAAMQAHR YDLQYQLYTL ALH RYLRHR IADYDYEHHF GGVIYLFLRG VDKEHPQQGI YTTRPNAGLI ALMDEMFAGM TLEEA |
-分子 #2: RecBCD enzyme subunit RecC
| 分子 | 名称: RecBCD enzyme subunit RecC / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号:  RecBCD RecBCD |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 128.974102 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MLRVYHSNRL DVLEALMEFI VERERLDDPF EPEMILVQST GMAQWLQMTL SQKFGIAANI DFPLPASFIW DMFVRVLPEI PKESAFNKQ SMSWKLMTLL PQLLEREDFT LLRHYLTDDS DKRKLFQLSS KAADLFDQYL VYRPDWLAQW ETGHLVEGLG E AQAWQAPL ...文字列: MLRVYHSNRL DVLEALMEFI VERERLDDPF EPEMILVQST GMAQWLQMTL SQKFGIAANI DFPLPASFIW DMFVRVLPEI PKESAFNKQ SMSWKLMTLL PQLLEREDFT LLRHYLTDDS DKRKLFQLSS KAADLFDQYL VYRPDWLAQW ETGHLVEGLG E AQAWQAPL WKALVEYTHQ LGQPRWHRAN LYQRFIETLE SATTCPPGLP SRVFICGISA LPPVYLQALQ ALGKHIEIHL LF TNPCRYY WGDIKDPAYL AKLLTRQRRH SFEDRELPLF RDSENAGQLF NSDGEQDVGN PLLASWGKLG RDYIYLLSDL ESS QELDAF VDVTPDNLLH NIQSDILELE NRAVAGVNIE EFSRSDNKRP LDPLDSSITF HVCHSPQREV EVLHDRLLAM LEED PTLTP RDIIVMVADI DSYSPFIQAV FGSAPADRYL PYAISDRRAR QSHPVLEAFI SLLSLPDSRF VSEDVLALLD VPVLA ARFD ITEEGLRYLR QWVNESGIRW GIDDDNVREL ELPATGQHTW RFGLTRMLLG YAMESAQGEW QSVLPYDESS GLIAEL VGH LASLLMQLNI WRRGLAQERP LEEWLPVCRD MLNAFFLPDA ETEAAMTLIE QQWQAIIAEG LGAQYGDAVP LSLLRDE LA QRLDQERISQ RFLAGPVNIC TLMPMRSIPF KVVCLLGMND GVYPRQLAPL GFDLMSQKPK RGDRSRRDDD RYLFLEAL I SAQQKLYISY IGRSIQDNSE RFPSVLVQEL IDYIGQSHYL PGDEALNCDE SEARVKAHLT CLHTRMPFDP QNYQPGERQ SYAREWLPAA SQAGKAHSEF VQPLPFTLPE TVPLETLQRF WAHPVRAFFQ MRLQVNFRTE DSEIPDTEPF ILEGLSRYQI NQQLLNALV EQDDAERLFR RFRAAGDLPY GAFGEIFWET QCQEMQQLAD RVIACRQPGQ SMEIDLACNG VQITGWLPQV Q PDGLLRWR PSLLSVAQGM QLWLEHLVYC ASGGNGESRL FLRKDGEWRF PPLAAEQALH YLSQLIEGYR EGMSAPLLVL PE SGGAWLK TCYDAQNDAM LDDDSTLQKA RTKFLQAYEG NMMVRGEGDD IWYQRLWRQL TPETMEAIVE QSQRFLLPLF RFN QS |
-分子 #3: RecBCD enzyme subunit RecD
| 分子 | 名称: RecBCD enzyme subunit RecD / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号:  RecBCD RecBCD |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 66.990367 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MKLQKQLLEA VEHKQLRPLD VQFALTVAGD EHPAVTLAAA LLSHDAGEGH VCLPLSRLEN NEASHPLLAT CVSEIGELQN WEECLLASQ AVSRGDEPTP MILCGDRLYL NRMWCNERTV ARFFNEVNHA IEVDEALLAQ TLDKLFPVSD EINWQKVAAA V ALTRRISV ...文字列: MKLQKQLLEA VEHKQLRPLD VQFALTVAGD EHPAVTLAAA LLSHDAGEGH VCLPLSRLEN NEASHPLLAT CVSEIGELQN WEECLLASQ AVSRGDEPTP MILCGDRLYL NRMWCNERTV ARFFNEVNHA IEVDEALLAQ TLDKLFPVSD EINWQKVAAA V ALTRRISV ISGGPGTGKT TTVAKLLAAL IQMADGERCR IRLAAPTGKA AARLTESLGK ALRQLPLTDE QKKRIPEDAS TL HRLLGAQ PGSQRLRHHA GNPLHLDVLV VDEASMIDLP MMSRLIDALP DHARVIFLGD RDQLASVEAG AVLGDICAYA NAG FTAERA RQLSRLTGTH VPAGTGTEAA SLRDSLCLLQ KSYRFGSDSG IGQLAAAINR GDKTAVKTVF QQDFTDIEKR LLQS GEDYI AMLEEALAGY GRYLDLLQAR AEPDLIIQAF NEYQLLCALR EGPFGVAGLN ERIEQFMQQK RKIHRHPHSR WYEGR PVMI ARNDSALGLF NGDIGIALDR GQGTRVWFAM PDGNIKSVQP SRLPEHETTW AMTVHKSQGS EFDHAALILP SQRTPV VTR ELVYTAVTRA RRRLSLYADE RILSAAIATR TERRSGLAAL FSSRE |
-分子 #4: Forked DNA substrate
| 分子 | 名称: Forked DNA substrate / タイプ: dna / ID: 4 詳細: We generated a forked DNA substrate with single-stranded overhangs by annealing two synthesised oligonucleotides コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 26.049602 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DG)(DA)(DG)(DC)(DG) (DA)(DC)(DT)(DG)(DC)(DA)(DC)(DT)(DA) (DC)(DA)(DA)(DC)(DA)(DG)(DA)(DA)(DC)(DC) (DA) (DT)(DG)(DG)(DT)(DT) ...文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DG)(DA)(DG)(DC)(DG) (DA)(DC)(DT)(DG)(DC)(DA)(DC)(DT)(DA) (DC)(DA)(DA)(DC)(DA)(DG)(DA)(DA)(DC)(DC) (DA) (DT)(DG)(DG)(DT)(DT)(DC)(DT)(DG) (DT)(DT)(DG)(DT)(DA)(DG)(DT)(DG)(DC)(DA) (DG)(DT) (DC)(DG)(DC)(DT)(DC)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: 1s blot before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 47710 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 1.4 µm / 倍率(公称値): 130000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 1.4 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 788 / 平均露光時間: 7.0 sec. / 平均電子線量: 45.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー