+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20798 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Integrin alpha-v beta-8 in complex with latent TGF-beta, Conformation ii (Primary map: sharpened. Additional map: unsharpened.) | ||||||||||||||||||||||||
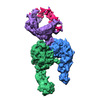
 マップデータ マップデータ | Integrin alpha-v beta-8 in complex with latent TGF-beta, Conformation ii, sharpened map | ||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Sus scrofa (ブタ) Sus scrofa (ブタ) | ||||||||||||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.6 Å クライオ電子顕微鏡法 / 解像度: 3.6 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Campbell MG / Cormier A / Cheng Y / Nishimura SL | ||||||||||||||||||||||||
| 資金援助 |  米国, 7件 米国, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2020 ジャーナル: Cell / 年: 2020タイトル: Cryo-EM Reveals Integrin-Mediated TGF-β Activation without Release from Latent TGF-β. 著者: Melody G Campbell / Anthony Cormier / Saburo Ito / Robert I Seed / Andrew J Bondesson / Jianlong Lou / James D Marks / Jody L Baron / Yifan Cheng / Stephen L Nishimura /  要旨: Integrin αvβ8 binds with exquisite specificity to latent transforming growth factor-β (L-TGF-β). This binding is essential for activating L-TGF-β presented by a variety of cell types. ...Integrin αvβ8 binds with exquisite specificity to latent transforming growth factor-β (L-TGF-β). This binding is essential for activating L-TGF-β presented by a variety of cell types. Inhibiting αvβ8-mediated TGF-β activation blocks immunosuppressive regulatory T cell differentiation, which is a potential therapeutic strategy in cancer. Using cryo-electron microscopy, structure-guided mutagenesis, and cell-based assays, we reveal the binding interactions between the entire αvβ8 ectodomain and its intact natural ligand, L-TGF-β, as well as two different inhibitory antibody fragments to understand the structural underpinnings of αvβ8 binding specificity and TGF-β activation. Our studies reveal a mechanism of TGF-β activation where mature TGF-β signals within the confines of L-TGF-β and the release and diffusion of TGF-β are not required. The structural details of this mechanism provide a rational basis for therapeutic strategies to inhibit αvβ8-mediated L-TGF-β activation. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20798.map.gz emd_20798.map.gz | 1.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20798-v30.xml emd-20798-v30.xml emd-20798.xml emd-20798.xml | 14.6 KB 14.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20798.png emd_20798.png | 93.1 KB | ||
| その他 |  emd_20798_additional.map.gz emd_20798_additional.map.gz emd_20798_additional_1.map.gz emd_20798_additional_1.map.gz | 52 MB 52 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20798 http://ftp.pdbj.org/pub/emdb/structures/EMD-20798 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20798 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20798 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6ujaC  6ujbC  6ujcC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10343 (タイトル: CryoEM dataset containing multiple conformations of the asymmetric αVβ8 integrin bound to latent TGF-β on a holey carbon grid (strongly preferred orientations) EMPIAR-10343 (タイトル: CryoEM dataset containing multiple conformations of the asymmetric αVβ8 integrin bound to latent TGF-β on a holey carbon grid (strongly preferred orientations)Data size: 1.2 TB Data #1: Unaligned 80-frame movies of αVβ8 integrin bound to latent TGF-β on a holey carbon grid [micrographs - multiframe] Data #2: Dose-weighted aligned micrographs of αVβ8 integrin bound to latent TGF-β on a holey carbon grid [micrographs - single frame] Data #3: Dose-weighted aligned particle stacks of αVβ8 integrin bound to latent TGF-β on a holey carbon grid [picked particles - single frame - processed])  EMPIAR-10344 (タイトル: CryoEM dataset containing multiple conformations of the asymmetric αVβ8 integrin bound to latent TGF-β on a graphene oxide grid (preferred orientations) EMPIAR-10344 (タイトル: CryoEM dataset containing multiple conformations of the asymmetric αVβ8 integrin bound to latent TGF-β on a graphene oxide grid (preferred orientations)Data size: 2.2 TB Data #1: Unaligned 80-frame movies of αVβ8 integrin bound to latent TGF-β on a graphene oxide grid [micrographs - multiframe] Data #2: Dose-weighted aligned micrographs of αVβ8 integrin bound to latent TGF-β on a graphene oxide grid [micrographs - single frame] Data #3: Dose-weighted aligned particle stacks of αVβ8 integrin bound to latent TGF-β on a graphene oxide grid [picked particles - single frame - processed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20798.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20798.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Integrin alpha-v beta-8 in complex with latent TGF-beta, Conformation ii, sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.345 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Integrin alpha-v beta-8 in complex with latent TGF-beta,...
| ファイル | emd_20798_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Integrin alpha-v beta-8 in complex with latent TGF-beta, Conformation ii (Additional unsharpened map.) | ||||||||||||
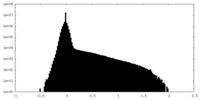
| 投影像・断面図 |
| ||||||||||||
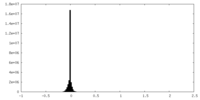
| 密度ヒストグラム |
-追加マップ: Integrin alpha-v beta-8 in complex with latent TGF-beta,...
| ファイル | emd_20798_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Integrin alpha-v beta-8 in complex with latent TGF-beta, Conformation ii (Additional unsharpened map.) | ||||||||||||
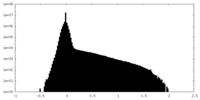
| 投影像・断面図 |
| ||||||||||||
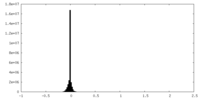
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Binary complex of Integrin alpha-v beta-8 with Latent TGF-beta-1 ...
| 全体 | 名称: Binary complex of Integrin alpha-v beta-8 with Latent TGF-beta-1 (L-TGF-b1) |
|---|---|
| 要素 |
|
-超分子 #1: Binary complex of Integrin alpha-v beta-8 with Latent TGF-beta-1 ...
| 超分子 | 名称: Binary complex of Integrin alpha-v beta-8 with Latent TGF-beta-1 (L-TGF-b1) タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 分子量 | 理論値: 260 KDa |
-超分子 #2: alpha-v beta-8 integrin
| 超分子 | 名称: alpha-v beta-8 integrin / タイプ: complex / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ) |
-超分子 #3: Transforming Growth Factor Beta-1 proprotein
| 超分子 | 名称: Transforming Growth Factor Beta-1 proprotein / タイプ: complex / ID: 3 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Sus scrofa (ブタ) Sus scrofa (ブタ) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換細胞: HEK293A Homo sapiens (ヒト) / 組換細胞: HEK293A |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 70.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
|---|---|
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 32689 |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X