+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of quinol-dependent Nitric Oxide Reductase (qNOR) from Alcaligenes xylosoxidans at 2.2 A resolution | |||||||||
 マップデータ マップデータ | main map used for model building | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | quinol-dependent Nitric Oxide Reductase /  proton transfer / proton transfer /  quinol binding (ヒドロキノン) / quinol binding (ヒドロキノン) /  ubiquinol oxidase / ubiquinol oxidase /  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 nitric oxide reductase (cytochrome c) / nitric oxide reductase activity / nitric oxide reductase (cytochrome c) / nitric oxide reductase activity /  cytochrome-c oxidase activity / cytochrome-c oxidase activity /  細胞呼吸 / 細胞呼吸 /  heme binding / heme binding /  生体膜 生体膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Achromobacter xylosoxidans (バクテリア) Achromobacter xylosoxidans (バクテリア) | |||||||||
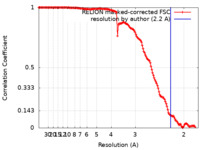
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.2 Å クライオ電子顕微鏡法 / 解像度: 2.2 Å | |||||||||
 データ登録者 データ登録者 | Flynn A / Antonyuk SV / Eady RR / Muench SP / Hasnain SS | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: A 2.2 Å cryoEM structure of a quinol-dependent NO Reductase shows close similarity to respiratory oxidases. 著者: Alex J Flynn / Svetlana V Antonyuk / Robert R Eady / Stephen P Muench / S Samar Hasnain /  要旨: Quinol-dependent nitric oxide reductases (qNORs) are considered members of the respiratory heme-copper oxidase superfamily, are unique to bacteria, and are commonly found in pathogenic bacteria where ...Quinol-dependent nitric oxide reductases (qNORs) are considered members of the respiratory heme-copper oxidase superfamily, are unique to bacteria, and are commonly found in pathogenic bacteria where they play a role in combating the host immune response. qNORs are also essential enzymes in the denitrification pathway, catalysing the reduction of nitric oxide to nitrous oxide. Here, we determine a 2.2 Å cryoEM structure of qNOR from Alcaligenes xylosoxidans, an opportunistic pathogen and a denitrifying bacterium of importance in the nitrogen cycle. This high-resolution structure provides insight into electron, substrate, and proton pathways, and provides evidence that the quinol binding site not only contains the conserved His and Asp residues but also possesses a critical Arg (Arg720) observed in cytochrome bo, a respiratory quinol oxidase. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16041.map.gz emd_16041.map.gz | 14.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16041-v30.xml emd-16041-v30.xml emd-16041.xml emd-16041.xml | 22 KB 22 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16041_fsc.xml emd_16041_fsc.xml | 14 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16041.png emd_16041.png | 112.4 KB | ||
| マスクデータ |  emd_16041_msk_1.map emd_16041_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| その他 |  emd_16041_half_map_1.map.gz emd_16041_half_map_1.map.gz emd_16041_half_map_2.map.gz emd_16041_half_map_2.map.gz | 192.5 MB 192.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16041 http://ftp.pdbj.org/pub/emdb/structures/EMD-16041 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16041 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16041 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8bgwMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16041.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16041.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | main map used for model building | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.91 Å | ||||||||||||||||||||||||||||||||||||
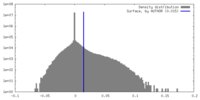
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16041_msk_1.map emd_16041_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
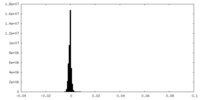
| 密度ヒストグラム |
-ハーフマップ: half map used in refinement
| ファイル | emd_16041_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map used in refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
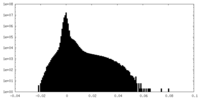
| 密度ヒストグラム |
-ハーフマップ: half map used for cross validation
| ファイル | emd_16041_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map used for cross validation | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
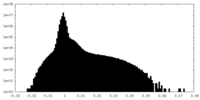
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : quinol-dependent Nitric Oxide Reductase
| 全体 | 名称: quinol-dependent Nitric Oxide Reductase |
|---|---|
| 要素 |
|
-超分子 #1: quinol-dependent Nitric Oxide Reductase
| 超分子 | 名称: quinol-dependent Nitric Oxide Reductase / タイプ: cell / ID: 1 / 親要素: 0 / 含まれる分子: #1 / 詳細: quinol-dependent Nitric Oxide Reductase |
|---|---|
| 由来(天然) | 生物種:   Achromobacter xylosoxidans (バクテリア) Achromobacter xylosoxidans (バクテリア) |
-分子 #1: Nitric oxide reductase subunit B
| 分子 | 名称: Nitric oxide reductase subunit B / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号:  nitric oxide reductase (cytochrome c) nitric oxide reductase (cytochrome c) |
|---|---|
| 由来(天然) | 生物種:   Achromobacter xylosoxidans (バクテリア) Achromobacter xylosoxidans (バクテリア) |
| 分子量 | 理論値: 84.724867 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MGPYRRLWFT LIAVLAVTFA LLGFYGGEVY RQAPPIPEEV ASADGTRLFG RDDILDGQTA WQSIGGMQLG SIWGHGAYQA PDWTADWLH RELMAWLDLA ARDAHGRDYG QLDAPAQAAL REQLKAEYRA NRADAAGGKL TLSPRRAQAV AQTEAYYDQL F SDAPALHR ...文字列: MGPYRRLWFT LIAVLAVTFA LLGFYGGEVY RQAPPIPEEV ASADGTRLFG RDDILDGQTA WQSIGGMQLG SIWGHGAYQA PDWTADWLH RELMAWLDLA ARDAHGRDYG QLDAPAQAAL REQLKAEYRA NRADAAGGKL TLSPRRAQAV AQTEAYYDQL F SDAPALHR SRENYAMKEN TLPDANRRRQ MTHFFFWTAW AAATEREGTS VTYTNNWPHE PLIGNHPSSE NVMWSIISVV VL LAGIGLL IWAWAFLRGK EEDEPPAPAR DPLTTFALTP SQRALGKYLF LVVALFGFQV LLGGFTAHYT VEGQKFYGID LSQ WFPYSL VRTWHIQSAL FWIATGFLAA GLFLAPLING GRDPKYQKAG VDILFWALVL VVVGSFAGNY LAIAQIMPPD LNFW LGHQG YEYVDLGRLW QIGKFAGICF WLVLMLRGIV PALRTPGGDK NLLALLTASV GAIGLFYGAG FFYGERTHLT VMEYW RWWI VHLWVEGFFE VFATTALAFI FSTLGLVSRR MATTASLASA SLFMLGGIPG TFHHLYFAGT TTPVMAVGAS FSALEV VPL IVLGHEAWEN WRLKTRAPWM ENLKWPLMCF VAVAFWNMLG AGVFGFMINP PVSLYYIQGL NTTPVHAHAA LFGVYGF LA LGFTLLVLRY IRPQYALSPG LMKLAFWGLN LGLALMIFTS LLPIGLIQFH ASVSEGMWYA RSEAFMQQDI LKTLRWGR T FGDVVFLLGA LAMVVQVILG LLSGKPAAAE PVLRAEPARR UniProtKB: Nitric oxide reductase subunit B |
-分子 #2: PROTOPORPHYRIN IX CONTAINING FE
| 分子 | 名称: PROTOPORPHYRIN IX CONTAINING FE / タイプ: ligand / ID: 2 / コピー数: 4 / 式: HEM |
|---|---|
| 分子量 | 理論値: 616.487 Da |
| Chemical component information |  ChemComp-HEM: |
-分子 #3: FE (III) ION
| 分子 | 名称: FE (III) ION / タイプ: ligand / ID: 3 / コピー数: 2 / 式: FE |
|---|---|
| 分子量 | 理論値: 55.845 Da |
-分子 #4: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #5: decyl 4-O-alpha-D-glucopyranosyl-1-thio-beta-D-glucopyranoside
| 分子 | 名称: decyl 4-O-alpha-D-glucopyranosyl-1-thio-beta-D-glucopyranoside タイプ: ligand / ID: 5 / コピー数: 2 / 式: 10M |
|---|---|
| 分子量 | 理論値: 498.628 Da |
| Chemical component information | 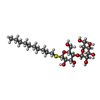 ChemComp-10M: |
-分子 #6: UBIQUINONE-1
| 分子 | 名称: UBIQUINONE-1 / タイプ: ligand / ID: 6 / コピー数: 2 / 式: UQ1 |
|---|---|
| 分子量 | 理論値: 250.29 Da |
| Chemical component information | 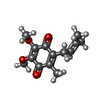 ChemComp-UQ1: |
-分子 #7: (1R)-2-{[(R)-(2-AMINOETHOXY)(HYDROXY)PHOSPHORYL]OXY}-1-[(DODECANO...
| 分子 | 名称: (1R)-2-{[(R)-(2-AMINOETHOXY)(HYDROXY)PHOSPHORYL]OXY}-1-[(DODECANOYLOXY)METHYL]ETHYL (9Z)-OCTADEC-9-ENOATE タイプ: ligand / ID: 7 / コピー数: 13 / 式: LOP |
|---|---|
| 分子量 | 理論値: 661.89 Da |
| Chemical component information |  ChemComp-LOP: |
-分子 #8: water
| 分子 | 名称: water / タイプ: ligand / ID: 8 / コピー数: 449 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 7 / 構成要素 - 濃度: 50.0 mM 構成要素 - 式: Tris-HCL  トリスヒドロキシメチルアミノメタン トリスヒドロキシメチルアミノメタン構成要素 - 名称: Tris-HCL  トリスヒドロキシメチルアミノメタン トリスヒドロキシメチルアミノメタン詳細: 50mM Tris-HCl, 150mM NaCl, 0.05% DTM |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: Sample at 3 mg/mL was applied to glow-discharged Quantifoil Au R1.2/1.3 holey carbo grids. |
| 詳細 | Sample was monodisperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 130000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 130000 |
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris / エネルギーフィルター - スリット幅: 10 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最低: 80.0 K / 最高: 114.0 K |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 5466 / 平均露光時間: 6.11 sec. / 平均電子線量: 34.9 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: RECIPROCAL / プロトコル: RIGID BODY FIT / 温度因子: 47 当てはまり具合の基準: Cross-correlation coefficient |
|---|---|
| 得られたモデル |  PDB-8bgw: |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)