+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Subtomogram average of authentic mumps virus nucleocapsid from HeLa cell lysate of short helical pitch | |||||||||
 マップデータ マップデータ | Minor conformation of authentic mumps virus nucleocapsid from the lysate of HeLa cells after stress treatment by subtomogram averaging | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Helical filament /  Nucleocapsid (カプシド) / Protein-RNA complex / Nucleocapsid (カプシド) / Protein-RNA complex /  Scaffold (足場) / Scaffold (足場) /  VIRUS (ウイルス) VIRUS (ウイルス) | |||||||||
| 生物種 |  Mumps virus genotype A (ウイルス) Mumps virus genotype A (ウイルス) | |||||||||
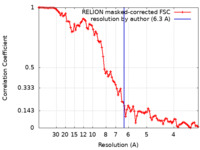
| 手法 | サブトモグラム平均法 /  クライオ電子顕微鏡法 / 解像度: 6.3 Å クライオ電子顕微鏡法 / 解像度: 6.3 Å | |||||||||
 データ登録者 データ登録者 | Mahamid J / Zhang X / Pflaesterer T | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2023 ジャーナル: Cell / 年: 2023タイトル: Molecular mechanisms of stress-induced reactivation in mumps virus condensates. 著者: Xiaojie Zhang / Sindhuja Sridharan / Ievgeniia Zagoriy / Christina Eugster Oegema / Cyan Ching / Tim Pflaesterer / Herman K H Fung / Isabelle Becher / Ina Poser / Christoph W Müller / ...著者: Xiaojie Zhang / Sindhuja Sridharan / Ievgeniia Zagoriy / Christina Eugster Oegema / Cyan Ching / Tim Pflaesterer / Herman K H Fung / Isabelle Becher / Ina Poser / Christoph W Müller / Anthony A Hyman / Mikhail M Savitski / Julia Mahamid /  要旨: Negative-stranded RNA viruses can establish long-term persistent infection in the form of large intracellular inclusions in the human host and cause chronic diseases. Here, we uncover how cellular ...Negative-stranded RNA viruses can establish long-term persistent infection in the form of large intracellular inclusions in the human host and cause chronic diseases. Here, we uncover how cellular stress disrupts the metastable host-virus equilibrium in persistent infection and induces viral replication in a culture model of mumps virus. Using a combination of cell biology, whole-cell proteomics, and cryo-electron tomography, we show that persistent viral replication factories are dynamic condensates and identify the largely disordered viral phosphoprotein as a driver of their assembly. Upon stress, increased phosphorylation of the phosphoprotein at its interaction interface with the viral polymerase coincides with the formation of a stable replication complex. By obtaining atomic models for the authentic mumps virus nucleocapsid, we elucidate a concomitant conformational change that exposes the viral genome to its replication machinery. These events constitute a stress-mediated switch within viral condensates that provide an environment to support upregulation of viral replication. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13136.map.gz emd_13136.map.gz | 8.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13136-v30.xml emd-13136-v30.xml emd-13136.xml emd-13136.xml | 12.6 KB 12.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13136_fsc.xml emd_13136_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13136.png emd_13136.png | 40.3 KB | ||
| Filedesc metadata |  emd-13136.cif.gz emd-13136.cif.gz | 5.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13136 http://ftp.pdbj.org/pub/emdb/structures/EMD-13136 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13136 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13136 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13136.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13136.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Minor conformation of authentic mumps virus nucleocapsid from the lysate of HeLa cells after stress treatment by subtomogram averaging | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.6938 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Authentic Mumps virus nucleocapsid-RNA complex
| 全体 | 名称: Authentic Mumps virus nucleocapsid-RNA complex |
|---|---|
| 要素 |
|
-超分子 #1: Authentic Mumps virus nucleocapsid-RNA complex
| 超分子 | 名称: Authentic Mumps virus nucleocapsid-RNA complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Mumps virus genotype A (ウイルス) Mumps virus genotype A (ウイルス) |
-分子 #1: Mumps virus nucleocapsid
| 分子 | 名称: Mumps virus nucleocapsid / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mumps virus genotype A (ウイルス) / 株: Enders Mumps virus genotype A (ウイルス) / 株: Enders |
| 配列 | 文字列: MSSVLKAFER FTIEQELQDR GEEGSIPPET LKSAVKVFVI NTPNPTTRYQ MLNFCLRIIC SQNARASHRV GALITLFSLP SAGMQNHIRL ADRSPEAQIE RCEIDGFEPG TYRLIPNARA NLTANEIAAY ALLADDLPPT INNGTPYVHA DVEGQPCDEI EQFLDRCYSV ...文字列: MSSVLKAFER FTIEQELQDR GEEGSIPPET LKSAVKVFVI NTPNPTTRYQ MLNFCLRIIC SQNARASHRV GALITLFSLP SAGMQNHIRL ADRSPEAQIE RCEIDGFEPG TYRLIPNARA NLTANEIAAY ALLADDLPPT INNGTPYVHA DVEGQPCDEI EQFLDRCYSV LIQAWVMVCK CMTAYDQPAG SADRRFAKYQ QQGRLEARYM LQPEAQRLIQ TAIRKSLVVR QYLTFELQLA RRQGLLSNRY YAMVGDIGKY IENSGLTAFF LTLKYALGTK WSPLSLAAFT GELTKLRSLM MLYRDIGEQA RYLALLEAPQ IMDFAPGGYP LIFSYAMGVG TVLDAQMRNY TYARPFLNGY YFQIGVETAR RQQGTVDNRV ADDLGLTPEQ RTEVTQLVDR LARGRGAGIP GGPVNPFVPP VQQQQPAAVY ADIPALEESD DDGDEDGGAG FQNGVQVPAV RQGGQTDFRA QPLQDPIQAQ LFMPLYPQVS NIPSNQNHQI NRIGGLENQD LLRYNENGDS QQDARGEHGN TFPNNPNQNA QLQVGDWDE |
-分子 #2: RNA
| 分子 | 名称: RNA / タイプ: rna / ID: 2 |
|---|---|
| 由来(天然) | 生物種:  Mumps virus genotype A (ウイルス) Mumps virus genotype A (ウイルス) |
| 配列 | 文字列: UUUUUU |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 2.5 µm / 倍率(公称値): 81000 Bright-field microscopy / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 2.5 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / 平均電子線量: 2.5 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー