+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of DHQS/EPSPS dimer from Candida albicans Aro1 | |||||||||
 マップデータ マップデータ | Sharpened map from focused refinement of DHQS/EPSPS region | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  BIOSYNTHETIC PROTEIN (生合成) BIOSYNTHETIC PROTEIN (生合成) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 3-dehydroquinate synthase / 3-dehydroquinate synthase /  3-dehydroquinate synthase activity / 3-dehydroquinate synthase activity /  shikimate kinase / shikimate kinase /  shikimate kinase activity / shikimate dehydrogenase (NADP+) / shikimate kinase activity / shikimate dehydrogenase (NADP+) /  3-ホスホシキミ酸-1-カルボキシビニルトランスフェラーゼ / 3-ホスホシキミ酸-1-カルボキシビニルトランスフェラーゼ /  3-phosphoshikimate 1-carboxyvinyltransferase activity / shikimate 3-dehydrogenase (NADP+) activity / 3-phosphoshikimate 1-carboxyvinyltransferase activity / shikimate 3-dehydrogenase (NADP+) activity /  3-dehydroquinate dehydratase / 3-dehydroquinate dehydratase /  3-dehydroquinate dehydratase activity ... 3-dehydroquinate dehydratase activity ... 3-dehydroquinate synthase / 3-dehydroquinate synthase /  3-dehydroquinate synthase activity / 3-dehydroquinate synthase activity /  shikimate kinase / shikimate kinase /  shikimate kinase activity / shikimate dehydrogenase (NADP+) / shikimate kinase activity / shikimate dehydrogenase (NADP+) /  3-ホスホシキミ酸-1-カルボキシビニルトランスフェラーゼ / 3-ホスホシキミ酸-1-カルボキシビニルトランスフェラーゼ /  3-phosphoshikimate 1-carboxyvinyltransferase activity / shikimate 3-dehydrogenase (NADP+) activity / 3-phosphoshikimate 1-carboxyvinyltransferase activity / shikimate 3-dehydrogenase (NADP+) activity /  3-dehydroquinate dehydratase / 3-dehydroquinate dehydratase /  3-dehydroquinate dehydratase activity / chorismate biosynthetic process / aromatic amino acid family biosynthetic process / amino acid biosynthetic process / 3-dehydroquinate dehydratase activity / chorismate biosynthetic process / aromatic amino acid family biosynthetic process / amino acid biosynthetic process /  リン酸化 / リン酸化 /  ATP binding / ATP binding /  metal ion binding / metal ion binding /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Candida albicans (酵母) Candida albicans (酵母) | |||||||||
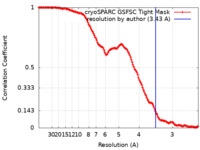
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.43 Å クライオ電子顕微鏡法 / 解像度: 3.43 Å | |||||||||
 データ登録者 データ登録者 | Quade B / Borek D / Otwinowski Z | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Life Sci Alliance / 年: 2022 ジャーナル: Life Sci Alliance / 年: 2022タイトル: Molecular analysis and essentiality of Aro1 shikimate biosynthesis multi-enzyme in . 著者: Peter J Stogios / Sean D Liston / Cameron Semper / Bradley Quade / Karolina Michalska / Elena Evdokimova / Shane Ram / Zbyszek Otwinowski / Dominika Borek / Leah E Cowen / Alexei Savchenko /   要旨: In the human fungal pathogen , encodes an essential multi-enzyme that catalyses consecutive steps in the shikimate pathway for biosynthesis of chorismate, a precursor to folate and the aromatic ...In the human fungal pathogen , encodes an essential multi-enzyme that catalyses consecutive steps in the shikimate pathway for biosynthesis of chorismate, a precursor to folate and the aromatic amino acids. We obtained the first molecular image of Aro1 that reveals the architecture of all five enzymatic domains and their arrangement in the context of the full-length protein. Aro1 forms a flexible dimer allowing relative autonomy of enzymatic function of the individual domains. Our activity and in cellulo data suggest that only four of Aro1's enzymatic domains are functional and essential for viability of , whereas the 3-dehydroquinate dehydratase (DHQase) domain is inactive because of active site substitutions. We further demonstrate that in , the type II DHQase Dqd1 can compensate for the inactive DHQase domain of Aro1, suggesting an unrecognized essential role for this enzyme in shikimate biosynthesis. In contrast, in and , which do not encode a Dqd1 homolog, Aro1 DHQase domains are enzymatically active, highlighting diversity across species. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26358.map.gz emd_26358.map.gz | 230.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26358-v30.xml emd-26358-v30.xml emd-26358.xml emd-26358.xml | 23 KB 23 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26358_fsc.xml emd_26358_fsc.xml | 13.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26358.png emd_26358.png | 89.3 KB | ||
| Filedesc metadata |  emd-26358.cif.gz emd-26358.cif.gz | 6.9 KB | ||
| その他 |  emd_26358_additional_1.map.gz emd_26358_additional_1.map.gz emd_26358_additional_2.map.gz emd_26358_additional_2.map.gz emd_26358_half_map_1.map.gz emd_26358_half_map_1.map.gz emd_26358_half_map_2.map.gz emd_26358_half_map_2.map.gz | 123.1 MB 213.6 MB 226.4 MB 226.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26358 http://ftp.pdbj.org/pub/emdb/structures/EMD-26358 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26358 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26358 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26358.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26358.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map from focused refinement of DHQS/EPSPS region | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.2495 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Raw map from focused refinement of DHQS/EPSPS region
| ファイル | emd_26358_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
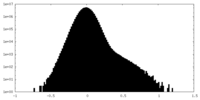
| 注釈 | Raw map from focused refinement of DHQS/EPSPS region | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
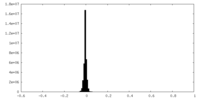
| 密度ヒストグラム |
-追加マップ: Postprocessed map of DHQS and EPSPS region from...
| ファイル | emd_26358_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
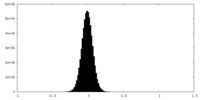
| 注釈 | Postprocessed map of DHQS and EPSPS region from broken particles at 2.94A. Used for model building. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
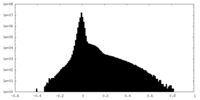
| 密度ヒストグラム |
-ハーフマップ: Half map B from focused refinement of DHQS/EPSPS region
| ファイル | emd_26358_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
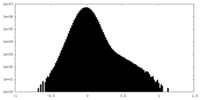
| 注釈 | Half map B from focused refinement of DHQS/EPSPS region | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
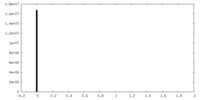
| 密度ヒストグラム |
-ハーフマップ: Half map A from focused refinement of DHQS/EPSPS region
| ファイル | emd_26358_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A from focused refinement of DHQS/EPSPS region | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
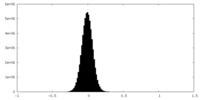
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Aro1 from Candida albicans
| 全体 | 名称: Aro1 from Candida albicans |
|---|---|
| 要素 |
|
-超分子 #1: Aro1 from Candida albicans
| 超分子 | 名称: Aro1 from Candida albicans / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Candida albicans (酵母) / 株: CaLC6830 Candida albicans (酵母) / 株: CaLC6830 |
| 分子量 | 理論値: 169.4 KDa |
-分子 #1: Pentafunctional AROM polypeptide
| 分子 | 名称: Pentafunctional AROM polypeptide / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号:  3-dehydroquinate synthase 3-dehydroquinate synthase |
|---|---|
| 由来(天然) | 生物種:   Candida albicans (酵母) / 株: SC5314 / ATCC MYA-2876 Candida albicans (酵母) / 株: SC5314 / ATCC MYA-2876 |
| 分子量 | 理論値: 169.59625 KDa |
| 配列 | 文字列: MSIEKVPILG KETIHVGYGI ADHIVREVIA NLASSTYVIV TDTNMARTPQ YSKLTDDFKT NLSEKRPESR LLTYCVSPGE NNKNRATKA AVEDFLLQQG CTRDTVILAV GGGVIGDMIG FVAATFMRGV RVVQVPTTLL AMVDSSVGGK TAIDTPLGKN F IGAFHQPE ...文字列: MSIEKVPILG KETIHVGYGI ADHIVREVIA NLASSTYVIV TDTNMARTPQ YSKLTDDFKT NLSEKRPESR LLTYCVSPGE NNKNRATKA AVEDFLLQQG CTRDTVILAV GGGVIGDMIG FVAATFMRGV RVVQVPTTLL AMVDSSVGGK TAIDTPLGKN F IGAFHQPE YVFCDVSFLE TLPARQFING MAEVVKTAAI WNEEEFTRLE NFSKKFLSVV TSKKPDLQSI KAELVKTVLE SV RVKAGVV SSDEKEAGLR NLLNFGHTIG HAIEAVLTPE ALHGECVSIG MIKEAELSRY LGILPPVAVA RLSKCLVAYG LPV SIDDKE FLKKVGPKRH YVEIDILLKK MAIDKKNDGS KIRCVLLEKI GKCYQLKAHQ VSKQDLSFVL TDEVLVHPFT NPPK ENIIV PPGSKSISNR ALILAALGNG TVRVKNLLHS DDTKHMLDAV ASLKGAEIST EDNGETIVVK GNGGNLVTSG EELYL GNAG TASRFLTTVA SLVGKSQASD DVILTGNARM QERPIGPLVD ALGSNGSEIE YLNKQGSLPL KISAGNGLKG GRIELA ATI SSQYVSSILM CAPYAKEPVT LALVGGKPIS QLYIDMTCAM MKSFGIEVTK STTEEYTYHI PKGTYKNPSE YVIESDA SS ATYPLAFAAM TGTSCTIPNI GSSSLQGDAK FAVDVLKPMG CKVEQTTTST TVTGPPRGHL KPLPHVDMEP MTDAFLTA S VVAAVAKGGS STSITGIANQ RVKECNRIEA MVTELAKFGV PANELPDGIE IHGIDIEDLK TPEISKRGVS SYDDHRVAM SFSLLAGLCK EPVLILERST TGKTWPGWWD ILHSKFKIEL DGYEPPFNTD KHVDKSSDKS IIVIGMRGTG KSTLSEWLAS FLGFKMLDM DKYLEEKLGT GIKSLIKAKG WEYFRQEEAI VAKECFTKFS KGYVLSTGGG IVEGEDARQQ LKSYADNGGI V LHLHRDLD ETVTFLAADT TRPAYSSEVQ EVWLRREKWY HECSNYHFYS SHCSTEDEFN HLRRSFVNYI KLITGAERPV VP AGRSAAV VLTSPDLNEV VGDLESITIG ADAVELRVDL FKDTSAEFVA AQIAVIRKHA DLPIIYTVRT VSQGGKFPDE NVD ELKSLL LLGIRLGVAY VDLQLTAPNE LIEEISSKKG FTRVIGTYQD INGELKWNNV EWKNKYNQGV SMNADIVRLV GKAN SIQDN LDLENFKKQN TLKPLIAFNL GSQGKLSQVL NGTFTPISHK LLPNDEEFLT IGELNQTYFD IGGFTAKKFW VIGSP IEHS RSPNLHNAGY KALNLPYQFG RFEATDVDVV YDNLINKPDF GGLAITMPLK LDIMKFATKL SDAAETIGAV NTLIPI EGG YFGDNTDWVG ISNSFIRAGV PPKSSSNGLV VGAGGTSRAA IYALHQMGCA KIYLVNRTAA KLEELVKSFP KDYNLEI VE TEQQADKASK VSLAVSCIPA DKPLDGEVLK KIERILSNGS EQSAGFKPTL LEASYKPRVT PIMKLTEEQY KWKVIPGV E MLVNQGDRQF KLHTGFTAPY EIIHRAVVEE UniProtKB: Pentafunctional AROM polypeptide |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.2 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 80 sec. / 前処理 - 雰囲気: AIR | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 105000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 83.5 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT | ||||||||
| 得られたモデル |  PDB-7u5t: |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X