+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 70S ribosome with a and P-site tRNAs in chloramphenicol-treated Mycoplasma pneumoniae cells, K3 data | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  In situ (In situ) / In situ (In situ) /  cryo-electron tomography / bacterial ribosome / cryo-electron tomography / bacterial ribosome /  Chloramphenicol (クロラムフェニコール) / Chloramphenicol (クロラムフェニコール) /  antibiotic (抗生物質) / antibiotic (抗生物質) /  RIBOSOME (リボソーム) RIBOSOME (リボソーム) | |||||||||
| 生物種 |  Mycoplasmoides pneumoniae M129 (バクテリア) Mycoplasmoides pneumoniae M129 (バクテリア) | |||||||||
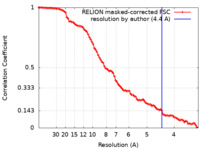
| 手法 | サブトモグラム平均法 /  クライオ電子顕微鏡法 / 解像度: 4.4 Å クライオ電子顕微鏡法 / 解像度: 4.4 Å | |||||||||
 データ登録者 データ登録者 | Xue L / Spahn C / Schacherl M / Mahamid J | |||||||||
| 資金援助 |  米国, 米国,  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structural evidence of context-dependent action of chloramphenicol in cells 著者: Xue L / Spahn C / Schacherl M / Mahamid J | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17138.map.gz emd_17138.map.gz | 7.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17138-v30.xml emd-17138-v30.xml emd-17138.xml emd-17138.xml | 16.3 KB 16.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17138_fsc.xml emd_17138_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17138.png emd_17138.png | 226.8 KB | ||
| マスクデータ |  emd_17138_msk_1.map emd_17138_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17138.cif.gz emd-17138.cif.gz | 4.9 KB | ||
| その他 |  emd_17138_half_map_1.map.gz emd_17138_half_map_1.map.gz emd_17138_half_map_2.map.gz emd_17138_half_map_2.map.gz | 49.7 MB 49.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17138 http://ftp.pdbj.org/pub/emdb/structures/EMD-17138 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17138 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17138 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17138.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17138.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.7005 Å | ||||||||||||||||||||||||||||||||||||
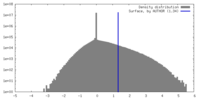
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17138_msk_1.map emd_17138_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
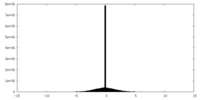
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_17138_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
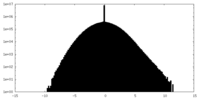
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_17138_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Mycoplasma pneumoniae M129 cells treated with chloramphenicol
| 全体 | 名称: Mycoplasma pneumoniae M129 cells treated with chloramphenicol |
|---|---|
| 要素 |
|
-超分子 #1: Mycoplasma pneumoniae M129 cells treated with chloramphenicol
| 超分子 | 名称: Mycoplasma pneumoniae M129 cells treated with chloramphenicol タイプ: cell / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  Mycoplasmoides pneumoniae M129 (バクテリア) Mycoplasmoides pneumoniae M129 (バクテリア) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 詳細: Modified Hayflick medium: 14.7g/l Difco PPLO(Becton Dickinson), 20% (v/v) Gibco horse serum (New Zealand origin), 100 mM HEPES-Na; pH 7.4, 1% (w/w) glucose, 0.002% (w/w) phenol red, 1000 U/ml penicillin G. |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE-PROPANE / 装置: HOMEMADE PLUNGER 詳細: Back-side blotting for 2-3 seconds before plunging using a manual plunger without an environmental control chamber.. |
| 詳細 | Mycoplasma pneumoniae M129 cells were grown on gold Quantifoil grids at 37 Celsius in modified Hayflick medium. Treatment with chloramphenicol at a final concentration of 0.2 mg/ml was performed for approximately 15 minutes before plunge freezing. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.25 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 64000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.25 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 64000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 1 / 平均電子線量: 3.34 e/Å2 詳細: Gatan K3 camera in non-CDS counting mode, targeted dose rate on camera ~20 e/pixel/second, 10 frames per tilt image, constant exposure time for each tilt, pixel size 1.329A |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)