+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of C-mannosyltransferase CeDPY19, in apo state, bound to CMT2-Fab and anti-Fab nanobody | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein C-linked glycosylation via 2'-alpha-mannosyl-L-tryptophan /  mannosyltransferase activity / nuclear inner membrane / mannosyltransferase activity / nuclear inner membrane /  転移酵素; グリコシル基を移すもの; 六炭糖残基を移すもの / 転移酵素; グリコシル基を移すもの; 六炭糖残基を移すもの /  nervous system development / nervous system development /  細胞分化 / carbohydrate metabolic process / endoplasmic reticulum membrane / perinuclear region of cytoplasm / 細胞分化 / carbohydrate metabolic process / endoplasmic reticulum membrane / perinuclear region of cytoplasm /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Caenorhabditis elegans (センチュウ) / synthetic construct (人工物) Caenorhabditis elegans (センチュウ) / synthetic construct (人工物) | |||||||||
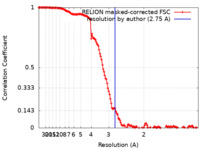
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.75 Å クライオ電子顕微鏡法 / 解像度: 2.75 Å | |||||||||
 データ登録者 データ登録者 | Bloch JS / Mukherjee S / Irobalieva R / Kossiakoff AA / Goddard-Borger ED / Locher KP | |||||||||
| 資金援助 |  スイス, 1件 スイス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Chem Biol / 年: 2023 ジャーナル: Nat Chem Biol / 年: 2023タイトル: Structure, sequon recognition and mechanism of tryptophan C-mannosyltransferase. 著者: Joël S Bloch / Alan John / Runyu Mao / Somnath Mukherjee / Jérémy Boilevin / Rossitza N Irobalieva / Tamis Darbre / Nichollas E Scott / Jean-Louis Reymond / Anthony A Kossiakoff / Ethan D ...著者: Joël S Bloch / Alan John / Runyu Mao / Somnath Mukherjee / Jérémy Boilevin / Rossitza N Irobalieva / Tamis Darbre / Nichollas E Scott / Jean-Louis Reymond / Anthony A Kossiakoff / Ethan D Goddard-Borger / Kaspar P Locher /    要旨: C-linked glycosylation is essential for the trafficking, folding and function of secretory and transmembrane proteins involved in cellular communication processes. The tryptophan C- ...C-linked glycosylation is essential for the trafficking, folding and function of secretory and transmembrane proteins involved in cellular communication processes. The tryptophan C-mannosyltransferase (CMT) enzymes that install the modification attach a mannose to the first tryptophan of WxxW/C sequons in nascent polypeptide chains by an unknown mechanism. Here, we report cryogenic-electron microscopy structures of Caenorhabditis elegans CMT in four key states: apo, acceptor peptide-bound, donor-substrate analog-bound and as a trapped ternary complex with both peptide and a donor-substrate mimic bound. The structures indicate how the C-mannosylation sequon is recognized by this CMT and its paralogs, and how sequon binding triggers conformational activation of the donor substrate: a process relevant to all glycosyltransferase C superfamily enzymes. Our structural data further indicate that the CMTs adopt an unprecedented electrophilic aromatic substitution mechanism to enable the C-glycosylation of proteins. These results afford opportunities for understanding human disease and therapeutic targeting of specific CMT paralogs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14780.map.gz emd_14780.map.gz | 228.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14780-v30.xml emd-14780-v30.xml emd-14780.xml emd-14780.xml | 18.1 KB 18.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14780_fsc.xml emd_14780_fsc.xml | 14.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14780.png emd_14780.png | 98.2 KB | ||
| その他 |  emd_14780_half_map_1.map.gz emd_14780_half_map_1.map.gz emd_14780_half_map_2.map.gz emd_14780_half_map_2.map.gz | 194.3 MB 194.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14780 http://ftp.pdbj.org/pub/emdb/structures/EMD-14780 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14780 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14780 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14780.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14780.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.66 Å | ||||||||||||||||||||||||||||||||||||
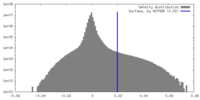
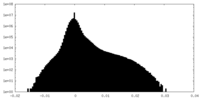
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_14780_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
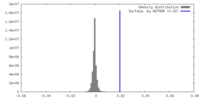
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_14780_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
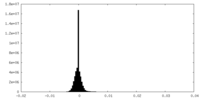
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : C-mannosyltransferase CeDPY19, in apo state, bound to CMT2-Fab an...
| 全体 | 名称: C-mannosyltransferase CeDPY19, in apo state, bound to CMT2-Fab and anti-Fab nanobody |
|---|---|
| 要素 |
|
-超分子 #1: C-mannosyltransferase CeDPY19, in apo state, bound to CMT2-Fab an...
| 超分子 | 名称: C-mannosyltransferase CeDPY19, in apo state, bound to CMT2-Fab and anti-Fab nanobody タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:   Caenorhabditis elegans (センチュウ) Caenorhabditis elegans (センチュウ) |
-分子 #1: CMT2-Fab heavy chain
| 分子 | 名称: CMT2-Fab heavy chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 25.000723 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF NISSSSIHWV RQAPGKGLEW VASISSSYGY TSYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC ARSSSVYWSW WGYSAFDYWG QGTLVTVSSA STKGPSVFPL APSSKSTSGG TAALGCLVKD Y FPEPVTVS ...文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF NISSSSIHWV RQAPGKGLEW VASISSSYGY TSYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC ARSSSVYWSW WGYSAFDYWG QGTLVTVSSA STKGPSVFPL APSSKSTSGG TAALGCLVKD Y FPEPVTVS WNSGALTSGV HTFPAVLQSS GLYSLSSVVT VPSSSLGTQT YICNVNHKPS NTKVDKKVEP KSCDKTHT |
-分子 #2: Anti-Fab nanobody
| 分子 | 名称: Anti-Fab nanobody / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 13.390644 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: GSQVQLQESG GGLVQPGGSL RLSCAASGRT ISRYAMSWFR QAPGKEREFV AVARRSGDGA FYADSVQGRF TVSRDDAKNT VYLQMNSLK PEDTAVYYCA IDSDTFYSGS YDYWGQGTQV TVSS |
-分子 #3: CMT2-Fab light chain
| 分子 | 名称: CMT2-Fab light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 23.23875 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQGASEPITF GQGTKVEIKR TVAAPSVFIF PPSDSQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD ...文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQGASEPITF GQGTKVEIKR TVAAPSVFIF PPSDSQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD SKDSTYSLSS TLTLSKADYE KHKVYACEVT HQGLSSPVTK SFNRGEC |
-分子 #4: C-mannosyltransferase dpy-19
| 分子 | 名称: C-mannosyltransferase dpy-19 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO EC番号:  転移酵素; グリコシル基を移すもの; 六炭糖残基を移すもの 転移酵素; グリコシル基を移すもの; 六炭糖残基を移すもの |
|---|---|
| 由来(天然) | 生物種:   Caenorhabditis elegans (センチュウ) Caenorhabditis elegans (センチュウ) |
| 分子量 | 理論値: 80.892602 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MAKKPKNSPE KSKYSSDTSS SLYSQTWLAS VVIIGLLVGY INYQHVYTLF ENDKHFSHLA DFEREMAYRT EMGLYYSYYK TIINAPSFL EGVQEITHDT VTEHGHEINT LNRFNLYPEV ILAFLYRPFR AFAKSANWQI ELCWQVNRGE LRPVESCEGI G NPHYFYIT ...文字列: MAKKPKNSPE KSKYSSDTSS SLYSQTWLAS VVIIGLLVGY INYQHVYTLF ENDKHFSHLA DFEREMAYRT EMGLYYSYYK TIINAPSFL EGVQEITHDT VTEHGHEINT LNRFNLYPEV ILAFLYRPFR AFAKSANWQI ELCWQVNRGE LRPVESCEGI G NPHYFYIT GVFIVAGTVA SSIFYLGVLV SDSIFGGFLS VLCFAFNHGE ATRVQWTPPL RESFAFPFII GHIAILTFVI KY KKSGHSM ILLLTSMAVP ALLFWQFTQF AFFTQICSIF LAFSLDLIPF STAKTVIHSH IISFLIGFLL LFGNEMMITA LYF PSILAL GMIIYISPLL SNLKFRPAYV LFLAIIFASI TLGLKIGLSK GLGIEDDAHI FDILRSKFTS FANFHTRLYT CSAE FDFIQ YSTIEKLCGT LLIPLALISL VTFVFNFVKN TNLLWRNSEE IGENGEILYN VVQLCCSTVM AFLIMRLKLF MTPHL CIVA ALFANSKLLG GDRISKTIRV SALVGVIAIL FYRGIPNIRQ QLNVKGEYSN PDQEMLFDWI QHNTKQDAVF AGTMPV MAN VKLTTLRPIV NHPHYEHVGI RERTLKVYSM FSKKPIAEVH KIMKEMGVNY FVFQLMNCSN DERRPECVYR GMWDEED PK NSGRTALCDL WILAANSKDN SRIAPFKIVY NANRNYIVLK ILEDYKDHDG DYKDHDIDYK DDDDK |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 4 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 0.8 µm / 最小 デフォーカス(公称値): 0.6 µm Bright-field microscopy / 最大 デフォーカス(公称値): 0.8 µm / 最小 デフォーカス(公称値): 0.6 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 48.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)