+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CRYO-EM STRUCTURE OF SARS-COV-2 SPIKE : H11-B5 nanobody complex | |||||||||
 マップデータ マップデータ | Final sharpened map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 virion component / Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion ... virion component / Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion ... virion component / Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / receptor-mediated endocytosis of virus by host cell / Attachment and Entry / virion component / Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / receptor-mediated endocytosis of virus by host cell / Attachment and Entry /  membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell /  receptor ligand activity / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / receptor ligand activity / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / エンベロープ (ウイルス) / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) / Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) /   Lama glama (ラマ) / Lama glama (ラマ) /   Escherichia virus T4 (T4ファージ) Escherichia virus T4 (T4ファージ) | |||||||||
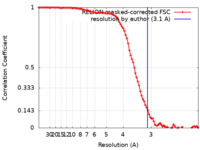
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.1 Å クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Weckener M / Naismith JH | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2022 ジャーナル: Proc Natl Acad Sci U S A / 年: 2022タイトル: Correlation between the binding affinity and the conformational entropy of nanobody SARS-CoV-2 spike protein complexes. 著者: Halina Mikolajek / Miriam Weckener / Z Faidon Brotzakis / Jiandong Huo / Evmorfia V Dalietou / Audrey Le Bas / Pietro Sormanni / Peter J Harrison / Philip N Ward / Steven Truong / Lucile ...著者: Halina Mikolajek / Miriam Weckener / Z Faidon Brotzakis / Jiandong Huo / Evmorfia V Dalietou / Audrey Le Bas / Pietro Sormanni / Peter J Harrison / Philip N Ward / Steven Truong / Lucile Moynie / Daniel K Clare / Maud Dumoux / Joshua Dormon / Chelsea Norman / Naveed Hussain / Vinod Vogirala / Raymond J Owens / Michele Vendruscolo / James H Naismith /   要旨: Camelid single-domain antibodies, also known as nanobodies, can be readily isolated from naïve libraries for specific targets but often bind too weakly to their targets to be immediately useful. ...Camelid single-domain antibodies, also known as nanobodies, can be readily isolated from naïve libraries for specific targets but often bind too weakly to their targets to be immediately useful. Laboratory-based genetic engineering methods to enhance their affinity, termed maturation, can deliver useful reagents for different areas of biology and potentially medicine. Using the receptor binding domain (RBD) of the severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) spike protein and a naïve library, we generated closely related nanobodies with micromolar to nanomolar binding affinities. By analyzing the structure-activity relationship using X-ray crystallography, cryoelectron microscopy, and biophysical methods, we observed that higher conformational entropy losses in the formation of the spike protein-nanobody complex are associated with tighter binding. To investigate this, we generated structural ensembles of the different complexes from electron microscopy maps and correlated the conformational fluctuations with binding affinity. This insight guided the engineering of a nanobody with improved affinity for the spike protein. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14543.map.gz emd_14543.map.gz | 8.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14543-v30.xml emd-14543-v30.xml emd-14543.xml emd-14543.xml | 29.4 KB 29.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14543_fsc.xml emd_14543_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14543.png emd_14543.png | 121.8 KB | ||
| マスクデータ |  emd_14543_msk_1.map emd_14543_msk_1.map | 67 MB |  マスクマップ マスクマップ | |
| その他 |  emd_14543_additional_1.map.gz emd_14543_additional_1.map.gz emd_14543_additional_2.map.gz emd_14543_additional_2.map.gz emd_14543_half_map_1.map.gz emd_14543_half_map_1.map.gz emd_14543_half_map_2.map.gz emd_14543_half_map_2.map.gz | 4.4 MB 52 MB 52.1 MB 52.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14543 http://ftp.pdbj.org/pub/emdb/structures/EMD-14543 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14543 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14543 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7z85MC  7z1aC  7z1bC  7z1cC  7z1dC  7z1eC  7z6vC  7z7xC  7z86C  7z9qC  7z9rC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14543.map.gz / 形式: CCP4 / 大きさ: 67 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14543.map.gz / 形式: CCP4 / 大きさ: 67 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14543_msk_1.map emd_14543_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Sharpened map, output from LocScale.
| ファイル | emd_14543_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map, output from LocScale. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Map from final 3D refinement
| ファイル | emd_14543_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map from final 3D refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map from final 3D refinement
| ファイル | emd_14543_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map from final 3D refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map from final 3D refinement
| ファイル | emd_14543_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map from final 3D refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex between Spike and nanobody H11-B5
| 全体 | 名称: Complex between Spike and nanobody H11-B5 |
|---|---|
| 要素 |
|
-超分子 #1: Complex between Spike and nanobody H11-B5
| 超分子 | 名称: Complex between Spike and nanobody H11-B5 / タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 分子量 | 理論値: 15 KDa |
-超分子 #2: Spike glycoprotein trimer with fibritin tag
| 超分子 | 名称: Spike glycoprotein trimer with fibritin tag / タイプ: complex / キメラ: Yes / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換株: HEK 293T Homo sapiens (ヒト) / 組換株: HEK 293T |
-超分子 #3: Nanobody H11-B5
| 超分子 | 名称: Nanobody H11-B5 / タイプ: complex / キメラ: Yes / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:   Lama glama (ラマ) Lama glama (ラマ) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換株: WK6 Escherichia coli (大腸菌) / 組換株: WK6 |
-分子 #1: Spike glycoprotein,Fibritin
| 分子 | 名称: Spike glycoprotein,Fibritin / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia virus T4 (T4ファージ) Escherichia virus T4 (T4ファージ) |
| 分子量 | 理論値: 139.877906 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDG VYFASTEKSN IIRGWIFGTT LDSKTQSLLI VNNATNVVIK VCEFQFCNDP FLGVYYHKNN KSWMESEFRV Y SSANNCTF ...文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDG VYFASTEKSN IIRGWIFGTT LDSKTQSLLI VNNATNVVIK VCEFQFCNDP FLGVYYHKNN KSWMESEFRV Y SSANNCTF EYVSQPFLMD LEGKQGNFKN LREFVFKNID GYFKIYSKHT PINLVRDLPQ GFSALEPLVD LPIGINITRF QT LLALHRS YLTPGDSSSG WTAGAAAYYV GYLQPRTFLL KYNENGTITD AVDCALDPLS ETKCTLKSFT VEKGIYQTSN FRV QPTESI VRFPNITNLC PFGEVFNATR FASVYAWNRK RISNCVADYS VLYNSASFST FKCYGVSPTK LNDLCFTNVY ADSF VIRGD EVRQIAPGQT GKIADYNYKL PDDFTGCVIA WNSNNLDSKV GGNYNYLYRL FRKSNLKPFE RDISTEIYQA GSTPC NGVE GFNCYFPLQS YGFQPTNGVG YQPYRVVVLS FELLHAPATV CGPKKSTNLV KNKCVNFNFN GLTGTGVLTE SNKKFL PFQ QFGRDIADTT DAVRDPQTLE ILDITPCSFG GVSVITPGTN TSNQVAVLYQ DVNCTEVPVA IHADQLTPTW RVYSTGS NV FQTRAGCLIG AEHVNNSYEC DIPIGAGICA SYQTQTNSPG SASSVASQSI IAYTMSLGAE NSVAYSNNSI AIPTNFTI S VTTEILPVSM TKTSVDCTMY ICGDSTECSN LLLQYGSFCT QLNRALTGIA VEQDKNTQEV FAQVKQIYKT PPIKDFGGF NFSQILPDPS KPSKRSFIED LLFNKVTLAD AGFIKQYGDC LGDIAARDLI CAQKFNGLTV LPPLLTDEMI AQYTSALLAG TITSGWTFG AGAALQIPFA MQMAYRFNGI GVTQNVLYEN QKLIANQFNS AIGKIQDSLS STASALGKLQ DVVNQNAQAL N TLVKQLSS NFGAISSVLN DILSRLDPPE AEVQIDRLIT GRLQSLQTYV TQQLIRAAEI RASANLAATK MSECVLGQSK RV DFCGKGY HLMSFPQSAP HGVVFLHVTY VPAQEKNFTT APAICHDGKA HFPREGVFVS NGTHWFVTQR NFYEPQIITT DNT FVSGNC DVVIGIVNNT VYDPLQPELD SFKEELDKYF KNHTSPDVDL GDISGINASV VNIQKEIDRL NEVAKNLNES LIDL QELGK YEQGSGYIPE APRDGQAYVR KDGEWVLLST FLSLLNDIFE AQKIEWHEKH HHHHH |
-分子 #2: Nanobody H11-B5
| 分子 | 名称: Nanobody H11-B5 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Lama glama (ラマ) Lama glama (ラマ) |
| 分子量 | 理論値: 14.960648 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: QVQLVESGGG LMQAGGSLRL SCAVSGRTFS TAAMGWFRQA PGKEREFVAA IRWSGGSAYY ADSVKGRFTI SRDKAKNTVY LQMNSLKYE DTAVYYCASY QATRSLLSDY ATWPYDYWGQ GTQVTVSSKH HHHHH |
-分子 #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 4 / コピー数: 26 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| グリッド | モデル: Homemade / 材質: COPPER/RHODIUM / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | |||||||||
| 凍結 | 凍結剤: ETHANE / 装置: SPOTITON 詳細: SPT Labtech prototype 300 mesh 1.2/2.0 nanowire grids with a highly reproduceable rectangular bar cross-section were used. The grids were glow-discharged on low for 190 s (Plasma Cleaner PDC- ...詳細: SPT Labtech prototype 300 mesh 1.2/2.0 nanowire grids with a highly reproduceable rectangular bar cross-section were used. The grids were glow-discharged on low for 190 s (Plasma Cleaner PDC-002-CE, Harrick Plasma) to activate the nanowires. Approximately 6 nL of the complex were applied to the grids using a Chameleon EP system (SPT Labtech) at 83% relative humidity and ambient temperature.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 81000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 81000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 詳細 | EPU auto-function coma free correction |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 平均露光時間: 3.0 sec. / 平均電子線量: 45.0 e/Å2 / 詳細: The images were collected as 50 frame movies |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)