+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1391 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structured mRNAs regulate translation initiation by binding to the platform of the ribosome. | |||||||||
 マップデータ マップデータ | Cryo-EM map of the E.coli 70S pre-initiation complex elucidating the entrapment mechanism by the auto-regulatory S15-rpsOmRNA. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mRNA base-pairing translational repressor activity / small ribosomal subunit rRNA binding /  ribosomal small subunit assembly / cytosolic small ribosomal subunit / cytoplasmic translation / ribosomal small subunit assembly / cytosolic small ribosomal subunit / cytoplasmic translation /  rRNA binding / structural constituent of ribosome / rRNA binding / structural constituent of ribosome /  翻訳 (生物学) / 翻訳 (生物学) /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / クライオ電子顕微鏡法 /  ネガティブ染色法 / 解像度: 10.0 Å ネガティブ染色法 / 解像度: 10.0 Å | |||||||||
 データ登録者 データ登録者 | Marzi S | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2007 ジャーナル: Cell / 年: 2007タイトル: Structured mRNAs regulate translation initiation by binding to the platform of the ribosome. 著者: Stefano Marzi / Alexander G Myasnikov / Alexander Serganov / Chantal Ehresmann / Pascale Romby / Marat Yusupov / Bruno P Klaholz /  要旨: Gene expression can be regulated at the level of initiation of protein biosynthesis via structural elements present at the 5' untranslated region of mRNAs. These folded mRNA segments may bind to the ...Gene expression can be regulated at the level of initiation of protein biosynthesis via structural elements present at the 5' untranslated region of mRNAs. These folded mRNA segments may bind to the ribosome, thus blocking translation until the mRNA unfolds. Here, we report a series of cryo-electron microscopy snapshots of ribosomal complexes directly visualizing either the mRNA structure blocked by repressor protein S15 or the unfolded, active mRNA. In the stalled state, the folded mRNA prevents the start codon from reaching the peptidyl-tRNA (P) site inside the ribosome. Upon repressor release, the mRNA unfolds and moves into the mRNA channel allowing translation initiation. A comparative structure and sequence analysis suggests the existence of a universal stand-by site on the ribosome (the 30S platform) dedicated for binding regulatory 5' mRNA elements. Different types of mRNA structures may be accommodated during translation preinitiation and regulate gene expression by transiently stalling the ribosome. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1391.map.gz emd_1391.map.gz | 1.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1391-v30.xml emd-1391-v30.xml emd-1391.xml emd-1391.xml | 11.7 KB 11.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1391.gif 1391.gif | 29 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1391 http://ftp.pdbj.org/pub/emdb/structures/EMD-1391 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1391 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1391 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1391.map.gz / 形式: CCP4 / 大きさ: 6.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1391.map.gz / 形式: CCP4 / 大きさ: 6.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of the E.coli 70S pre-initiation complex elucidating the entrapment mechanism by the auto-regulatory S15-rpsOmRNA. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
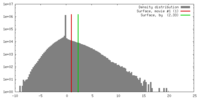
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : 70S pre-initiation complex entrapped by S15-rpsOmRNA
| 全体 | 名称: 70S pre-initiation complex entrapped by S15-rpsOmRNA |
|---|---|
| 要素 |
|
-超分子 #1000: 70S pre-initiation complex entrapped by S15-rpsOmRNA
| 超分子 | 名称: 70S pre-initiation complex entrapped by S15-rpsOmRNA タイプ: sample / ID: 1000 詳細: 70S E. coli ribosome complexed with S15-rpsOmRNA,stalling Translation Initiation at the pre-initiation step. The structure reveals the molecular details of the Entrapment mechanism. 集合状態: monomer / Number unique components: 3 |
|---|---|
| 分子量 | 理論値: 2.5 MDa |
-超分子 #1: 70S
| 超分子 | 名称: 70S / タイプ: complex / ID: 1 / Name.synonym: 70S / 詳細: E. coli 70S ribosome / 組換発現: No / Ribosome-details: ribosome-prokaryote: ALL |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-分子 #1: rpsO messanger RNA
| 分子 | 名称: rpsO messanger RNA / タイプ: rna / ID: 1 / Name.synonym: S15mRNA / 分類: OTHER / Structure: SINGLE STRANDED / Synthetic?: Yes |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: GGGCGAAUUC GAGCUCGGUA CCCAACGUCG CGUAAAUUGU UUAACACUUU GCGUAACGUA CACUGGGAUC GCUGAAUUAG AGAUCGGCGU CCUUUCAUUC UAUAUACUAA GGAGGUUAAA AUGUCUCUAA GUACUGAAGC AACAGCUAAA AUCGUUUCUG AGUUUGGUCG ACCUGCAGGC AUGCA |
-分子 #2: Ribosomal protein S15
| 分子 | 名称: Ribosomal protein S15 / タイプ: protein_or_peptide / ID: 2 / Name.synonym: S15 / 詳細: rpsO gene / 集合状態: monomeric / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) / 組換プラスミド: pET11c Escherichia coli BL21(DE3) (大腸菌) / 組換プラスミド: pET11c |
| 配列 | GO:  翻訳 (生物学) / InterPro: 翻訳 (生物学) / InterPro:  Ribosomal protein S15, bacterial-type Ribosomal protein S15, bacterial-type |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法, ネガティブ染色法,  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 20 mM Tris-HCl pH 7.5, 60 mM KCl, 1mM DTT, 7.5 mM MgCl2 |
| 染色 | タイプ: NEGATIVE / 詳細: no staining, cryo-EM with holey carbon grids |
| グリッド | 詳細: 300 mesh Cu/Rh |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER / 詳細: Vitrification instrument: home-made cryo-plunger / 手法: Blot for 2 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 51484 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 50000 Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Side entry liquid nitrogen-cooled cryo specimen holder 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 温度 | 平均: 77 K |
| アライメント法 | Legacy - 非点収差: lens astigmatism was corrected at 50,000 times magnification |
| 日付 | 2005年1月5日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: PRIMESCAN / デジタル化 - サンプリング間隔: 5 µm / 実像数: 67 / 平均電子線量: 20 e/Å2 / Od range: 1.8 / ビット/ピクセル: 16 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: individual particles |
|---|---|
| 最終 角度割当 | 詳細: beta gamma |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 10.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: IMAGIC-5 / 詳細: exact filtered back-projection / 使用した粒子像数: 31415 |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)