+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: SASBDB / ID: SASDES5 |
|---|---|
 試料 試料 | Neurotrypsin Scavenger Receptor Cysteine-Rich Domain 3 (mmNT-SRCR3) - from SEC-SAXS
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報zymogen activation / exocytosis / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; セリンエンドペプチターゼ / synaptic cleft / Schaffer collateral - CA1 synapse / terminal bouton / peptidase activity / presynapse / cytoplasmic vesicle / axon ...zymogen activation / exocytosis / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; セリンエンドペプチターゼ / synaptic cleft / Schaffer collateral - CA1 synapse / terminal bouton / peptidase activity / presynapse / cytoplasmic vesicle / axon / serine-type endopeptidase activity / dendrite / synapse / glutamatergic synapse / proteolysis / plasma membrane 類似検索 - 分子機能 |
| 生物種 |  |
 引用 引用 |  日付: 2019 Feb 12 日付: 2019 Feb 12タイトル: Structural characterization of the third scavenger receptor cysteine-rich domain of murine Neurotrypsin 著者: Canciani A / Catucci G |
 登録者 登録者 |
|
- 構造の表示
構造の表示
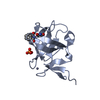
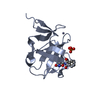
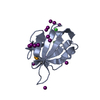
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-モデル
| モデル #2450 | 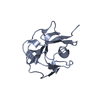 タイプ: atomic コメント: Model generated from PDB file by selecting only chain A カイ2乗値: 1.399  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
|---|
- 試料
試料
 試料 試料 | 名称: Neurotrypsin Scavenger Receptor Cysteine-Rich Domain 3 (mmNT-SRCR3) - from SEC-SAXS 試料濃度: 15 mg/ml |
|---|---|
| バッファ | 名称: 25 mM HEPES/NaOH, 0.1 M NaCl / pH: 8 |
| 要素 #1317 | 名称: mmNT-SRCR3 / タイプ: protein 記述: Mouse Neurotrypsin Scavenger Receptor Cysteine-Rich Domain 3 分子量: 13.165 / 分子数: 1 / 由来: Mus musculus / 参照: UniProt: O08762 配列: GTGEFTSGSG FPIRLVDGEN KKEGRVEVFV NGQWGTICDD GWTDKHAAVI CRQLGYKGPA RARTMAYFGE GKGPIHMDNV KCTGNEKALA DCVKQDIGRH NCRHSEDAGV ICDYLEKKAS S |
-実験情報
| ビーム | 設備名称: ESRF BM29 / 地域: Grenoble / 国: France  / 線源: X-ray synchrotron / 波長: 0.09919 Å / スペクトロメータ・検出器間距離: 2.87 mm / 線源: X-ray synchrotron / 波長: 0.09919 Å / スペクトロメータ・検出器間距離: 2.87 mm | ||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 1M / タイプ: Dectris / Pixsize x: 172 mm | ||||||||||||||||||||||||||||||||||||||||||
| スキャン | 測定日: 2017年5月20日 / 保管温度: 20 °C / セル温度: 20 °C / 照射時間: 1 sec. / フレーム数: 22 / 単位: 1/nm /
| ||||||||||||||||||||||||||||||||||||||||||
| 距離分布関数 P(R) |
| ||||||||||||||||||||||||||||||||||||||||||
| 結果 |
|
 ムービー
ムービー コントローラー
コントローラー


 SASDES5
SASDES5