| 登録情報 | データベース: PDB / ID: 9nit
|
|---|
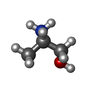
| タイトル | Crystal structure of Vibrio cholerae CqsR bound to L-alaninol |
|---|
 要素 要素 | histidine kinase |
|---|
 キーワード キーワード | SIGNALING PROTEIN / Quorum-sensing receptor / Double Cache domain / Periplasmic ligand-binding receptor / 2-amino alcohol receptor |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
phosphorelay sensor kinase activity / histidine kinase / membrane類似検索 - 分子機能 Methyl-accepting chemotaxis protein-like, first PDC sensor domain / His Kinase A (phospho-acceptor) domain / His Kinase A (phosphoacceptor) domain / Signal transduction histidine kinase, dimerisation/phosphoacceptor domain / Histidine kinase/HSP90-like ATPase / Signal transduction histidine kinase-related protein, C-terminal / Signal transduction histidine kinase, dimerisation/phosphoacceptor domain superfamily / Histidine kinase domain / Histidine kinase domain profile. / Response regulator receiver domain ...Methyl-accepting chemotaxis protein-like, first PDC sensor domain / His Kinase A (phospho-acceptor) domain / His Kinase A (phosphoacceptor) domain / Signal transduction histidine kinase, dimerisation/phosphoacceptor domain / Histidine kinase/HSP90-like ATPase / Signal transduction histidine kinase-related protein, C-terminal / Signal transduction histidine kinase, dimerisation/phosphoacceptor domain superfamily / Histidine kinase domain / Histidine kinase domain profile. / Response regulator receiver domain / cheY-homologous receiver domain / Signal transduction response regulator, receiver domain / Response regulatory domain profile. / CheY-like superfamily / Histidine kinase-, DNA gyrase B-, and HSP90-like ATPase / Histidine kinase-like ATPases / Histidine kinase/HSP90-like ATPase superfamily類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Vibrio cholerae (コレラ菌) Vibrio cholerae (コレラ菌) |
|---|
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 1.72 Å 分子置換 / 解像度: 1.72 Å |
|---|
 データ登録者 データ登録者 | Guarnaccia, A.M. / Neiditch, M.B. |
|---|
| 資金援助 |  米国, 2件 米国, 2件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Institute Of Allergy and Infectious Diseases (NIH/NIAID) | AI121337 |  米国 米国 | | National Institutes of Health/National Center for Advancing Translational Sciences (NIH/NCATS) | TL1TR003019 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Plos Pathog. / 年: 2025 ジャーナル: Plos Pathog. / 年: 2025
タイトル: Structure-function studies of Vibrio cholerae quorum-sensing receptor CqsR signal recognition.
著者: Guarnaccia, A.M. / Steenhaut, A.D. / Olenic, S.D. / Na, J. / Perez, L.J. / Ng, W.L. / Neiditch, M.B. |
|---|
| 履歴 | | 登録 | 2025年2月26日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2025年7月2日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2025年9月17日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year / _citation_author.identifier_ORCID |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  分子置換 / 解像度: 1.72 Å
分子置換 / 解像度: 1.72 Å  データ登録者
データ登録者 米国, 2件
米国, 2件  引用
引用 ジャーナル: Plos Pathog. / 年: 2025
ジャーナル: Plos Pathog. / 年: 2025 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 9nit.cif.gz
9nit.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb9nit.ent.gz
pdb9nit.ent.gz PDB形式
PDB形式 9nit.json.gz
9nit.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ni/9nit
https://data.pdbj.org/pub/pdb/validation_reports/ni/9nit ftp://data.pdbj.org/pub/pdb/validation_reports/ni/9nit
ftp://data.pdbj.org/pub/pdb/validation_reports/ni/9nit


 F&H 検索
F&H 検索 リンク
リンク 集合体
集合体
 要素
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 解析
解析 分子置換 / 解像度: 1.72→19.69 Å / SU ML: 0.18 / 交差検証法: FREE R-VALUE / σ(F): 0.03 / 位相誤差: 21.62 / 立体化学のターゲット値: ML
分子置換 / 解像度: 1.72→19.69 Å / SU ML: 0.18 / 交差検証法: FREE R-VALUE / σ(F): 0.03 / 位相誤差: 21.62 / 立体化学のターゲット値: ML ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj