+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 9c07 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of K2P13.1 (THIK1) S136P in detergent | |||||||||||||||||||||
 要素 要素 | Potassium channel subfamily K member 13 | |||||||||||||||||||||
 キーワード キーワード | TRANSPORT PROTEIN / Potassium channel / K2P channel | |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of excitatory synapse pruning / Tandem pore domain halothane-inhibited K+ channel (THIK) / regulation of NLRP3 inflammasome complex assembly / Phase 4 - resting membrane potential / potassium ion leak channel activity / regulation of resting membrane potential / outward rectifier potassium channel activity / monoatomic ion channel complex / potassium channel activity / potassium ion transmembrane transport ...regulation of excitatory synapse pruning / Tandem pore domain halothane-inhibited K+ channel (THIK) / regulation of NLRP3 inflammasome complex assembly / Phase 4 - resting membrane potential / potassium ion leak channel activity / regulation of resting membrane potential / outward rectifier potassium channel activity / monoatomic ion channel complex / potassium channel activity / potassium ion transmembrane transport / protein heterodimerization activity / metal ion binding / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.73 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Roy-Chowdhury, S. / Adberemane-Ali, F. / Minor, D.L. | |||||||||||||||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2025 ジャーナル: Nat Struct Mol Biol / 年: 2025タイトル: Structure of the human K13.1 channel reveals a hydrophilic pore restriction and lipid cofactor site. 著者: Shatabdi Roy-Chowdhury / Seil Jang / Fayal Abderemane-Ali / Fiona Naughton / Michael Grabe / Daniel L Minor /  要旨: Polyunsaturated fatty acid (PUFA) lipids modulate the neuronal and microglial leak potassium channel K13.1 (THIK1) and other voltage-gated ion channel (VGIC) superfamily members through poorly ...Polyunsaturated fatty acid (PUFA) lipids modulate the neuronal and microglial leak potassium channel K13.1 (THIK1) and other voltage-gated ion channel (VGIC) superfamily members through poorly understood mechanisms. Here we present cryo-electron microscopy structures of human THIK1 and mutants, revealing a unique two-chamber aqueous inner cavity obstructed by a hydrophilic barrier important for gating, the flow restrictor, and a P1-M4 intersubunit interface lipid at a site, the PUFA site, corresponding to the K small-molecule modulator pocket. This overlap, together with functional studies, indicates that PUFA site lipids are THIK1 cofactors. Comparison with a PUFA-responsive VGIC, K7.1, reveals a shared modulatory role for the pore domain intersubunit interface, providing a framework for understanding PUFA action on the VGIC superfamily. Our findings reveal the distinct THIK1 architecture, highlight the importance of the P1-M4 interface for K control by natural and synthetic ligands and should aid in the development of THIK subfamily modulators for neuroinflammation and autism. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  9c07.cif.gz 9c07.cif.gz | 136.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb9c07.ent.gz pdb9c07.ent.gz | 108.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  9c07.json.gz 9c07.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/c0/9c07 https://data.pdbj.org/pub/pdb/validation_reports/c0/9c07 ftp://data.pdbj.org/pub/pdb/validation_reports/c0/9c07 ftp://data.pdbj.org/pub/pdb/validation_reports/c0/9c07 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  45075MC  9bsnC  9bwsC  9byiC  9c09C C: 同じ文献を引用 ( M: このデータのモデリングに利用したマップデータ |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 1種, 2分子 UA
| #1: タンパク質 | 分子量: 39383.613 Da / 分子数: 2 断片: K2P13.1 (THIK1) residues 1 to 350 followed by SNS linker and 3c protease cleavage site 変異: N59Q. N65Q, S136P / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: KCNK13 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: KCNK13 / 発現宿主:  Homo sapiens (ヒト) / 参照: UniProt: Q9HB14 Homo sapiens (ヒト) / 参照: UniProt: Q9HB14 |
|---|
-非ポリマー , 6種, 36分子 
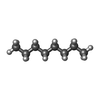
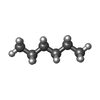








| #2: 化合物 | ChemComp-D12 / #3: 化合物 | ChemComp-OCT / #4: 化合物 | ChemComp-HEX / #5: 化合物 | #6: 化合物 | #7: 化合物 | ChemComp-K / |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|---|
| Has protein modification | N |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: homodimer of K2P13.1 (THIK1) S136P mutant / タイプ: COMPLEX / Entity ID: #1 / 由来: RECOMBINANT |
|---|---|
| 分子量 | 実験値: NO |
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 由来(組換発現) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 緩衝液 | pH: 7.8 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 試料支持 | グリッドの材料: GOLD / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 |
| 急速凍結 | 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277.15 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 800 nm |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 電子線照射量: 46 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| EMソフトウェア |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: NONE | ||||||||||||
| 対称性 | 点対称性: C2 (2回回転対称) | ||||||||||||
| 3次元再構成 | 解像度: 2.73 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 508529 / 対称性のタイプ: POINT |
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj









