+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 8vey | ||||||
|---|---|---|---|---|---|---|---|
| Title | Crystal structure of PRMT5:MEP50 in complex with MTA and TNG908 | ||||||
 Components Components |
| ||||||
 Keywords Keywords | TRANSFERASE/INHIBITOR / MTAP-null / SAM / MTA / inhibitor / MTA-cooperative / TRANSFERASE / TRANSFERASE-INHIBITOR complex | ||||||
| Function / homology |  Function and homology information Function and homology informationpositive regulation of adenylate cyclase-inhibiting dopamine receptor signaling pathway / peptidyl-arginine N-methylation / oocyte axis specification / type II protein arginine methyltransferase / protein-arginine omega-N symmetric methyltransferase activity / peptidyl-arginine methylation / Golgi ribbon formation / negative regulation of epithelial cell proliferation involved in prostate gland development / secretory columnal luminar epithelial cell differentiation involved in prostate glandular acinus development / histone H4R3 methyltransferase activity ...positive regulation of adenylate cyclase-inhibiting dopamine receptor signaling pathway / peptidyl-arginine N-methylation / oocyte axis specification / type II protein arginine methyltransferase / protein-arginine omega-N symmetric methyltransferase activity / peptidyl-arginine methylation / Golgi ribbon formation / negative regulation of epithelial cell proliferation involved in prostate gland development / secretory columnal luminar epithelial cell differentiation involved in prostate glandular acinus development / histone H4R3 methyltransferase activity / : / epithelial cell proliferation involved in prostate gland development / protein-arginine N-methyltransferase activity / methylosome / positive regulation of mRNA splicing, via spliceosome / methyl-CpG binding / endothelial cell activation / histone H3 methyltransferase activity / histone methyltransferase activity / regulation of mitotic nuclear division / negative regulation of gene expression via chromosomal CpG island methylation / Cul4B-RING E3 ubiquitin ligase complex / E-box binding / histone methyltransferase complex / positive regulation of oligodendrocyte differentiation / negative regulation of cell differentiation / spliceosomal snRNP assembly / ribonucleoprotein complex binding / regulation of ERK1 and ERK2 cascade / ubiquitin-like ligase-substrate adaptor activity / liver regeneration / regulation of signal transduction by p53 class mediator / methyltransferase activity / circadian regulation of gene expression / DNA-templated transcription termination / Regulation of TP53 Activity through Methylation / RMTs methylate histone arginines / protein polyubiquitination / p53 binding / transcription corepressor activity / snRNP Assembly / ubiquitin-dependent protein catabolic process / transcription coactivator activity / chromatin remodeling / protein heterodimerization activity / positive regulation of cell population proliferation / regulation of DNA-templated transcription / regulation of transcription by RNA polymerase II / chromatin / Golgi apparatus / nucleoplasm / identical protein binding / nucleus / cytosol / cytoplasm Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  FOURIER SYNTHESIS / Resolution: 2.44 Å FOURIER SYNTHESIS / Resolution: 2.44 Å | ||||||
 Authors Authors | Whittington, D.A. | ||||||
| Funding support |  United States, 1items United States, 1items
| ||||||
 Citation Citation |  Journal: J.Med.Chem. / Year: 2024 Journal: J.Med.Chem. / Year: 2024Title: Discovery of TNG908: A Selective, Brain Penetrant, MTA-Cooperative PRMT5 Inhibitor That Is Synthetically Lethal with MTAP -Deleted Cancers. Authors: Cottrell, K.M. / Briggs, K.J. / Whittington, D.A. / Jahic, H. / Ali, J.A. / Davis, C.B. / Gong, S. / Gotur, D. / Gu, L. / McCarren, P. / Tonini, M.R. / Tsai, A. / Wilker, E.W. / Yuan, H. / ...Authors: Cottrell, K.M. / Briggs, K.J. / Whittington, D.A. / Jahic, H. / Ali, J.A. / Davis, C.B. / Gong, S. / Gotur, D. / Gu, L. / McCarren, P. / Tonini, M.R. / Tsai, A. / Wilker, E.W. / Yuan, H. / Zhang, M. / Zhang, W. / Huang, A. / Maxwell, J.P. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  8vey.cif.gz 8vey.cif.gz | 394.7 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb8vey.ent.gz pdb8vey.ent.gz | Display |  PDB format PDB format | |
| PDBx/mmJSON format |  8vey.json.gz 8vey.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Summary document |  8vey_validation.pdf.gz 8vey_validation.pdf.gz | 1.1 MB | Display |  wwPDB validaton report wwPDB validaton report |
|---|---|---|---|---|
| Full document |  8vey_full_validation.pdf.gz 8vey_full_validation.pdf.gz | 1.1 MB | Display | |
| Data in XML |  8vey_validation.xml.gz 8vey_validation.xml.gz | 35.1 KB | Display | |
| Data in CIF |  8vey_validation.cif.gz 8vey_validation.cif.gz | 48.6 KB | Display | |
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/ve/8vey https://data.pdbj.org/pub/pdb/validation_reports/ve/8vey ftp://data.pdbj.org/pub/pdb/validation_reports/ve/8vey ftp://data.pdbj.org/pub/pdb/validation_reports/ve/8vey | HTTPS FTP |
-Related structure data
| Related structure data |  8veoC  8vetC  8veuC  8vewC  8vexC C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| Unit cell |
| |||||||||
| Components on special symmetry positions |
|
- Components
Components
-Protein , 2 types, 2 molecules AB
| #1: Protein | Mass: 73763.625 Da / Num. of mol.: 1 / Fragment: FULL LENGTH Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: PRMT5, HRMT1L5, IBP72, JBP1, SKB1 / Production host: Homo sapiens (human) / Gene: PRMT5, HRMT1L5, IBP72, JBP1, SKB1 / Production host:  Trichoplusia ni (cabbage looper) Trichoplusia ni (cabbage looper)References: UniProt: O14744, type II protein arginine methyltransferase |
|---|---|
| #2: Protein | Mass: 37862.406 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: WDR77, MEP50, WD45, HKMT1069, Nbla10071 / Production host: Homo sapiens (human) / Gene: WDR77, MEP50, WD45, HKMT1069, Nbla10071 / Production host:  Trichoplusia ni (cabbage looper) / References: UniProt: Q9BQA1 Trichoplusia ni (cabbage looper) / References: UniProt: Q9BQA1 |
-Non-polymers , 6 types, 195 molecules 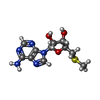








| #3: Chemical | ChemComp-MTA / | ||||||
|---|---|---|---|---|---|---|---|
| #4: Chemical | ChemComp-A1AAV / Mass: 409.505 Da / Num. of mol.: 1 / Source method: obtained synthetically / Formula: C21H23N5O2S / Feature type: SUBJECT OF INVESTIGATION | ||||||
| #5: Chemical | ChemComp-PEG / #6: Chemical | #7: Chemical | ChemComp-CL / #8: Water | ChemComp-HOH / | |
-Details
| Has ligand of interest | Y |
|---|---|
| Has protein modification | Y |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.78 Å3/Da / Density % sol: 55.83 % |
|---|---|
| Crystal grow | Temperature: 291 K / Method: vapor diffusion / pH: 6 Details: 10% PEG 4000, 0.2M magnesium chloride, 0.1M sodium citrate (pH 6.0) |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  Diamond Diamond  / Beamline: I03 / Wavelength: 0.97628 Å / Beamline: I03 / Wavelength: 0.97628 Å |
| Detector | Type: DECTRIS EIGER2 XE 16M / Detector: PIXEL / Date: Sep 18, 2020 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.97628 Å / Relative weight: 1 |
| Reflection | Resolution: 2.44→60.23 Å / Num. obs: 46656 / % possible obs: 99.9 % / Redundancy: 13.9 % / CC1/2: 0.999 / Rmerge(I) obs: 0.085 / Rpim(I) all: 0.024 / Rrim(I) all: 0.089 / Χ2: 1.01 / Net I/σ(I): 18.3 / Num. measured all: 647533 |
| Reflection shell | Resolution: 2.44→2.53 Å / % possible obs: 99.9 % / Redundancy: 13.9 % / Rmerge(I) obs: 1.134 / Num. measured all: 62519 / Num. unique obs: 4504 / CC1/2: 0.932 / Rpim(I) all: 0.315 / Rrim(I) all: 1.178 / Χ2: 0.97 / Net I/σ(I) obs: 2.3 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  FOURIER SYNTHESIS / Resolution: 2.44→51.33 Å / Cor.coef. Fo:Fc: 0.948 / Cor.coef. Fo:Fc free: 0.936 / SU B: 27.46 / SU ML: 0.29 / Cross valid method: THROUGHOUT / ESU R: 0.41 / ESU R Free: 0.269 / Stereochemistry target values: MAXIMUM LIKELIHOOD / Details: HYDROGENS ADDED IN THE RIDING POSITIONS FOURIER SYNTHESIS / Resolution: 2.44→51.33 Å / Cor.coef. Fo:Fc: 0.948 / Cor.coef. Fo:Fc free: 0.936 / SU B: 27.46 / SU ML: 0.29 / Cross valid method: THROUGHOUT / ESU R: 0.41 / ESU R Free: 0.269 / Stereochemistry target values: MAXIMUM LIKELIHOOD / Details: HYDROGENS ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Ion probe radii: 0.8 Å / Shrinkage radii: 0.8 Å / VDW probe radii: 1.2 Å / Solvent model: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 71.778 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: 1 / Resolution: 2.44→51.33 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
|
 Movie
Movie Controller
Controller



 PDBj
PDBj



